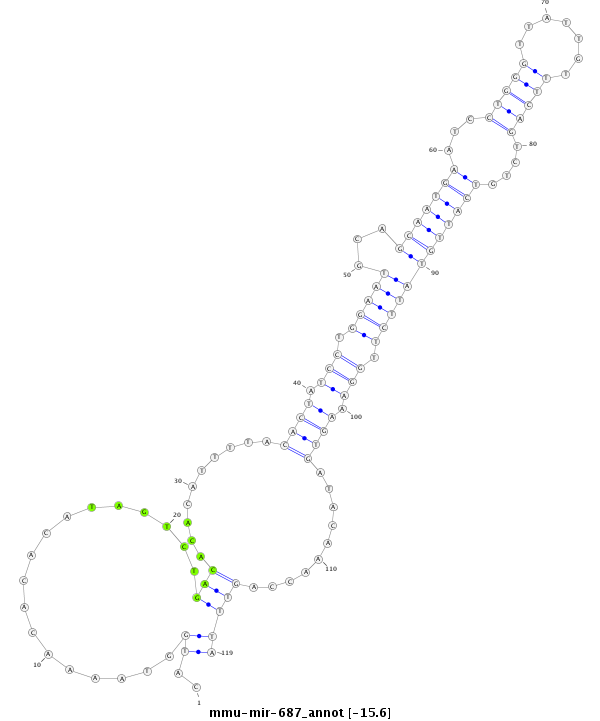


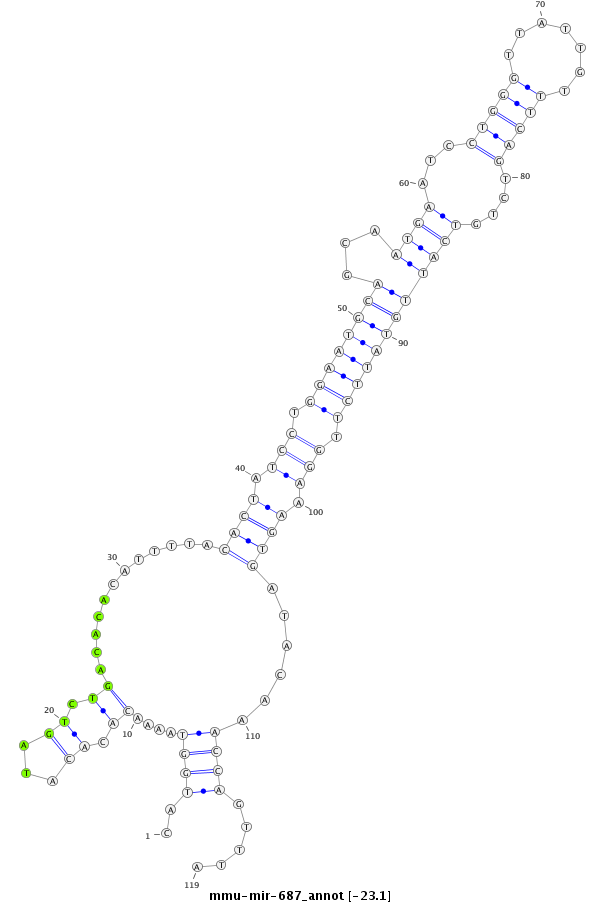
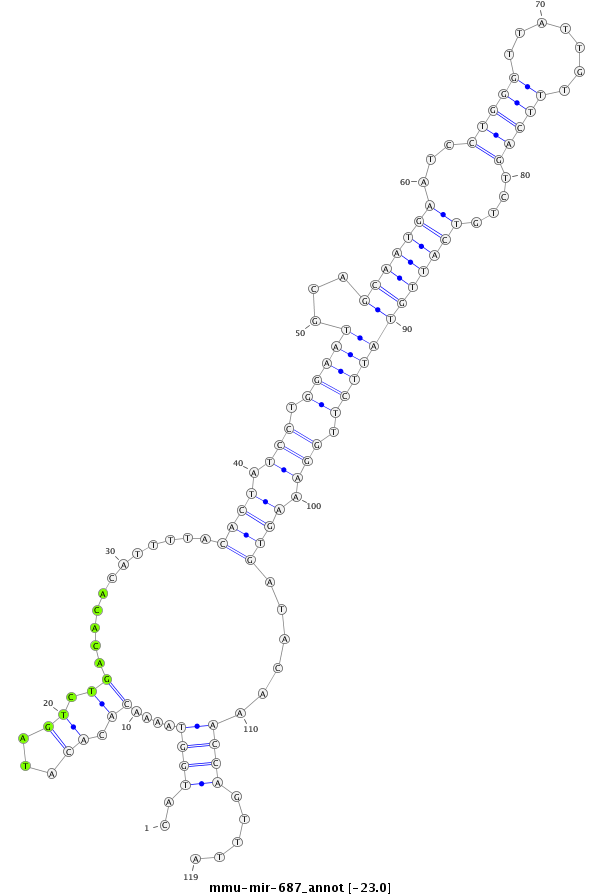
ID:mmu-mir-687 |
Coordinate:chr14:73206763-73206851 - |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -23.1 | -23.0 | -22.8 | -22.8 |
 |
 |
 |
 |
|
TTTATACCTCTAAATTCTTCTCCCACAGATGCTATCATGGTAAAACACACATAGTCTGACACACATTTTACACTATCCTGGAATGCAGCAATGAATCCTGGGTTATTGTTTCAGTCTGTCATTGTATTCTTGGAAAGTGATACAAACCAGTTTAGTTTGCATTATATATTTGGAGTTATTTTCTAAATT
***********************************..((..................(((..........((((.(((.(((((...(((((((...(((((.......)))))....)))))))))))).))).))))..........)))))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345197 brain |
SRR345200 brain |
SRR037901 testes |
|---|---|---|---|---|---|---|---|---|
| ....................TCCCGGAGACGCTATCATGGTA................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| ...............................................................................................TCCTGGGGTAGTGTTTCAGTC......................................................................... | 21 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 |
| ..............................................................................TGGAATGCGGTAATGAATCG........................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 |
| ..................................................................................................TGGGTTGTTGTGTCCGTCTGTC..................................................................... | 22 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 |
| ...................................................TAGTCTGACACA.............................................................................................................................. | 12 | 0 | 20 | 0.05 | 1 | 0 | 1 | 0 |
|
AAATATGGAGATTTAAGAAGAGGGTGTCTACGATAGTACCATTTTGTGTGTATCAGACTGTGTGTAAAATGTGATAGGACCTTACGTCGTTACTTAGGACCCAATAACAAAGTCAGACAGTAACATAAGAACCTTTCACTATGTTTGGTCAAATCAAACGTAATATATAAACCTCAATAAAAGATTTAA
***********************************..((..................(((..........((((.(((.(((((...(((((((...(((((.......)))))....)))))))))))).))).))))..........)))))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR391847 embryo |
SRR391852 embryo |
SRR345198 brain |
SRR095855BC7 heart |
SRR345200 brain |
SRR391845 embryo |
SRR345203 brain |
SRR037902 testes |
SRR039153 muscle |
SRR059767 thymus |
SRR345199 brain |
SRR345201 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....ATGGCTATTTAAGAATAGGGTG................................................................................................................................................................... | 22 | 3 | 4 | 1.00 | 4 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................GTCGTTACTTAGGA.......................................................................................... | 14 | 0 | 20 | 0.25 | 5 | 0 | 0 | 3 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................AGGGTGTCTAC.............................................................................................................................................................. | 11 | 0 | 20 | 0.10 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....TGGCTATTTAAGAATAGGGTG................................................................................................................................................................... | 21 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................TATGTGCGGTCAAATCTAACGT............................ | 22 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................AAGTCGTTACATAGGACCCA...................................................................................... | 20 | 2 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................AAAGTCAGACGGTAACATAG............................................................. | 20 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................CTATGTTTGGTCA...................................... | 13 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................GATGTATGTTCAAATCAAACGT............................ | 22 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................CCGTCGTTACTTAGGA.......................................................................................... | 16 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ............................................................................................CTGTGGACGCAATAACAAAGT............................................................................ | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr14:73206713-73206901 - | mmu-mir-687 | TTTATACCTCTAAATTCTTCTCCC-ACAG--A-TGCTATCATG-GTAAAACACACATAGTCTGACACACATTTTACACTATCCTGGAATGCAGCAA-----TGAATCCTGGGTTATTGTTTCAGTCTGTCATTGTATTCTTGGAAAGTGATACAAACCAGTTTAGTTTGCATTATATATTTGGAGTTATTTTCTAAATT |
| cavPor3 | scaffold_6:58424268-58424452 + | TCTG--TTTCCAGTTTACCTTCCTGGTAGAGA-AGCATTAGTA-GTAAAATACACTTGGTATGAAACACATTTTATACTCTTCCGTGACAG----GT-----GAGTTGTGGATTAT-TTTCCAGTTTACCTTTGGCGTTTTCAAAAGTAATATAAAATAGATCATTTCAGTTTATATATTTGAAATTATTTTCTAAACC | |
| dipOrd1 | Unknown | TTTGTATTTTCAGTTTCCCTTCCC-AGAGAGA-AGCAGTAATGGGTAAACAGCATTTATTATGGCATACATGTTATATGCTTTT---------------------TTCTAGGTTATTTTTCCAATTTATTATTGGTTTTTT---------------------------------------CNNAGTTATTTTCTAAATC | |
| hetGal2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| oryCun2 | Unknown | TGT--GTTTCTAGTTTACCTTTCTTACAGAATCAACAACAGTG-ATAAAATGCACTTGGTGTGGCACACAGTTTATACC---CTGTGACAG----ATGGGTTGAAAACTGAGC-A--TTTCTAGTTTATCAGTGGAGTTTTCAAAAGTAATACACGTGAGGTTATTTTTGTTTATATTTTTGAAGTTATTTTCTGAATC | |
| rn5 | chr15:58816652-58816840 - | TTTGTGTCTCTAGTTTTTTCTCCC-ACAGAGA-TGCTATCATG-GTA-ATCACACTTAGTCTGACACACATTTTACACTGTCCTGGAACGCA-CAG-----TGAATCCTGGGTTATTGTTTCAGTCTGTCATTGTATTTTTGGAAAGTGATACAAACCAGTTTGGTTTTACTCATATATTGGGAGTTATTTTCTAAATT | |
| speTri2 | Unknown | TTTGTGCTTCTAGTTTACCTCCCCTGTAGGCA-AGTAGTATTA-ATAAAATACACTTGATGTGACATACATTTCATACCCTCCTATGATAA----GT------------GAGTAATTTTTCCAGCTTAATATTGGAGTTTTCAGAAGTAATACAGACCAGGTTATTTT----TATGTATTTGCAATTATTTTTTAAATC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 04/30/2015 at 08:12 PM