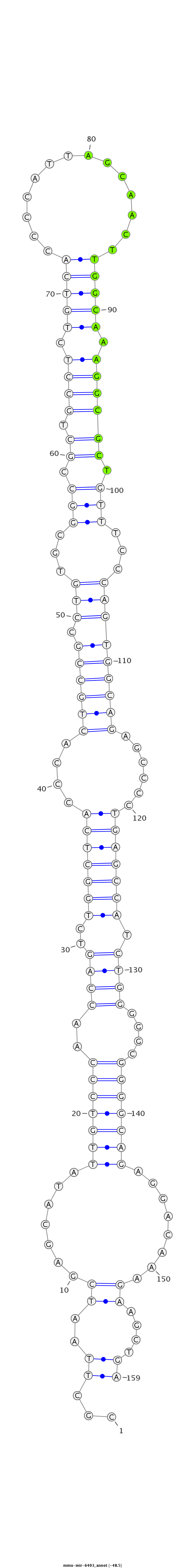
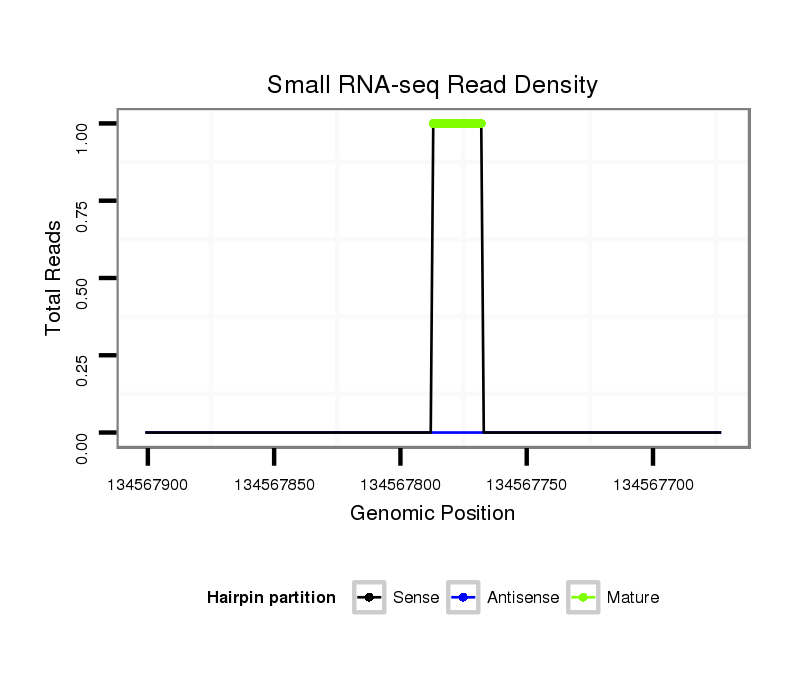
ID:mmu-mir-6403 |
Coordinate:chr4:134567723-134567851 - |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -56.5 | -56.0 | -55.9 | -55.9 |
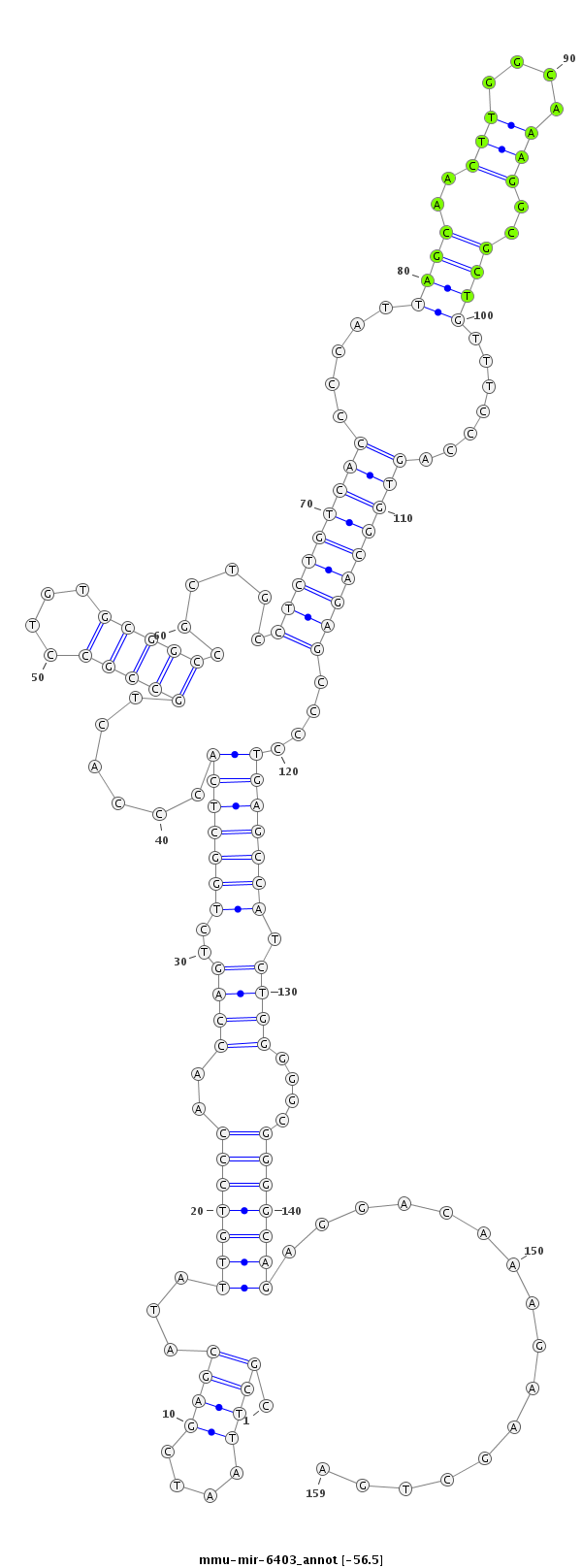 |
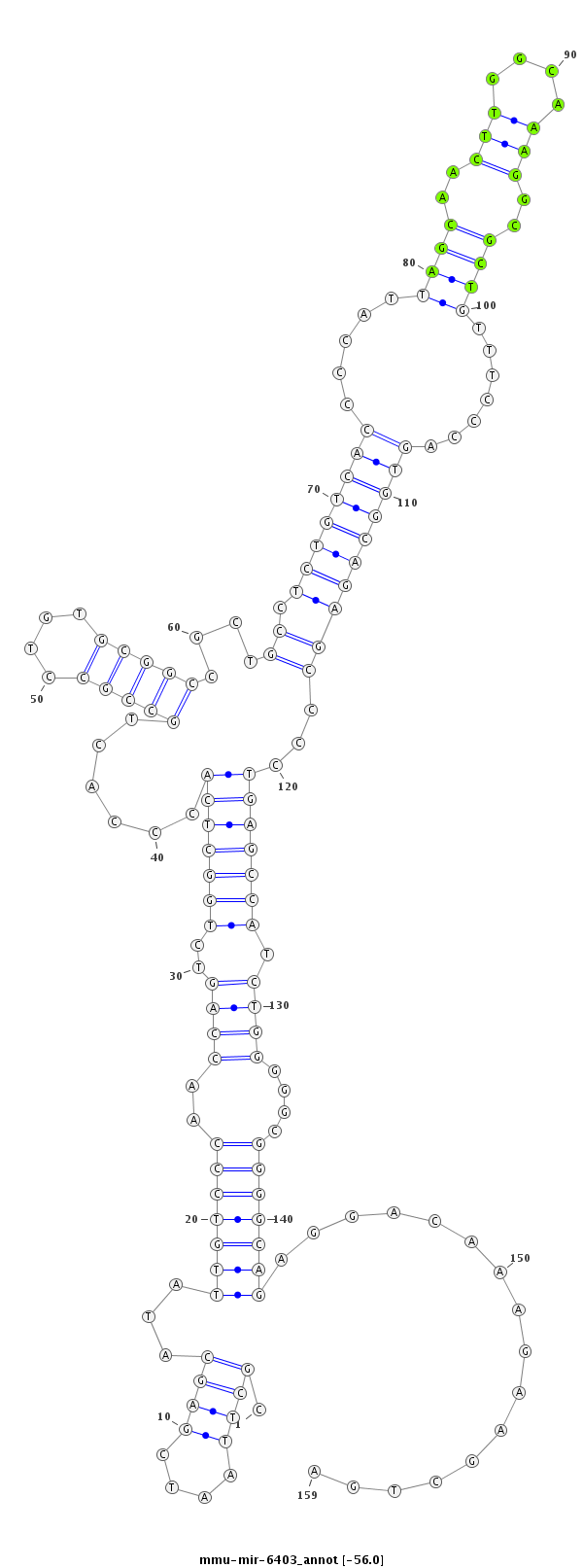 |
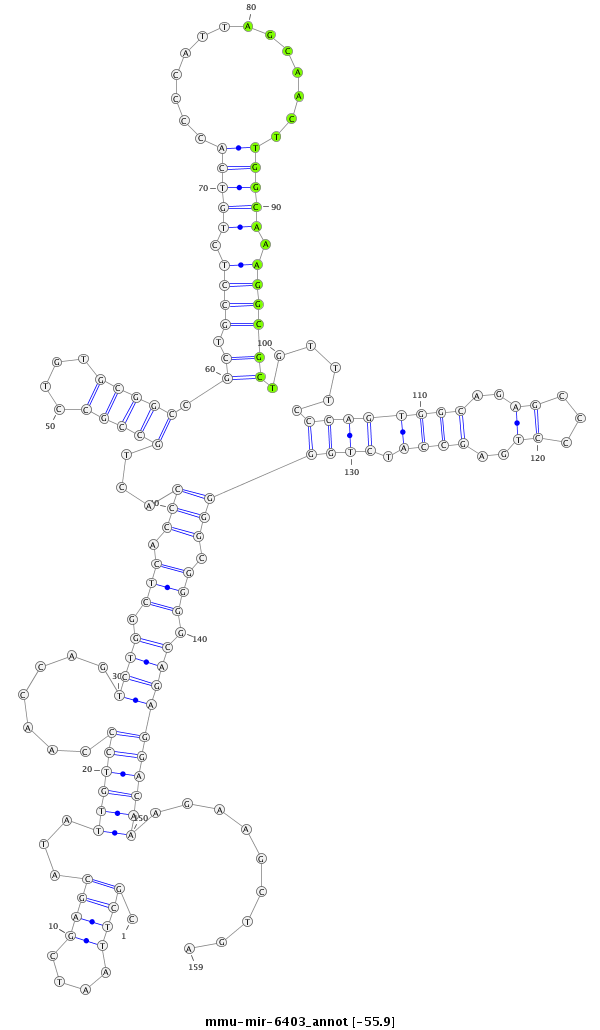 |
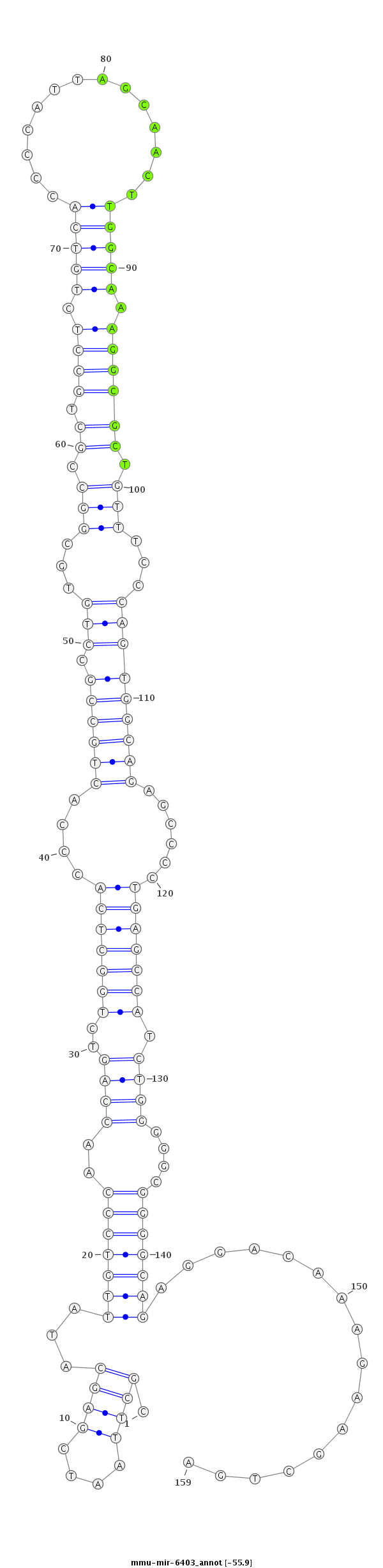 |
|
GTCTGGGACAATCATCCAGGGCTGTAAAAGACAGCCGCTTAATCGAGCATATTGTCCCAACCAGTCTGGCTCACCCACTGCCGCCTGTGCGGCCGCTGCCTCTGTCACCCCATTAGCAACTTGGCAAAGGCGCTGTTTCCCAGTGGCAGAGCCCCTGAGCCATCTGGGGGCGGGGCAGAGGACAAAGAAGCTGAGCCCAGTGACTCCTAAGGAGATACACCTCCAGACT
***********************************...((..((.......(((((((..((((..(((((((....((((((.(((...(((.((.((((.(((((..............))))).)))))).)))...)))))))))......))))))).))))....)))))))........))....))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR073955 blood |
SRR391849 embryo |
SRR037929 embryo |
SRR248524 testes |
SRR345196 brain |
SRR039152 muscle |
SRR345199 brain |
SRR345203 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................GCAACTTGGCAATGGCGCGG.............................................................................................. | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AGCAACTTGGCAAAGGCGCT............................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................GTCCGGCCGCTGCCTCTGTCC.......................................................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................TGCGGCCGCGGCCTCTG............................................................................................................................. | 17 | 1 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................GAGGACAAAGAAGCTG.................................... | 16 | 0 | 20 | 0.10 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................CTGGGGGCGGGCCAGACGA............................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................................................................................TGCGGCCGCACCCTCTATCA.......................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................TGCGGCCGCTGTCTCGGTCG.......................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CAGACCCTGTTAGTAGGTCCCGACATTTTCTGTCGGCGAATTAGCTCGTATAACAGGGTTGGTCAGACCGAGTGGGTGACGGCGGACACGCCGGCGACGGAGACAGTGGGGTAATCGTTGAACCGTTTCCGCGACAAAGGGTCACCGTCTCGGGGACTCGGTAGACCCCCGCCCCGTCTCCTGTTTCTTCGACTCGGGTCACTGAGGATTCCTCTATGTGGAGGTCTGA
***********************************...((..((.......(((((((..((((..(((((((....((((((.(((...(((.((.((((.(((((..............))))).)))))).)))...)))))))))......))))))).))))....)))))))........))....))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345201 brain |
SRR345198 brain |
SRR345200 brain |
SRR345199 brain |
SRR391846 embryo |
SRR345197 brain |
SRR345202 brain |
SRR039153 muscle |
SRR116846 epididymis |
SRR042464 thymus |
SRR095855BC7 heart |
SRR345203 brain |
SRR345204 brain |
SRR345207 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................AGTGGCCGACGGCGGCCACGCC......................................................................................................................................... | 22 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................TCTGTCGGCCAATTGGCTCAT.................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................TCGCGGACTCGGTAGGCCC............................................................. | 19 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................TCGGCGCATTAGCT....................................................................................................................................................................................... | 14 | 1 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................AGACAGTGGGGT..................................................................................................................... | 12 | 0 | 20 | 0.15 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CCGACCTGTTCTGTCGGCG............................................................................................................................................................................................... | 19 | 2 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................CATTGGCGTAATCGTGGAACC......................................................................................................... | 21 | 3 | 14 | 0.14 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................TTTCCGCGGCCAAGGGTCACT................................................................................... | 21 | 3 | 15 | 0.13 | 2 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................ATTAGCTCGTATAA................................................................................................................................................................................ | 14 | 0 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................GGGTGACGGCGG................................................................................................................................................ | 12 | 0 | 20 | 0.10 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................................................................................ACGCCGGCGCCGGATAGAGT.......................................................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................TCTCGGGGACTCGATAGGCCCG............................................................ | 22 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................GGCGACGGAGACAGCAGGGCA.................................................................................................................... | 21 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................TTCCGCGACAAAGGCGCTCC................................................................................... | 20 | 3 | 15 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................AGGCCGGCGACGGAGGCAG........................................................................................................................... | 19 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................TCTGTCGGCG............................................................................................................................................................................................... | 10 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................ATAACAGGGTAGGGCAGA.................................................................................................................................................................. | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................ACGGCGGACACGCCGGGA..................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................CGTTTTCTGTCGGCG............................................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................................ATTGGGTGACGGCGG................................................................................................................................................ | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................CCCGGCTTGTTCTGTCGGCG............................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................GACAAAGGGTCACGGTCGC.............................................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................CATTTTCAGTCGGCG............................................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................CTCTATGTGGAGG..... | 13 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr4:134567673-134567901 - | mmu-mir-6403 | GTCTGGGACAATCATCCAGGGCTGTAAAAGACAGCCGCTTAATCGAGCATATTGTCCCAACCAGTCTGGCTC-------ACCCACTGCCGCCTGTGCG-------GCCGC-----------TGCCTCTGTCACCCCATTAGCAACTTGGCAAAGGCGCTGTTT---CCCAGTGG--CAGAGCCCCTGAGCCATCTGGGG---------GCGGG--GCAGAGGACA-AAGAAGCTGAGCCCAG-TGACTCCTAAGGAGATACACCTCCAGACT |
| cavPor3 | scaffold_25:10279236-10279457 - | GTCTGGGACAATCAGCACAGACCATAAAAGACAGCCACTTAATAGAGCGTATTGTTCCTGGTGCCCT----------------GCTGGCGGCTGCCACAGTGCCGGCTGCCCTCACCCCAGTGCTGTCCTCACCCCATTAGCAACTTGGCAAAACAACTGTTG---CCTGATGGCCTGGAGTGGCTTCGACATTTGAAA-AG----------ATGGGAGGGA--CCACAG-------TTTAGCTGACCCCT-----------CCTGCAGGAA | |
| dipOrd1 | scaffold_29884:14279-14490 - | GTCTGGGACAATCAGGCCAGGCCATAAAAGACAGCCGCTTAATCGAGCATATTGTCCCAGCCAGCCTGATTT-------GCCCACTGCCGGCTGTGTG-------GCTGC-----------TCCTGCCCTCACCCCATTAGCAACTTGGCAAAACCACTGTTT---CCTGATGGGCCAGAGCTGCCACACCATTTGAAGAAG----------ATTAGAGGGG--CCAGA--------GTT-GCTGACTCCT-----------CTTCCAGGAA | |
| hetGal2 | Unknown | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | scaffold_2142:78805-78909 + | GTTTAGGTGGATCAGCACAGGCCTTAAAAGACAGAGGGTTAATAGAGCATCCTGTTCTGGCTGCCACAGCTGCTCCGTGGCCCACAGACAGCTGTCAC------GGCTGC-----------T------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| oryCun2 | chr13:127951503-127951669 - | GTCTGGGACGATCGGCCCGGGCCGTAAAAGACGGCCGCTTAATGGGGCGCCTTGTGCTGGCCAGCCGGGCTC------AGCCCACGGACGCCTGCCAC-------GCTGC-----------TGCCACCTTGGTCCCATTAGCAACTTGGCAGAGCAGCTCTGC---CAT-------------------------------------------G--GGAGAGGACC-AAGGCGCTGGACCC-------------------------------- | |
| rn5 | chr5:156566416-156566651 - | GTCTGGGACAATCACCCAGGGCTGGAAAAGACAGCCGCTTAATCGAGCATATTGTCCCAACCAGCCTGGCTT-------GCCTGCTGCTGGCTTTGCC-------GCCGG-----------TGCCTCTGTCACCCCATTAGCAGCTTGGCACAAGCGCTGTCT---CCCAGTGG--CAGAGCCT-GGAGCCATCTGGGG--GGGGAGGGCGGG--GGGGGGGGCA-AAGAAGCCGAGCCCAGTTGACTCCTAAGAAGATACATCTCCAGAAT | |
| speTri2 | JH393284:10362385-10362573 + | GTCTGGGACAGTCAGCCCC-ATCATGGAGGACGGTCCCTCGGCAGGGCCCAGTGCTCCCGCCAGCCTGGCT--------GCCCGCT---------------------------------------GCCCTCACCCTGTTAGCAACGTGGCACAACCACCGTGTTGTCCTGGGG--CTCCAGCCGCGTCACCATCTGAC--AG----------GTGGGAGGGG--C-GGGG-------CCTAGATCCCTGCT-----------CTTCCAGGAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 04/30/2015 at 11:50 PM