
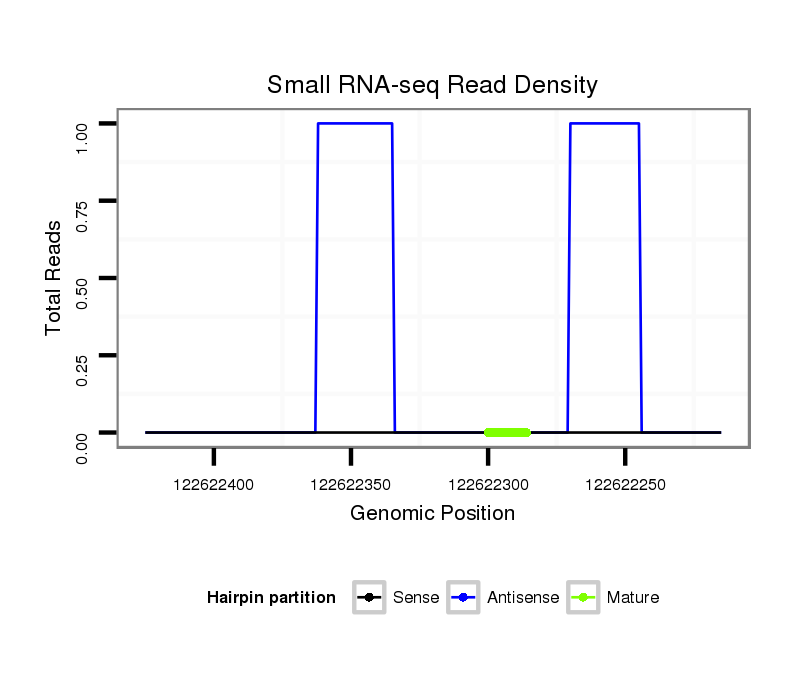
ID:mmu-mir-6396 |
Coordinate:chr8:122622265-122622375 - |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -56.2 | -55.9 | -55.9 |
 |
 |
 |
|
CCGTCACAAACCGTGCAGGAAGGTGTGGACCTGGGCATGGAGGGAGGAGCACAGGTGACTAGGGGCGAAGGAGCCAGTGCTTCAGACCTCAGAGGCCTCTCTGACTAGGGGGAGGTATCCGGGCAGGAGAGGTACCAGGGGCCTTAGCTCCTAGTCCTGGACTATGGCTACAGGAACAACAGGGTGATCTTGGTTATCCCCTAGTAGCTAT
***********************************..((.((.....((..((((..(((((((((.((((..((.(((((((....(((.((.((((((((......)))))))).)).))).....)))))))..))..)))).)))))))))))))..))....)).))....*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345198 brain |
SRR039154 muscle |
SRR391845 embryo |
SRR345205 brain |
SRR039153 muscle |
SRR345197 brain |
SRR039152 muscle |
SRR345203 brain |
SRR345204 brain |
SRR345199 brain |
SRR037925 embryo |
SRR345196 brain |
SRR553582 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................TACCCGGGGCCTTAGCTCGT........................................................... | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................AACAACAGGGTGATC...................... | 15 | 0 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...TCACAAACCGGACCGGAAGGTG.......................................................................................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTACTAGGGGGCTTAGTT.............................................................. | 22 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTACGAGGGTGCTTAGCT.............................................................. | 22 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................GGAGAGGTACCAGGG....................................................................... | 15 | 0 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTACCAGGTTGCTTAGCT.............................................................. | 22 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTACTAGGGTGCTTAGCT.............................................................. | 22 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTAGTAGGGGTCTTAGCT.............................................................. | 22 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................GAGGGAGGAGCATAGGTG.......................................................................................................................................................... | 18 | 1 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................CGGCAGGAGAGGTACC........................................................................... | 16 | 1 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTAGCAGGGTCCATAGCT.............................................................. | 22 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AGAGGTACCAGGTTCCTTAGTT.............................................................. | 22 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................GACCTCAGAGGGCTCTATG............................................................................................................ | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ................................................................................................................................................................................................GTTATCCCCTCGTAGATAC | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................................................................CGGAACGACAGGGTGAT....................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
GGCAGTGTTTGGCACGTCCTTCCACACCTGGACCCGTACCTCCCTCCTCGTGTCCACTGATCCCCGCTTCCTCGGTCACGAAGTCTGGAGTCTCCGGAGAGACTGATCCCCCTCCATAGGCCCGTCCTCTCCATGGTCCCCGGAATCGAGGATCAGGACCTGATACCGATGTCCTTGTTGTCCCACTAGAACCAATAGGGGATCATCGATA
***********************************..((.((.....((..((((..(((((((((.((((..((.(((((((....(((.((.((((((((......)))))))).)).))).....)))))))..))..)))).)))))))))))))..))....)).))....*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR248524 testes |
SRR345196 brain |
SRR073954 blood |
SRR345200 brain |
SRR345198 brain |
SRR391847 embryo |
SRR345207 brain |
SRR116846 epididymis |
SRR042464 thymus |
SRR042465 spleen |
SRR042466 spleen |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................CCGCTTCCTCGGTCACGAAGTCTGGAGT........................................................................................................................ | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................GGACCTGATACCGATGTCCTTGTTGT.............................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................TGATCCAGCTGCATAGGCCC........................................................................................ | 20 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................GCCCCCTCGATAGGCCCG....................................................................................... | 18 | 2 | 8 | 0.25 | 2 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................CCACCTCGATAGGCCCG....................................................................................... | 17 | 2 | 20 | 0.15 | 3 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................CGTCTTCTCGATGGTCCCTGGA................................................................... | 22 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 |
| ............................................................................................................................TCCTCTCGATGGTCCCCGGC................................................................... | 20 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................CTCCATAGGCGCGTC..................................................................................... | 15 | 1 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................CTCCATAGGCCCG....................................................................................... | 13 | 0 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................CTCCATGGTCCCCGGGA.................................................................. | 17 | 1 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................CGTCACGAAATCTGGAGTCCCC.................................................................................................................... | 22 | 3 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................TCCCCGCTTCCTC.......................................................................................................................................... | 13 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................GGTCACGAAATCTGAAGTCCCC.................................................................................................................... | 22 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................CCCCCCTCGATAGGCCCG....................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................CCTCCATAGGCC......................................................................................... | 12 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................CTCCATGGTCCCCGGG................................................................... | 16 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................CTGCCTCCATAGGCCCG....................................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr8:122622215-122622425 - | mmu-mir-6396 | CCGTCACAAACCGTGCAGGAAG---GTGTGGACCTGGGCATGGAGGGAGGAGCACAGGTGACTAGG-GGCGAAGGAGCCAGTGCTT---CAGACCTCAGAGGCCT----------------------------------------------------CTCTGACTAGGGGGAGGTATCC-GGGC--AGGAGAGGTACCAGG----------GGC------------------------------CTTAGCTCCTAGTCCTGGACTATGGCTACAGGAACAACAGG----GTGATCTTG-----------------GTTATCCCCTAGTAGCTAT |
| cavPor3 | scaffold_346:53006-53154 + | CAGGAGG---------TGGCAG---GCAGGGACCTAGGCACAGGC-----------------TGGG-GACAAAGGGACAAGGAACTGACCGGAAGGCAGA-------------------------------------------------------------------GGGGTGCCTGCCTGGGGATGGTGGAGATCACAGGGCTTCGCTGAGGC------------------------------CTGAG--------------CACTGGC-----A---------GAGGGTCCT------------------------------CTGAGTCTGC | |
| dipOrd1 | scaffold_183389:266-458 - | GGGGTGTGG----------------------ACGTGGGGTGTGGGTGTGGAg----------GgGGGggGGGGggGGGGggGGGTG---GGGATTGCTGGCGGCTCCTCCTGCAGGGAGGGATACTG---GGAAGCCAAGGCGGTGGC----------------------------------------------CGCCAGGCCCTG-TGAGGGAGCTGGGTGCGAGCAGGGGCGTTTGCCCAG-----------------------------------CCGCAGG----AGGAGCGTG-----------------CCTAGTCCAGAGAGAAGGT | |
| hetGal2 | Unknown | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | scaffold_13715:16490-16569 + | TCCTGCAGCTCTCA---------------------------------------------------------------------------------------------------------------------------------------------------------GGAGGCC----------------------TCAGCCCCAG-G---------GTGGGAGTACAGAGGCTTCTGTTC---T-------------------GGTGGGTT-----------------------------------------------CCAGCGTGCCGCTGT | |
| oryCun2 | Unknown | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| rn5 | chr19:66142264-66142485 - | CCATCCCAATCTGTGGAGGCGG---GTGTGGACCTGGGCATTGAGTGAGGATCCCAGCTGACTAGGAGGTGAAGGGGCCAGTGTTT---CAGACCACTGGGGCCT----------------------------------------------------CTCTGACTAAGGGGAGCTATCCTGGGC--GGGAGAGGTGTCAGGGGTTC-CTGGGGC------------------------------CTTAGCTCCTAGTCCTGGACTATGTCTACAGGAAGAACAGG----GTGAACTAG-----------------GTCATCCCCCAATAGCCAA | |
| speTri2 | JH393451:880811-881002 + | CCGCGGGTG---------GCCAAGGGCT-GGGCCCA--------------------------------------------------------------------CCCACCTGGGCCAAGCGGCAGTGAGGCGAGCTCTGGGCTGCGGCCCAGGCAGCCTCCAGCTGGGGAGGCC----------------------TGGGGTGGAG-T---------CCGTGGGCCCAGGAGCTTCCCTCCAGCT-------------------GGTGGCTTCGGA---------GGAGG----TTTATCCTGTCCAGAGAAGGGTTTGCCCCGAAGGGGCTGT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 05/01/2015 at 01:37 AM