
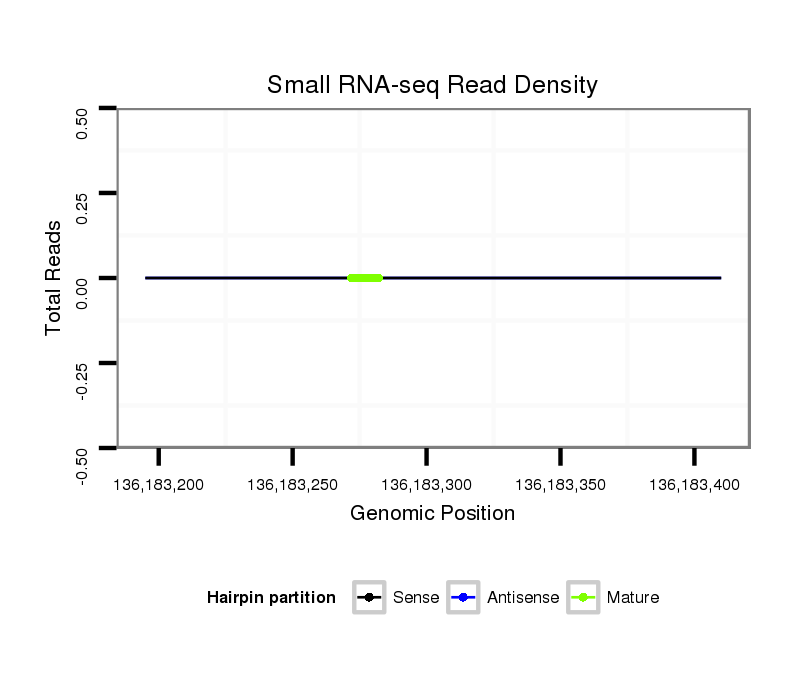
ID:mmu-mir-6383 |
Coordinate:chrX:136183245-136183360 + |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -36.1 | -35.9 | -35.8 |
 |
 |
 |
|
CTGACTGAAGTCACATTGTAAGTGATCCTGGCAAGGAACTTCCAGTTATCCAGCATGTGGAACACTGTAAAGTGCTGTGTCACGGACACTGACTTTTCAAAGTCCAGTAAGTCAGTCTGCTTAGAGGTCAGTGGGCAATACTATGCTATGTTCTGGCCCCATGTTTACTCATTCTGACATCTGTACTCTGCTTTGCTGACTTTCTACTTTGTATCT
***********************************..((((.((((..((((.....))))..))))..))))...(((((((((((..((((((.............)))))))))))......((((((..((((......)))).....))))))..(((......)))..)))))).*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345202 brain |
SRR345198 brain |
SRR345201 brain |
SRR345197 brain |
SRR345203 brain |
SRR391848 embryo |
SRR042480 kidney |
SRR391845 embryo |
SRR065053 jejunum |
SRR073955 blood |
SRR345200 brain |
SRR345204 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................TGGAACACTGTACAGCGCTGT.......................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TGCAAGTGATCCTGGTAAGG.................................................................................................................................................................................... | 20 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TGGAACATTGTAAAGTGCTTT.......................................................................................................................................... | 21 | 2 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TGGAACACTGTACAGTGCTTTGA........................................................................................................................................ | 23 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TGGAACACTGTATAGTGCTTT.......................................................................................................................................... | 21 | 2 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................GATGTGGAACACAGCAAAGTGCT............................................................................................................................................ | 23 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................TTCCAGTAATCCAGCATATG............................................................................................................................................................. | 20 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TCGAACACTGTATAGTGCT............................................................................................................................................ | 19 | 2 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......GAAGTCACATCGTAAG.................................................................................................................................................................................................. | 16 | 1 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................TGGAACACTGTACAGTGCTTT.......................................................................................................................................... | 21 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TGGAACACTGTATTGGGCTGT.......................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TGGAACACTGTACAGCGCAGT.......................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............................................................................TGTCACGGACA................................................................................................................................ | 11 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................GCTTAGAGGTCATGGGGC................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................TTCCAGTAATCCAGCAT................................................................................................................................................................ | 17 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
GACTGACTTCAGTGTAACATTCACTAGGACCGTTCCTTGAAGGTCAATAGGTCGTACACCTTGTGACATTTCACGACACAGTGCCTGTGACTGAAAAGTTTCAGGTCATTCAGTCAGACGAATCTCCAGTCACCCGTTATGATACGATACAAGACCGGGGTACAAATGAGTAAGACTGTAGACATGAGACGAAACGACTGAAAGATGAAACATAGA
***********************************..((((.((((..((((.....))))..))))..))))...(((((((((((..((((((.............)))))))))))......((((((..((((......)))).....))))))..(((......)))..)))))).*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345202 brain |
SRR037929 embryo |
SRR345200 brain |
SRR345197 brain |
SRR345196 brain |
SRR345199 brain |
SRR345201 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................................................................AAATGAGTACGACTGTAGGC................................. | 20 | 2 | 2 | 1.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................CTAGGGCTGTTCCTTGAAG.............................................................................................................................................................................. | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................AAGCTCTAGTCACCCGATATGAT......................................................................... | 23 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................ATTTCACGACAAAGTGC.................................................................................................................................... | 17 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................................................................AACGACTGAAAGATGAAT...... | 18 | 1 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................................................................AGGCCGGGGTACGAATGAG.............................................. | 19 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................CCGCCAGACAAATCTCCAGTC..................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chrX:136183195-136183410 + | mmu-mir-6383 | CTGACTGAA---GTCA-----------------CAT-TGTAAGTGATCCTGGCAAGGAACTTCCAGTTATCCAGCA---------TGTGGAACACTGT------AAAGTGCTGTGTCACGGACACTGACTTTTCAAAGTCCAGTAAGTCAGTCTGCTTAGAGGTCAGTGGGCAATACTATGCTA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTTCTGGCCCCATGTTTACT-CATTCTGACATCTGTACTCTGCTTTGCTGACTTTCTACTTTGTATCT |
| cavPor3 | scaffold_26:1717278-1717405 - | TTGTTTGAA---CTCAGAGATGAATTCTGTACACAC-TGT---TATTTATGGAAAAGAATTTACAGTTAACCAGAGCATAACGCTTCTGGATTACTGTGAACCTAAGGTTCTGTGTCATAACCACTATCATTTCA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dipOrd1 | Unknown | CTGACTAAA---CTCGCAACTGAATGTTCCACATATTTGTGGATGATAGTGGCAAGGGATTTCTAATGATCCAAGTTGTAATACTTATAAATTTCTGTGCACTTAAAGTGCTGTGTCATGACCACTGCCATTTCAAACGTCCACAAGTCAGCCTGCTTAGATTTTAATATGTAACATTATGCTCAATGTCTACCCTATTAAGATTCTTAATATTGATTGCTTATAGTTGAGAACTGCATCTAGGTAGAGTATCCTGCTAGGTTGATAAGGAGGCACACTAAAGCCATGTCAGAGCTTGAGCCAGGAAACTTTTTGGTTTCTGTTTCTGTTGTTGTAGGGGAGAGAATGTGGGTGGTACACACTGAGTGCTTTAAAAATTAAATTTATAGTGGAAAGAAAGCCTTTACTGACCTTTTGAGTCTATATCAGCTCCTCATGCTACATGATTCTTTAGACCCTGTGTTGACCACATCCTAATATGTTTATCTTTT----------------TTTGGGTTT | |
| hetGal2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| ochPri2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| oryCun2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| rn5 | chrX:106399963-106400185 - | TTGACTGAAGAAGTCA-----------------CAT-TGAAAACGGTCCTGGCAAGGAGCTTCCAGCTACGCAGCA---------TGTGGAGCACTGTAAACTGAAAGTGCTGTGTCATGGGTACCGACTTCTCAAAT-TCCAGTAATCAGTCGGCTTAGATGCCAATATGCTACGTTATGCTA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTTTTGGCCCCGGGTTTACT-CATTCTAACATCTGTACTCTGT-TTGCTGACTTTCCAATTCGTGTCT | |
| speTri2 | JH393357:7971033-7971189 - | CTGTCTAAA---CTCCCAGCTGAATGATTCACACAT-TGTGGATGATTGTGGCAAGGACTTTCTAGTCATTCAGGGCATAATGCTTAAGAATTACTGTGAACCTGAAGTGCTATCTCATAATCACTGCAGTTTCAAATGTCCATGAATTGATCTGCTTAGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 05/01/2015 at 02:11 AM