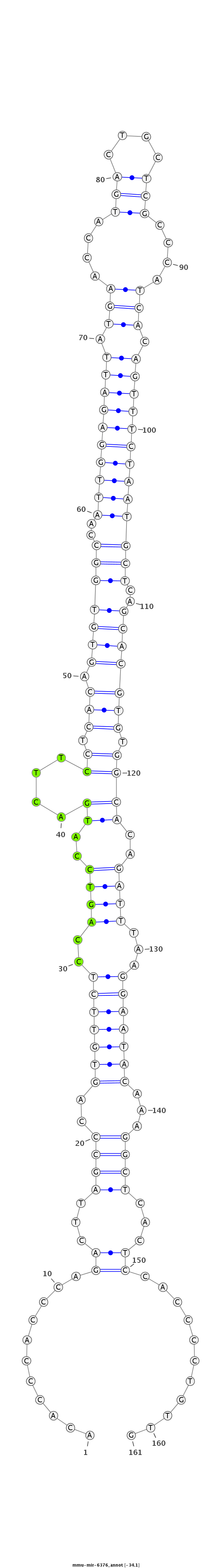
ID:mmu-mir-6376 |
Coordinate:chr6:88104128-88104258 - |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -33.9 | -33.8 | -33.7 |
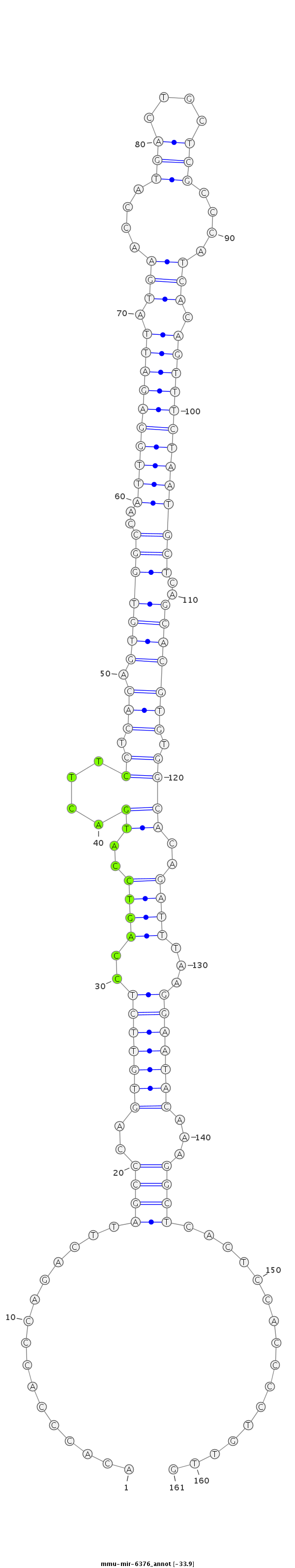 |
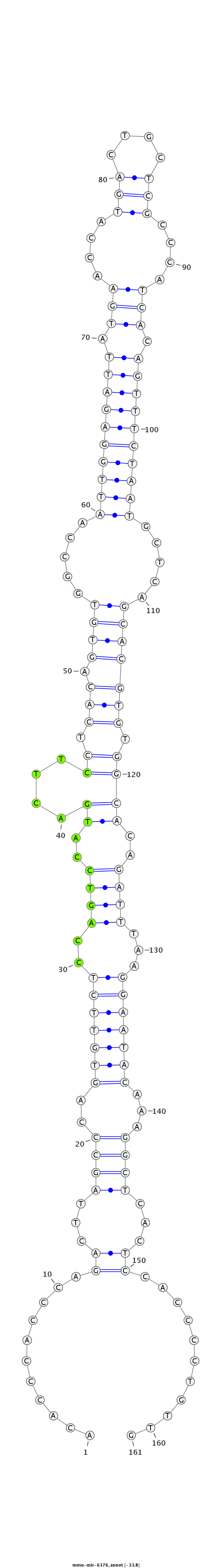 |
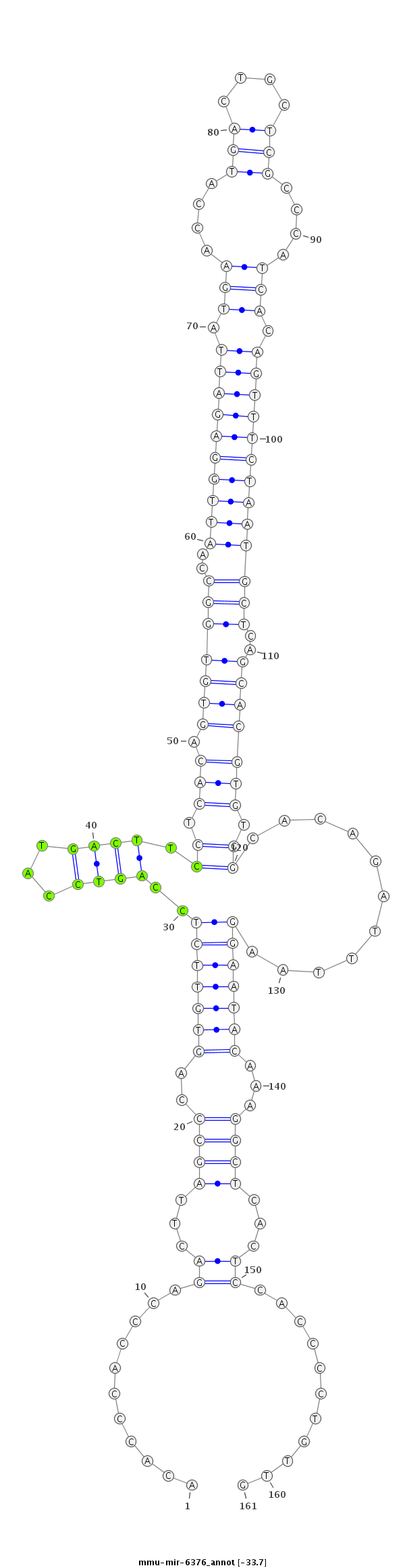 |
| mature | star |
|
AAGGCTCAGCTCTGGACTCCCCTCCCCTCAAACACACACCCACCCAGACTTAGCCCAGTGTTCTCCAGTCCATGACTTCCTCACAGTGTGGCCAATTGGAGATTATGAACCATGACTGCTCGCCCATCACAGTTTCTAATGCTCAGCACGTGTGGCACAGATTTAAGGAATACAAAGGCTCACTCCACCCCTGTTGTGTGGCCTCCGGACCTTACTGGGCCTCTCGAGACT
***********************************...........((...((((..(((((((..((((..((....((.(((.(((((((..((((((((((.(((....(((....)))....))).)))))))))))))..))))))).))))..))))...)))))))...))))...))...........*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR037929 embryo |
GSM510444 brain |
SRR345200 brain |
SRR037925 embryo |
SRR037906 brain |
SRR037923 embryo |
SRR345199 brain |
SRR037927 embryo |
SRR037928 embryo |
SRR073955 blood |
SRR345196 brain |
SRR345198 brain |
SRR116846 epididymis |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................CAGAGTTCTACAGTCCACGACTT......................................................................................................................................................... | 23 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................TGCTCGCCCATCACGGTTTA............................................................................................... | 20 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................CAGAGTTCTACAGTCCACGACT.......................................................................................................................................................... | 22 | 3 | 10 | 0.30 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 |
| ......................................................CCAGTGTTCTACAGTCCGTGAAT.......................................................................................................................................................... | 23 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................AGAGTTCTACAGTCCACGACTT......................................................................................................................................................... | 22 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................CCAGTCCATGACTTC........................................................................................................................................................ | 15 | 0 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................TATGAACCATCACTCGTCGCC........................................................................................................... | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................GTTCTACAGTCCACGACTT......................................................................................................................................................... | 19 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................TTCTACAGTCCACGACTTCC....................................................................................................................................................... | 20 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................AGATTTAAGGTATACAAA....................................................... | 18 | 1 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................CAGTGTTCTCCAGTCCGTCGC........................................................................................................................................................... | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................................ATGAACCCTGACGGCTCG............................................................................................................. | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................................................................................GCGTGGCTTCCGGAGCTTAC................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTCCGAGTCGAGACCTGAGGGGAGGGGAGTTTGTGTGTGGGTGGGTCTGAATCGGGTCACAAGAGGTCAGGTACTGAAGGAGTGTCACACCGGTTAACCTCTAATACTTGGTACTGACGAGCGGGTAGTGTCAAAGATTACGAGTCGTGCACACCGTGTCTAAATTCCTTATGTTTCCGAGTGAGGTGGGGACAACACACCGGAGGCCTGGAATGACCCGGAGAGCTCTGA
***********************************...........((...((((..(((((((..((((..((....((.(((.(((((((..((((((((((.(((....(((....)))....))).)))))))))))))..))))))).))))..))))...)))))))...))))...))...........*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR014235 testes |
SRR345200 brain |
SRR345201 brain |
SRR073954 blood |
SRR345197 brain |
SRR345198 brain |
SRR037925 embryo |
SRR391845 embryo |
SRR037901 testes |
SRR345202 brain |
SRR345205 brain |
SRR391848 embryo |
SRR391850 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................................................................................ATGTTTCCGATGGAGGTGGGGACAAT................................... | 26 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................CTGAATCGGGTCACGAGAG...................................................................................................................................................................... | 19 | 1 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................AGGTCAGTTGCTGAAGGAGT.................................................................................................................................................... | 20 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................GGTCAGCTGCTGAAGGAGT.................................................................................................................................................... | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................TGGGTGGGGCTCAATCGGG............................................................................................................................................................................... | 19 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................GGTCAGTTGCTGAAGGAGT.................................................................................................................................................... | 19 | 2 | 20 | 0.10 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................................................................................................GCAGCCTGGAATGGCCCG........... | 18 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................TGGCGAGCCGGTAGTGTCA.................................................................................................. | 19 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................ATGAGCCGGAGAGCT.... | 15 | 1 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................TAGGTACTGGCGAGCGGG.......................................................................................................... | 18 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................AGCTTGTGTGTGGGCGGGT......................................................................................................................................................................................... | 19 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................AGGTCAGCTGCTGCAGGAGT.................................................................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................AGGTCAGCTCCTGCAGGAGT.................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................GCGGGTAGTGT.................................................................................................... | 11 | 0 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................TGTCAAATCTTACGAGTCGT................................................................................... | 20 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................................................................................................................................GAGTGAGGTGGGGA....................................... | 14 | 0 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................TAGGTCAGTTGCTGAAGGAGT.................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................TTTCCTAGTGAGGTGGG......................................... | 17 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TACTTGGTACTGACAAGAG............................................................................................................ | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................................................................................................................AGCGGGTAGCGTCA.................................................................................................. | 14 | 1 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr6:88104078-88104308 - | mmu-mir-6376 | AAGGCTCAGCTCTGGACTCCCCTCCCCTCAAACACACACCCACCCAGACTTAGCCCAGTGTTCTCCAGTCCATGACTTCCTCAC-----AGTGTGGCCAATTGGAGATTATG----------------------------------AACCATGACTGCTC-GCCCATCACAGTTTCTAATGCTCAGCACGTGTGGCACAGATTTAAGGAATACAAAGGCTCACTCCACCCCTGTTGTGTGGCCTC-CGGACCTTACTGGGCCTCTC------------------------GAGACT |
| cavPor3 | Unknown | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dipOrd1 | Unknown | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| hetGal2 | Unknown | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | Unknown | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| oryCun2 | chr9:7522328-7522401 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCCACTCCTGGTGTGTGGTCTTACGGAGGCCACTCGACCTCTCCTATTGTGCTAGTTAGTACTTGCTAATACT | |
| rn5 | chr4:185264710-185264978 - | ACGGTTCCGCTCTGGACTACCCTCTGCTCCCACAAACACACATCCACACTAAGCCCAGTATTCCCCAGCCCATGACTTCCTCAGAGAAGAGTATGGTCAATTTAAGATTATGAATTAGAGAGGTATCTGCCTGTGACTCTCCCATGAACCATCTCTGCTCTGCCCATCACAGCTTCCAATGCTCAGCATGAGTGGC--AGATTTAAGGAACACAAAGGCCCACTCCACCACTGCTGTGTGGCCTT-TGGGCCTCACTGGACCTCTT------------------------GAGACT | |
| speTri2 | Unknown | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 05/01/2015 at 12:32 AM