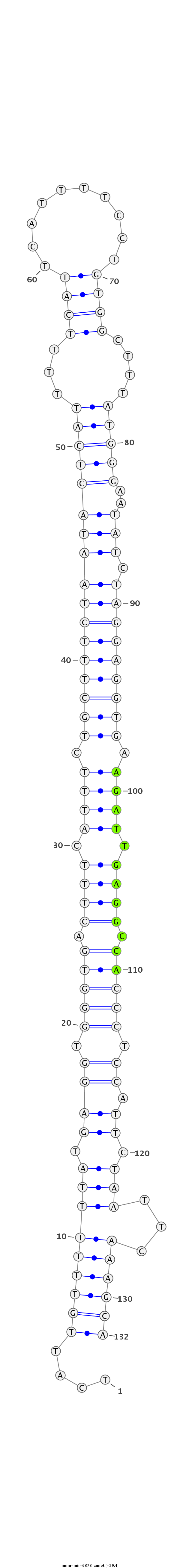
ID:mmu-mir-6373 |
Coordinate:chr6:102794384-102794485 + |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -29.3 | -29.3 | -29.2 |
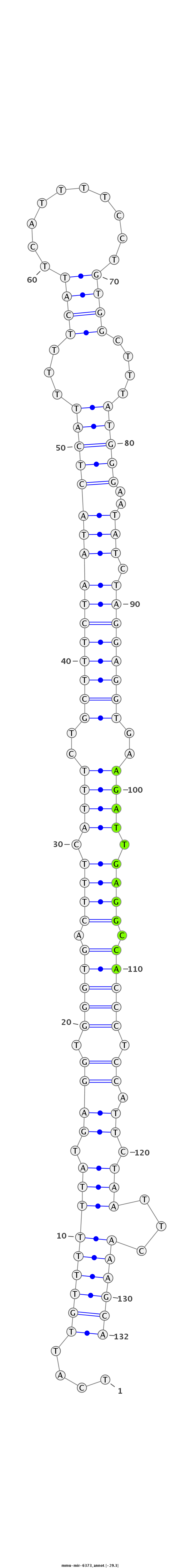 |
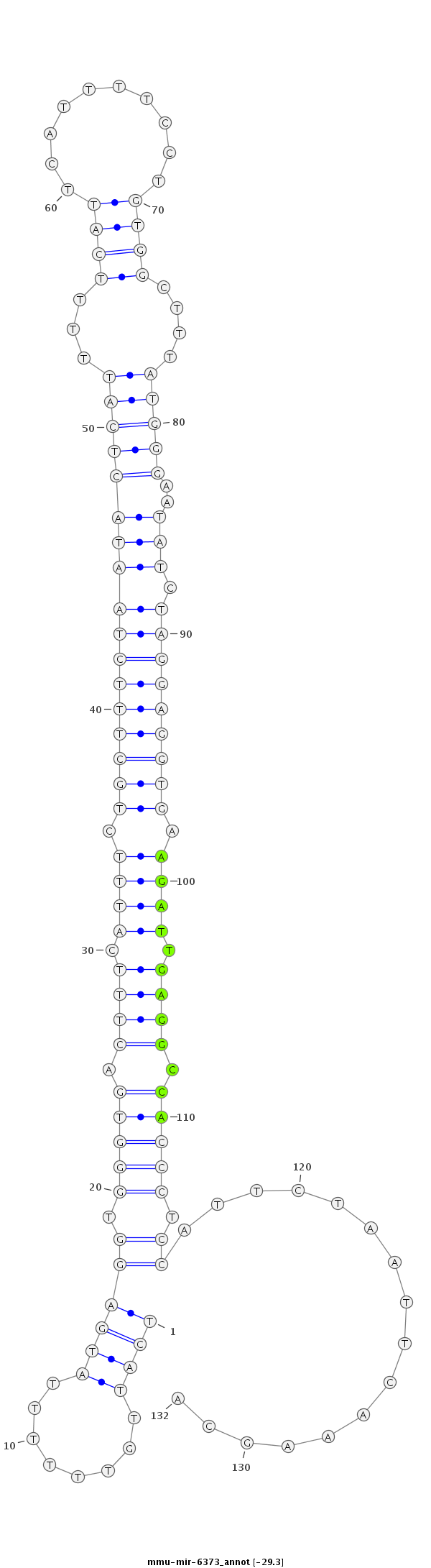 |
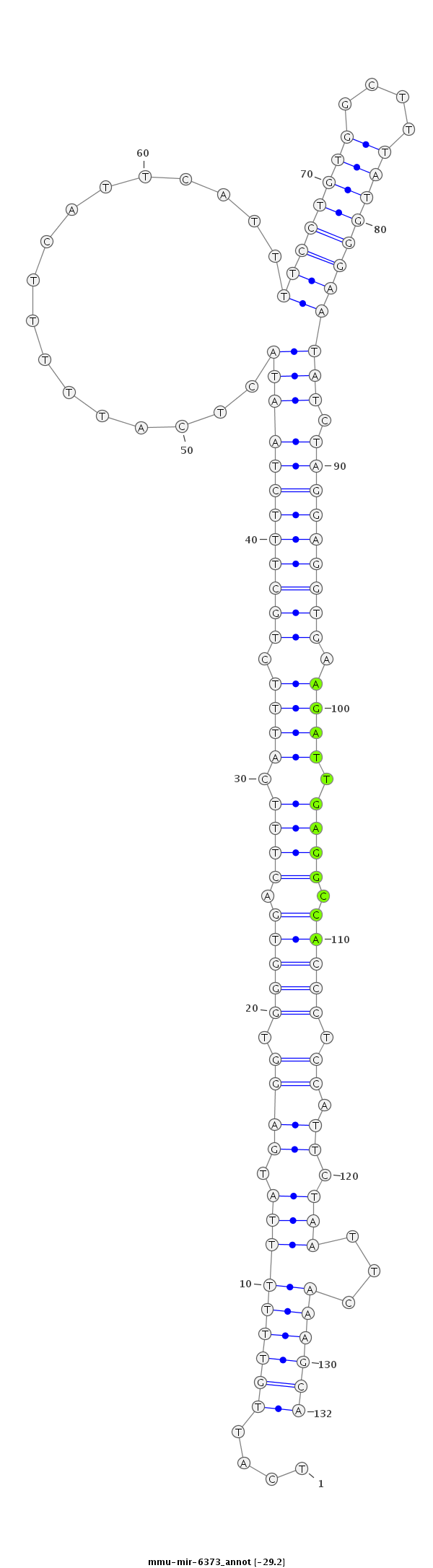 |
|
ACTACTCTACAACTTAGGTTATTTCAGTACTATTGTCATTGTTTTTTATGAGGTGGGTGACTTTCATTTCTGCTTTCTAATACTCATTTTTCATTCATTTTCCTGTGGCTTTATGGGAATATCTAGGAGGTGAAGATTGAGGCCACCCTCCATTCTAATTCAAAGCAGGGGAAGCTCCCATAGTGGTCTTAGGATTCGTTCA
***********************************....(((((((((.((((.(((((.((((.((((.(((((((((((((((((...((((..........))))....)))))..))).))))))))).)))).)))).))))).)).)).)))...))))))*********************************** |
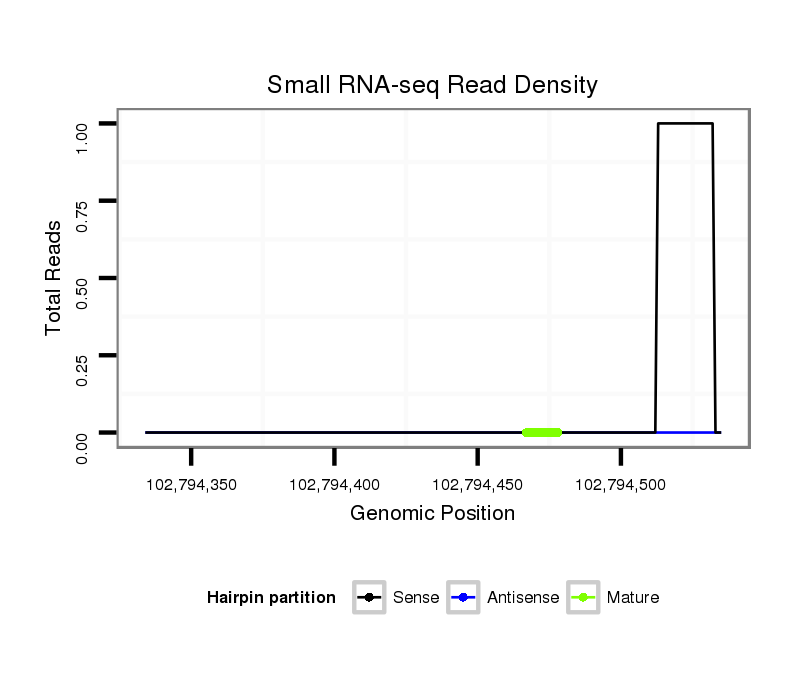
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR039154 muscle |
SRR116846 epididymis |
SRR391851 embryo |
SRR095855BC2 heart |
SRR345196 brain |
SRR345199 brain |
SRR345200 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................................................................................ATAGTGGTCTTAGGATTCGT... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................ATGGGATTATCTAGTAGGT....................................................................... | 19 | 2 | 17 | 0.18 | 3 | 0 | 3 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................GAGGAGGTGAAGATTGA.............................................................. | 17 | 1 | 18 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................AGATTGAGGCCA......................................................... | 12 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................................GGTGACGATTGAGGC........................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................GAGGTGGGTTACTTTC......................................................................................................................................... | 16 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ............................................................................................................................................................................................TTAGGATTCG.... | 10 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
TGATGAGATGTTGAATCCAATAAAGTCATGATAACAGTAACAAAAAATACTCCACCCACTGAAAGTAAAGACGAAAGATTATGAGTAAAAAGTAAGTAAAAGGACACCGAAATACCCTTATAGATCCTCCACTTCTAACTCCGGTGGGAGGTAAGATTAAGTTTCGTCCCCTTCGAGGGTATCACCAGAATCCTAAGCAAGT
***********************************....(((((((((.((((.(((((.((((.((((.(((((((((((((((((...((((..........))))....)))))..))).))))))))).)))).)))).))))).)).)).)))...))))))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR039152 muscle |
SRR345201 brain |
SRR345203 brain |
SRR059769 spleen |
SRR345200 brain |
SRR345202 brain |
SRR345199 brain |
SRR553585 kidney |
SRR077866 liver |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............ATCCAATAAAGTCATGACA......................................................................................................................................................................... | 19 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TGAATCCAATAGAGGCATGACAA........................................................................................................................................................................ | 23 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............ATCCAATAAAGTCAAGATA......................................................................................................................................................................... | 19 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................ACCGAAATACCCTAATCGA.............................................................................. | 19 | 2 | 20 | 0.15 | 3 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 |
| ............................................................................................................................................CCGGTGGGAGGTAAGATGAT.......................................... | 20 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............GAAGCCAATAGAGTCATGACAA........................................................................................................................................................................ | 22 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............ATCCAATAGAGTCATGACAA........................................................................................................................................................................ | 20 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................GTAAAGACGAAAGATT.......................................................................................................................... | 16 | 0 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........................................................................................................ACCGAAATACCCTAATCG............................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr6:102794334-102794535 + | mmu-mir-6373 | A-CTACTCTACAACTTAGGTTATTTCAGTACTATTGTCATTGTTTTTTATGAGGTGGGTGACTTTCATTTCTGCTTTCTA-ATACTCATTTTTCATTCATTTTCCTGTG-----------GCTTTATGGGAATATCTAGGAGGTGAAGATTGAGGCCACCCTCCATTCTAATTCAAAGCAGGGGAA--GCTCCCATAGTGGTCTTAGGATTCGTTCA |
| cavPor3 | scaffold_16:26788634-26788840 + | AACTGTT----ACCCAAGATTATGCCATTTCTGTCAATATCATCTTTGACAAGTTGTATAGCTATTGTTTTTATATGCAACATAATCATTTTTTCCATATG-----GTA-ATTGTATCTGATTCTCTTGTTGTTTCTAGGTAGTTAACAGCAAGACTACCCTCTACCTTAGTTCAAAGCATAGACACATGATCTGTCTAGGTGGAAGGACTGACTCA | |
| dipOrd1 | scaffold_2418:64441-64564 - | A-TTACTCCCTAACCTAGT---------------------------------------------------------------------------------------ATTTGTTATATGGATTTTCCTTCTTTTTTCTAGATGGTTGAGTGTAGGTG---CCTCAATTATAGCCCAAGATAAGCACA--TAAACTGTCTTGGTCACAAGATTGATTCT | |
| hetGal2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | scaffold_6150:3791-3988 + | C----------AACAAACGTTATGTTAA--CGACGGTTAATATTTATTTCAAGGTGGGAAGAT--GCTTGCTGTTTGTCAAACAGCCATTCTCTGCTTTGG-----ACATCTTACTTTGAATTTCCTTCTGGTACCTGGGTGATTAAGAGTGAAGTTGTTCTCATCTTTAGATCAAAGCACAGGCACATGACCTCTCTTGACCACCAGGCAGGTACA | |
| oryCun2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| rn5 | chr4:199914145-199914347 - | A-CTACTCTGCAATTTAGATTATTTCAGTACTATTGTCATTGTGTTTGATGAGGTGGGTGACTTTTATTTCTGCTTTCCAAATACTCATTTTCTCCTCATTTTCCTGTG-----------GTTTTATGTGAGTTTCTAGGTGGTTAAGACTGAGTCTGTCCTTCATTCTAGTTCGAAGCAGAGAAA--CCTCCTATCTTGGTTTTAGGATTCATTTA | |
| speTri2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 05/01/2015 at 12:36 AM