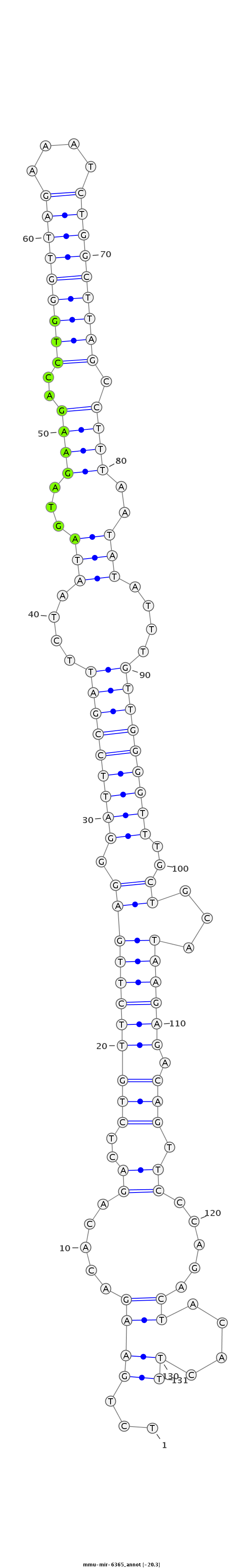
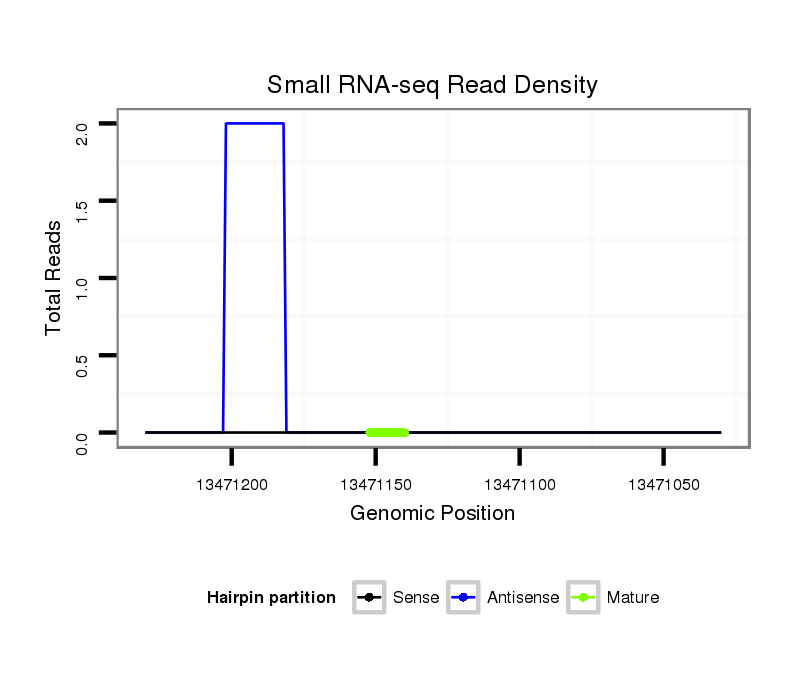
ID:mmu-mir-6365 |
Coordinate:chr16:13471080-13471180 - |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -28.3 | -28.3 | -28.2 | -28.0 |
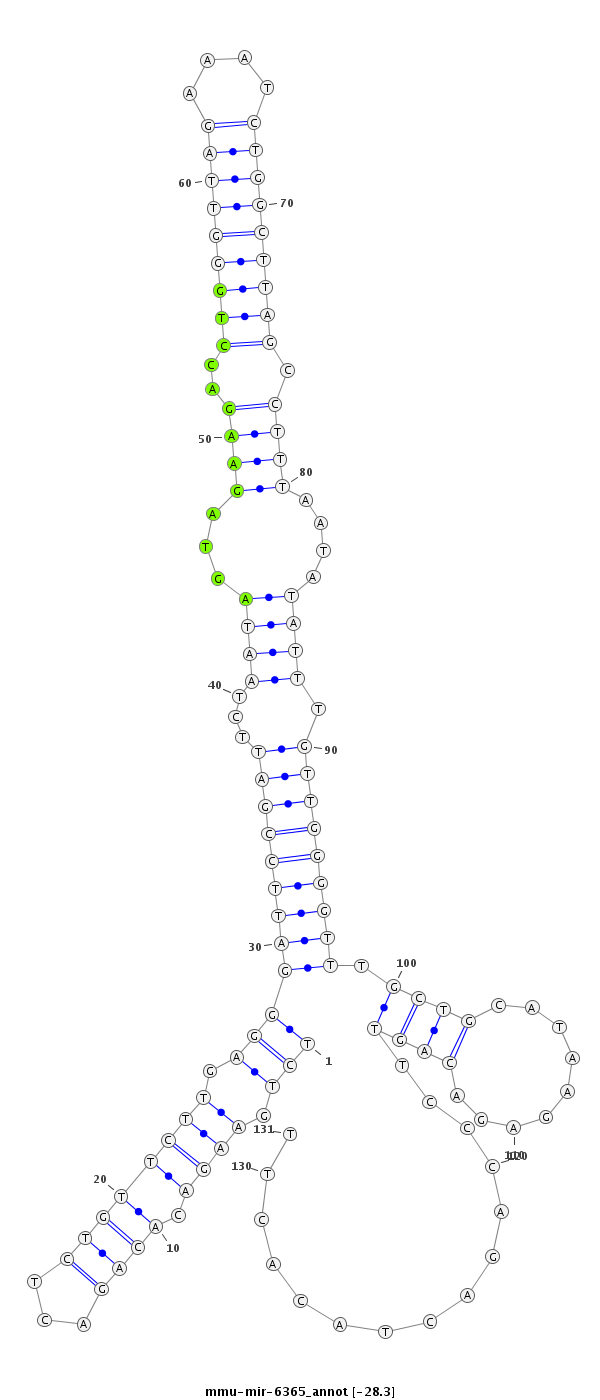 |
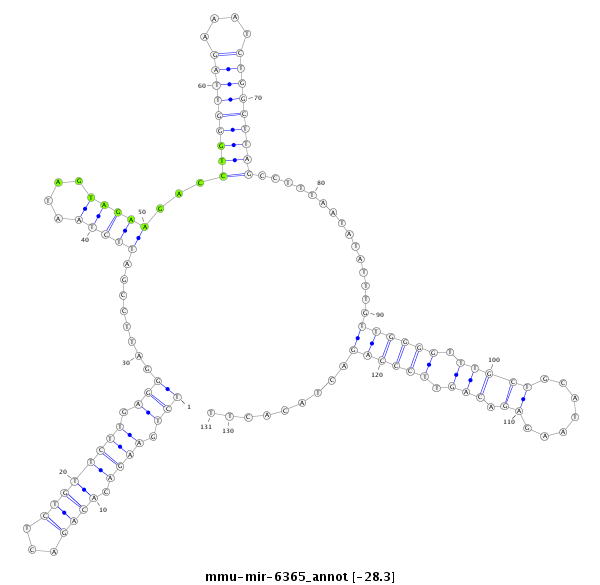 |
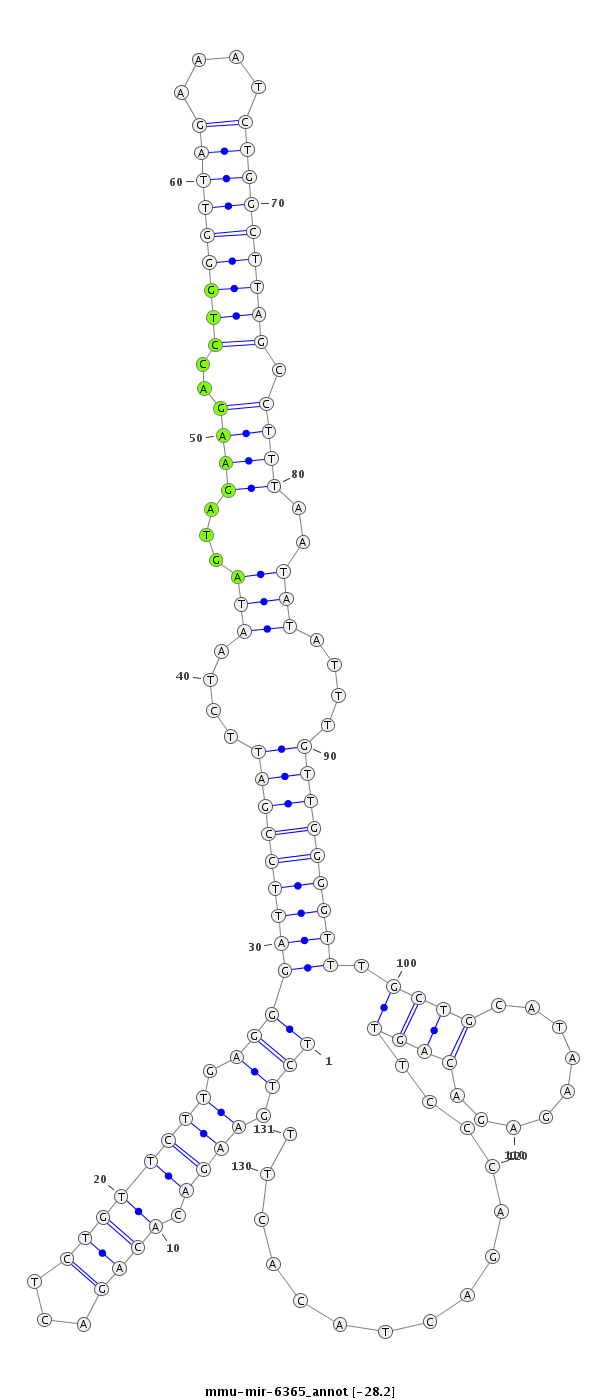 |
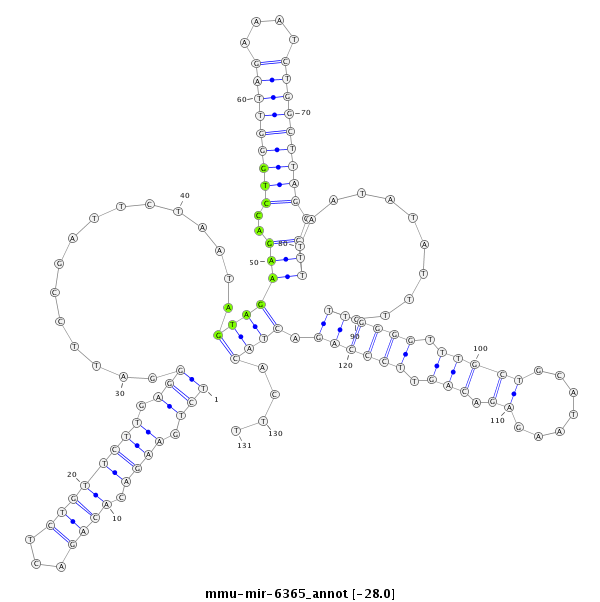 |
|
TGTTGGTATAGTTTCCTGGAGGTTCCCTACTGAGCTCTGAAGACACAGACTCTGTTCTTGAGGGATTCCGATTCTAATAGTAGAAGACCTGGGTTAGAAATCTGGCTTAGCCTTTAATATATTTGTTGGGGTTTGCTGCATAAGAGACAGTTCCCAGACTACACTTTAAAGCAGCCTAGAGTGAATGGTTTTATGCACCAA
***********************************...((((.....((..(((((((((((.(((((((((....(((...((((..(((((((((....))))))))).))))..)))....)))))))))..))...)))))).))).)).....))....))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR073954 blood |
SRR553602 testes |
SRR040488 liver |
SRR039152 muscle |
SRR345197 brain |
SRR345199 brain |
SRR345202 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................CACTTTAAAGCAGCTTAGA..................... | 19 | 1 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................GGTTAGAAATCTGGCAGAGCCTTC...................................................................................... | 24 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................ATATTTGATGGGGTTTGCT................................................................ | 19 | 1 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................AGGGATTCCGATTATAAC........................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................AGTAGAAGACCTG.............................................................................................................. | 13 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............................................................................................................................................................CACACTTTAAAGCGGCCTTGA..................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........................................................................TCTAATAGTAGAAACCCTG.............................................................................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
ACAACCATATCAAAGGACCTCCAAGGGATGACTCGAGACTTCTGTGTCTGAGACAAGAACTCCCTAAGGCTAAGATTATCATCTTCTGGACCCAATCTTTAGACCGAATCGGAAATTATATAAACAACCCCAAACGACGTATTCTCTGTCAAGGGTCTGATGTGAAATTTCGTCGGATCTCACTTACCAAAATACGTGGTT
***********************************...((((.....((..(((((((((((.(((((((((....(((...((((..(((((((((....))))))))).))))..)))....)))))))))..))...)))))).))).)).....))....))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR116846 epididymis |
SRR042471 spleen |
SRR039154 muscle |
SRR065059 pancreas |
SRR345198 brain |
SRR039153 muscle |
SRR345199 brain |
SRR345200 brain |
SRR391845 embryo |
SRR391847 embryo |
SRR391853 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................TGACTCGAGACTTCTGTGTCT........................................................................................................................................................ | 21 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............ACGGACCTTCAAGGGATGA.......................................................................................................................................................................... | 19 | 2 | 20 | 0.55 | 11 | 10 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..........CAAAGGACCGTCAAGGGATGA.......................................................................................................................................................................... | 21 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............ACGGACCGCCAAGGGATGA.......................................................................................................................................................................... | 19 | 2 | 13 | 0.31 | 4 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TACGGACCTCCAAGGGATG........................................................................................................................................................................... | 19 | 2 | 20 | 0.20 | 4 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........TAAGGACCTTCAAGGGATGA.......................................................................................................................................................................... | 20 | 2 | 17 | 0.18 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............CGGACCGCCAAGGGATGA.......................................................................................................................................................................... | 18 | 2 | 20 | 0.15 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TAAGGACCGTCAAGGGATGACTC....................................................................................................................................................................... | 23 | 3 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TGAGGACCTCCAAGGGATGA.......................................................................................................................................................................... | 20 | 2 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............AAGGACCCCCAAGGGATAAC......................................................................................................................................................................... | 20 | 2 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................CCCAATCTTAAGATCGAAGCGG......................................................................................... | 22 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........CAAAGGCCCTCCAAGG............................................................................................................................................................................... | 16 | 1 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........AACGGACCGCCAAGGGAT............................................................................................................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........CAAGGACCGCCAAGGGAT............................................................................................................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................................................................................................CGGATCTCACT................. | 11 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........AACGGACCGCCAACGGATGA.......................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TAAGGACCTTCAAGGGATG........................................................................................................................................................................... | 19 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................CCCTAAGGCTGAGACTAT.......................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................GAACTCCCTAAGGCAAC................................................................................................................................ | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr16:13471030-13471230 - | mmu-mir-6365 | TGTTGGTATAGT---TTCCTGGAGGT------------------TCCCTACTGAGCTCTGAAGACACAGACTCTGTTCTTGAGGGATTCCGATTCTAATAGTAGAAGACCTGGGTTAGAAATCTGGCTTAGCCTTTAA--------------------TATATTTGTTGGGGTTTGCTGCATAAGAGACAGTTCCCAGACTACACTTTAAAGCAG--CCTAGAGTGA-------ATGGTTTTATGCACCAA |
| cavPor3 | scaffold_4:33169789-33169985 + | TGTTGATATCTCTGTTCCCCAAAGTCCCCACACTCAGGAGTTAT------------------------------GTCCTCACCAGTTCCTGCCTCTAA---TGGCAGATCAGGGTGGGAATCCTGGCAC--CCTCTCA-----------------TGGAGTATTTGTTAGCTTTTGCTGTGTAAGCAGCCCCTCAAATGTAATAATGTCAAACAG--CCAAGTGCGATTTTTCCACGATTTTGTGGGTGAA | |
| dipOrd1 | Unknown | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| hetGal2 | Unknown | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | Unknown | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| oryCun2 | Unknown | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| rn5 | Unknown | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| speTri2 | JH393311:2618976-2619201 - | CGTGGGTATCTCTT-TCCCTGGAGAG------GAAAGGGGCTGTGTCCTCTTCAGCTCGGTGGCTACAGTCTCTGCCTCGGAGAAGCCCAGATTCTAGAAATGGGTGACCAGGGTTAGAATCCCTCCCTGGCCTCTGGATGTtTgTGTTtGggTgTGggGgGTttgGtGgttTgtggt------------------tttTGTtGTtGgTTttttTttgGGttTGTGtTTTTTTgtTGtTggTgtGGGTGGG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 04/30/2015 at 08:30 PM