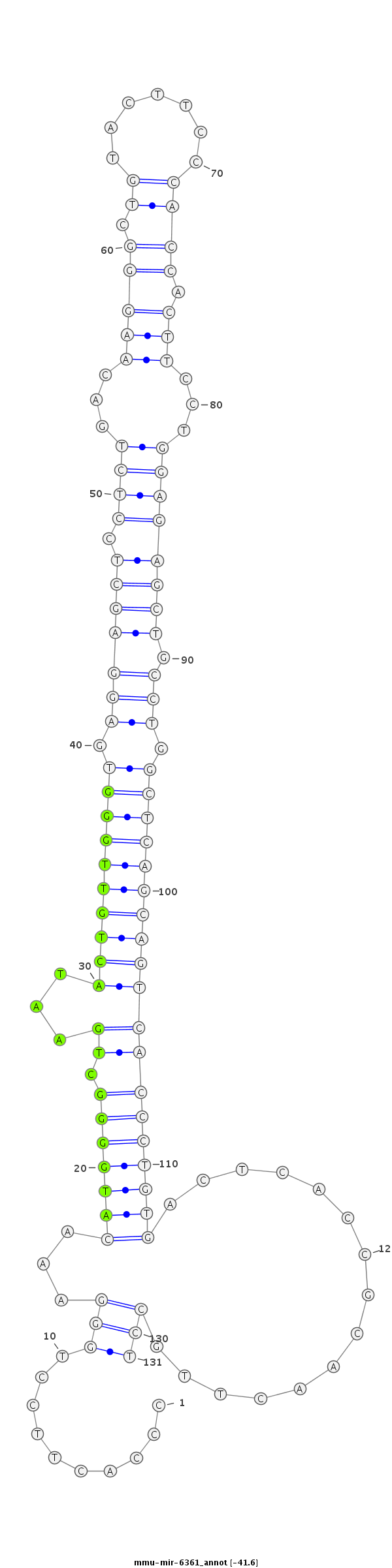
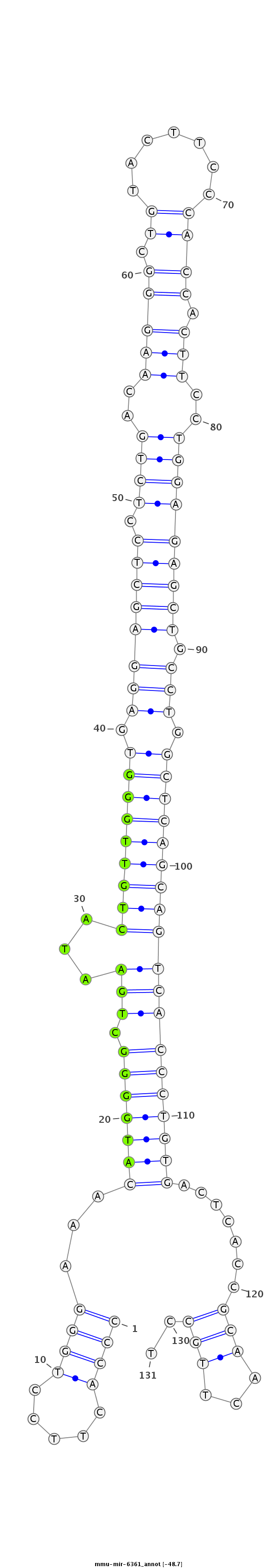
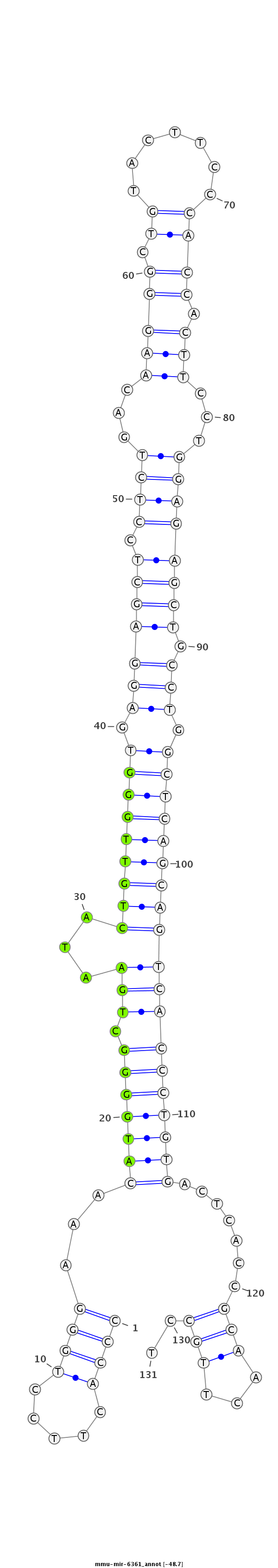
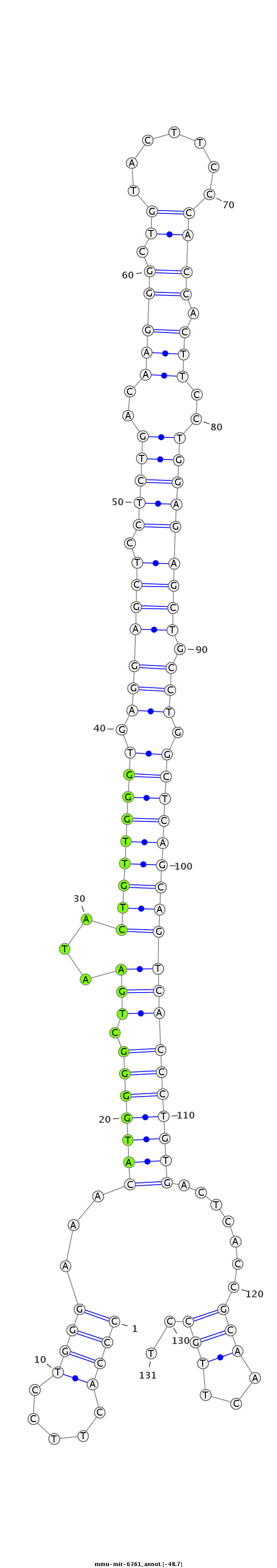
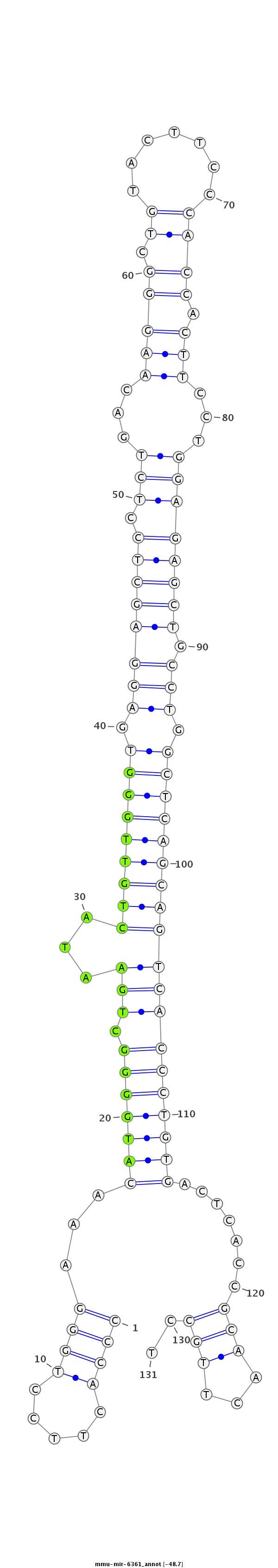
ID:mmu-mir-6361 |
Coordinate:chr18:32880573-32880673 - |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -48.7 | -48.7 | -48.7 | -48.7 |
 |
 |
 |
 |
|
GCTGAAGGAAAGTGAAACTCTACACCCAGCTTTCGCCCACTTCCTGGGAAACATGGGGCTGAATACTGTTGGGTGAGGAGCTCCTCTGACAAGGGCTGTACTTCCCACCACTTCCTGGAGAGCTGCCTGGCTCAGCAGTCACCCTGTGACTCACCGCAACTTGCCTGTGTCTCCTTCCTGTTCATGCAAGAGCCAACCTGG
***********************************..........(((...(((((((.((...((((((((((.(((((((.((((...(((((.((.......)))).)))...)))))))).))).)))))))))))))))))))...............)))*********************************** |
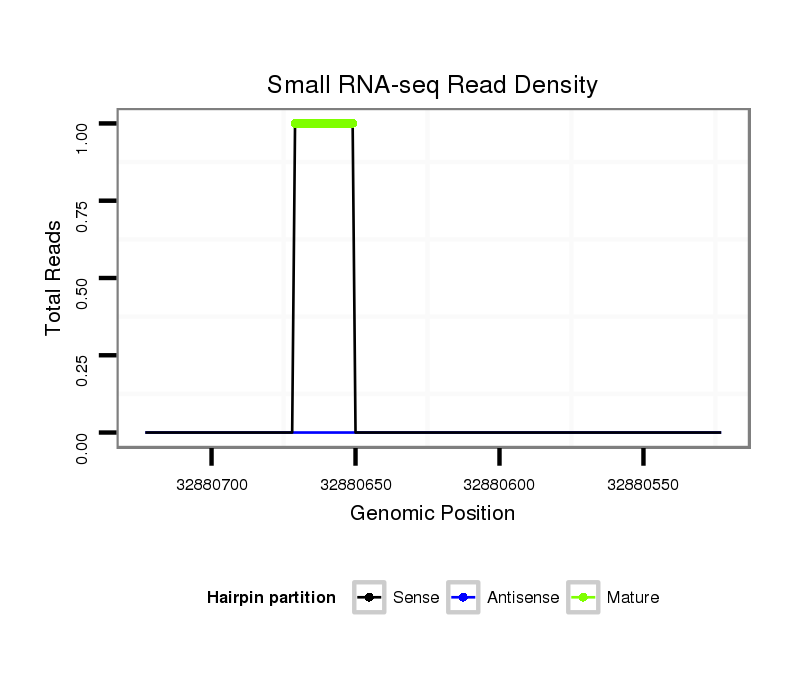
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR037924 embryo |
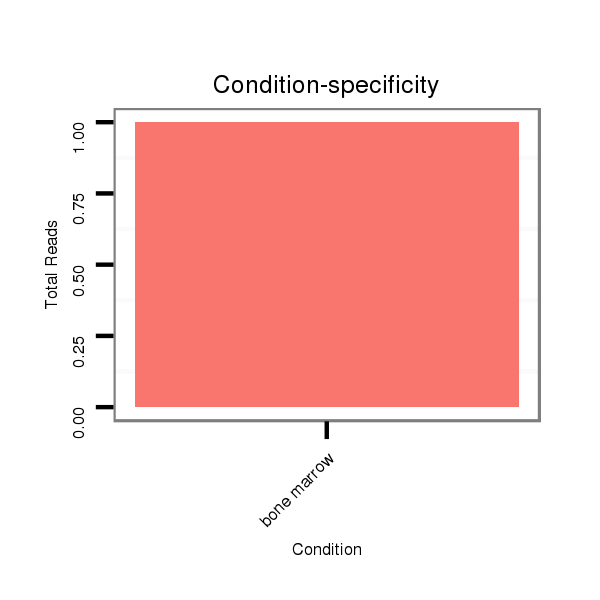
SRR042457 bone marrow |
SRR345197 brain |
SRR345198 brain |
SRR345201 brain |
SRR073954 blood |
SRR077866 liver |
SRR206941 brain |
SRR345200 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................TGGGGCAGTATACTGTTGGGT............................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................ATGGGGCTGAATACTGTTGGG................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................TGCGTGAGGAGCTCCTCTGA................................................................................................................ | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................TGCGTGAGGAGCTCCTCTG................................................................................................................. | 19 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................AGCTTTCGCCGAATTCCGGG.......................................................................................................................................................... | 20 | 3 | 10 | 0.20 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| ...................................................................................CGCGGAGAAGGGCTGTACTTC................................................................................................. | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................GACGAGGGCTGTTCTTCCCAAC............................................................................................ | 22 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................AAGCATCGGGCGGAATACTG..................................................................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CGACTTCCTTTCACTTTGAGATGTGGGTCGAAAGCGGGTGAAGGACCCTTTGTACCCCGACTTATGACAACCCACTCCTCGAGGAGACTGTTCCCGACATGAAGGGTGGTGAAGGACCTCTCGACGGACCGAGTCGTCAGTGGGACACTGAGTGGCGTTGAACGGACACAGAGGAAGGACAAGTACGTTCTCGGTTGGACC
***********************************..........(((...(((((((.((...((((((((((.(((((((.((((...(((((.((.......)))).)))...)))))))).))).)))))))))))))))))))...............)))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345202 brain |
SRR345200 brain |
SRR391850 embryo |
SRR391847 embryo |
SRR345198 brain |
SRR248526 testes |
SRR039152 muscle |
SRR042483 muscle |
SRR039153 muscle |
SRR391846 embryo |
SRR345197 brain |
SRR345199 brain |
SRR037929 embryo |
SRR039185 fibroblast |
SRR095855BC7 heart |
SRR345196 brain |
SRR345201 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................................AGTCGTCAGTGGCGCGCTGAGT................................................ | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................TCGTCAGTGGGGCACTGAGT................................................ | 20 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................AGCGGGTGAATGACCCTT....................................................................................................................................................... | 18 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................TTTGTTCCCCGTCTTATGACA.................................................................................................................................... | 21 | 2 | 6 | 0.33 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................TTGGTACCCCGACTGATGACA.................................................................................................................................... | 21 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................CGGACCGAGTCGTCAG............................................................. | 16 | 0 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................GTCGTTAGTGGGACACAGAGT................................................ | 21 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........TTCACTTTGAGATGAGGGC............................................................................................................................................................................. | 19 | 2 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................TTTGTTCCACGCCTTATGACAA................................................................................................................................... | 22 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................CGTCAGTGGCACACTGAAT................................................ | 19 | 2 | 20 | 0.10 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................................GCCGTCAGTGGCACACTGAAT................................................ | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................CTTGTACCCCGGCTGATGACA.................................................................................................................................... | 21 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................AGTGGGACACTGAGT................................................ | 15 | 0 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TGATATGTGGGTCGATGGCGG.................................................................................................................................................................... | 21 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............TTGAGATGTGCGTCGCAA........................................................................................................................................................................ | 18 | 2 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................CTTGGTTCCCCGACTGATGACA.................................................................................................................................... | 22 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................TCGTCAGTGGCACACGGAGT................................................ | 20 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................AGATGTGGGCCGAAGGCG..................................................................................................................................................................... | 18 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................CGTAAGTGGCACACTGAGT................................................ | 19 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................TCGTTAGTGGGGCGCTGAGTG............................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................................................................................................CGTCAGTGGGGCACCGAGT................................................ | 19 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................TAGGGTGGTGAAGGACC................................................................................... | 17 | 1 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............TTGATATGTGGGTGGAAGGCG..................................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................TTTGTTCCCCGCCTCATGACA.................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................GTTCTGGACATGATGGGTGGT........................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................TTTGTAGCCCGTCTGATGACA.................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................GTCGAAAGCG..................................................................................................................................................................... | 10 | 0 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................CACTGGGTGGCGTTGCTCGG.................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................ATGAAGGGGGATGAAGCACCT.................................................................................. | 21 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................CGGCAGTGGGACACT.................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr18:32880523-32880723 - | mmu-mir-6361 | GCTGAAGGAAAGTGAAACTCTACACCCAGC-TTTCGCCCACTTCCTGGGAAACATGGGGC-TG---AATACTG-TTGGGTGAGGAGCTCCTCTGACAAGGGCTGTACTTCCCACCACTTCCTGGAGAGCTGCCTGGCTCAGC-AGTCACCCTGTGACTCACCGCAACTTGCCTGTGTCTCCT---TCCT-GTTCATGCA-------------------------------AGAGCCAACCTGG |
| cavPor3 | scaffold_1:1835746-1835966 - | GCTGAC---AAGTGAAAATTTCCACC-AACATCCCTCATGCTTTCTGGGAAATCTCAGGCATGA--AACAGTG-CTTGGT-AGGATGTCTTATGAGAAGCACTGCCTTCCCCACTGCTGTCGGGATGAGTCCCTGGCTTGGCCCAAGACCCAGTGACTCATGGCTGCTC-CTTCTCTCTCAC--ATCCT-------GCTAGC-CGGCA--CAGAGGGA-AGACCAGGCTTAGCACTCACCTGA | |
| dipOrd1 | scaffold_24044:1175-1391 + | GCTGAAGGAGAGTGAAACTCTCCACCCAATATTTTTCATGCTTTCTGGGGAATCTCAGGCATGTACTGCACCGCTTAGGTGAGGATCTCTTCTGCCAAGAGCTATACTTCCCACCACTTCTTGGAGGTGTGCCTG-----------------GTGACTCATGGCCATTT-TTTG-GTTTCCT--CACTT-GCTCATGTCCAT-GGAAG--CAGATAGG-AAATGGGACTCAGAGCTCACCTGG | |
| hetGal2 | Unknown | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | Unknown | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| oryCun2 | chr11:30855979-30856215 - | GTCAAAGGAGTGTGAAACTTTCCGCCCAGTATTTCTCATGCTTCCCAGGAAATCTCAG-CACATACCACACCATCCTAGTGAGGATCTCTTCTGCCAAGAGCCTTTCTTCCCACCACTTCCTGGAGGAGTTCCTAGCTCAACCACATACCAGGTGACTCA-GGCCACCACCCTGAATCTCCCACCTCCCGATTCTTGCCACTCAGGTG--CAGAGAGG-AGACAGAG-TCAGGGATGACTTGG | |
| rn5 | chr18:25390911-25391113 - | GCTGAAGAAAAGTGAAACTCTACACCCAG-ATTCCGCCCACTTCCTGGGAAACATGGGACCTG---AACACCG-TTGGGTGAGGAGCTCCTCTAACCAGGGTTGTACTTCCCACCACTTCCTGGAGAGCTGCCTGGGTCAGC-AGACACCCTGTGACTCATGGCTACCTGCCTGTGTCTCCT--TTTCT-GTTCATGCC-------------------------------AGAGCAGACCTGG | |
| speTri2 | JH393341:5552210-5552430 - | GCTGAAAGAGAGTGAAACTTTCCACCCAACATTCCTCATGACTTCTGGGAAATCTCAGGCACGTAGCACACAGCTTAGGTGAAGATCTCTTCTGCCAAG-CCTGCCCTTCCCATCACTTTCTGGAGCAGTTCCTG-----------------GTGACTCATGGC-ATTTCCCTGCATCTGCT--CTCCCAATTCATATCAGT-GGGTGCATAGAGAGGAAAACAGGGCCCAGTGCTTACCTGG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 04/30/2015 at 08:32 PM