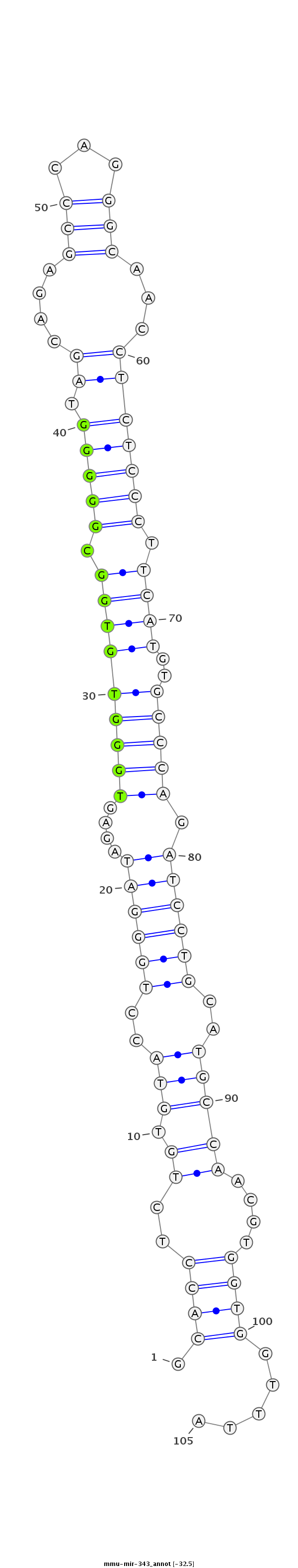
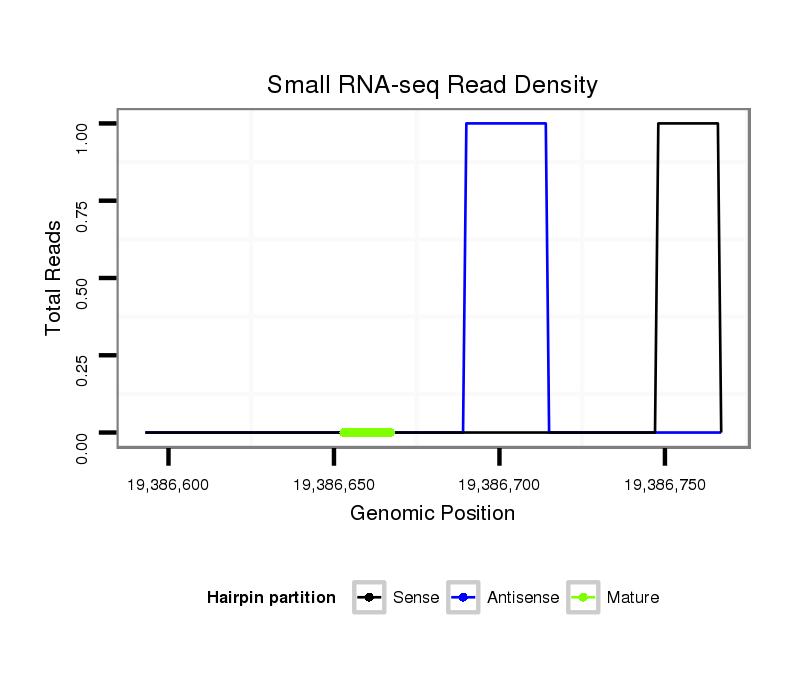
ID:mmu-mir-343 |
Coordinate:chr7:19386643-19386717 + |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -32.3 | -32.3 | -32.2 |
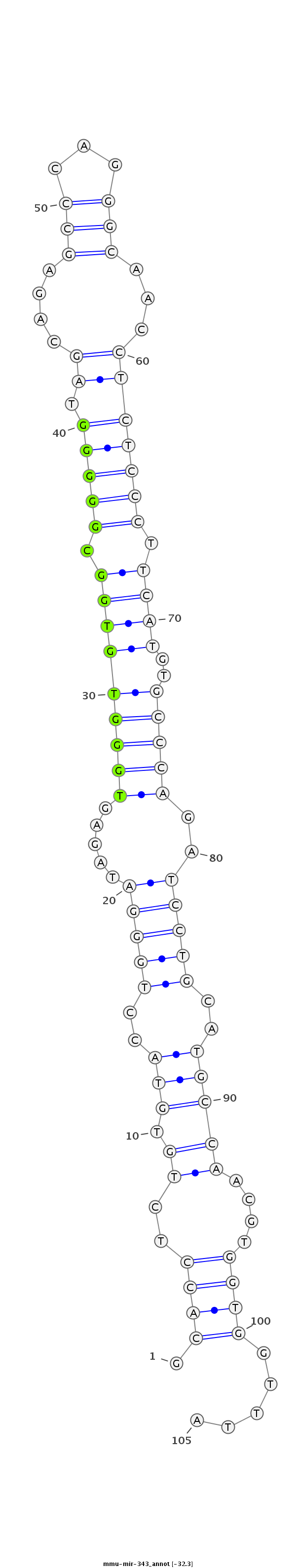 |
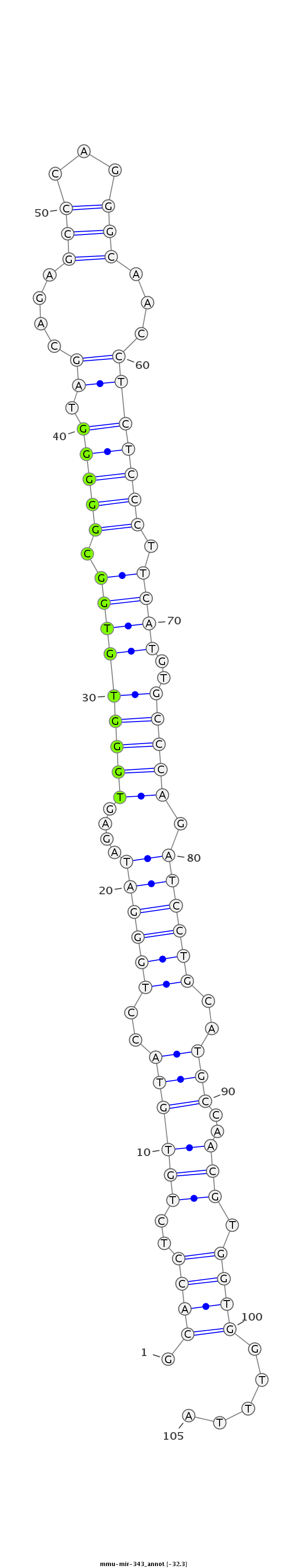 |
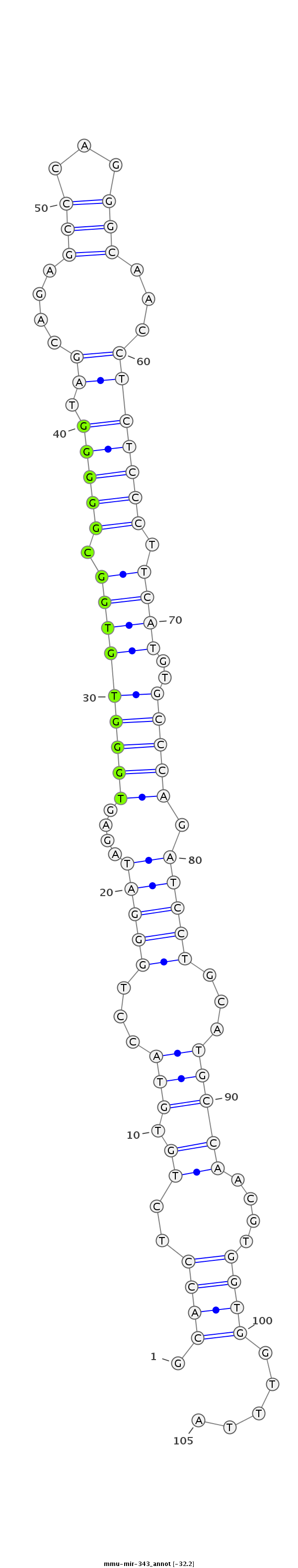 |
| mature | star |
|
CCATCTTTCCACCCGTCGTTGTGTCTGTCTTTGTGGCACCTCTGTGTACCTGGGATAGAGTGGGTGTGGCGGGGGTAGCAGAGCCCAGGGCAACCTCTCCCTTCATGTGCCCAGATCCTGCATGCCAACGTGGTGGTTTACAGCTACCACTACCTCCTGGACCCCAAGATCGCAG
***********************************.((((..((.(((..((((((....(((((((((.(((((.((....(((...)))...))))))).))))..))))).))))))..)))))....)))).....*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR391853 embryo |
SRR037901 testes |
SRR391852 embryo |
SRR014235 testes |
SRR345197 brain |
SRR037925 embryo |
SRR345200 brain |
SRR037903 testes |
SRR391847 embryo |
SRR073955 blood |
SRR116846 epididymis |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................CCTGGACCCCAAGATCGCA. | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................TGTAGCGGGGGTAGCAGGGCCCA........................................................................................ | 23 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................ACTCGATCCTGCATGCCAACGT............................................ | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................GGGACCCCAAGATCGCA. | 17 | 1 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................GCGGGGGTGGCAGAGACCAGGGAA................................................................................... | 24 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................TCGGGGGTAGCAGAGCC.......................................................................................... | 17 | 1 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....CTTTCCACCCGACGTCGT......................................................................................................................................................... | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................TGGGTGTGGCGGGGG.................................................................................................... | 15 | 0 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ACATCTCTCCACTCGTCGTTG.......................................................................................................................................................... | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................GGATAAAGTCGGTGTAGCGG....................................................................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................TCCTGCATTCCACCGTGGTGT....................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..........................................................................GTAGCGGAGCCCGGGGCA................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................AGCGTGGGGGTTTACAGC............................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
GGTAGAAAGGTGGGCAGCAACACAGACAGAAACACCGTGGAGACACATGGACCCTATCTCACCCACACCGCCCCCATCGTCTCGGGTCCCGTTGGAGAGGGAAGTACACGGGTCTAGGACGTACGGTTGCACCACCAAATGTCGATGGTGATGGAGGACCTGGGGTTCTAGCGTC
***********************************.((((..((.(((..((((((....(((((((((.(((((.((....(((...)))...))))))).))))..))))).))))))..)))))....)))).....*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR248523 testes |
SRR553584 heart |
SRR345200 brain |
SRR391847 embryo |
SRR014235 testes |
SRR073955 blood |
SRR037901 testes |
SRR391846 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................AGGGAAGTACACGGGTCTAGGACGT..................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .GGAGAAAGGTGGGCAGCA............................................................................................................................................................ | 18 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............GGCAGCAACACAGA..................................................................................................................................................... | 14 | 0 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................GGCTAGGACGTACGGTGGCA............................................ | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................GGCGCTGGAGGACCTGGTGTTC....... | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................GGACGTAAGGTTGCGCCA......................................... | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........TGCGCAGCAAAACGGACAG.................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................TCGTTGGAGAGCGAAGTA..................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................CCACACCGCCCACATCTT............................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr7:19386593-19386767 + | mmu-mir-343 | CCATCTTTCCACCCGTCGTTGTGTCTGTC--TTTGTGGCACCTCT-GTGTACCTGGGATAG---AGTGG-----GTGTGGCGGGGGT---AGCA---GAGCCC--AGG-------------GCAACC------TC--TCC--CTTCATGTGCCCAGATCCTGCATGCCAACGTGGTGGTTTACAGCTACCACTACCTCCTGGACCCCAAGATCGCAG |
| cavPor3 | scaffold_80:5852124-5852280 - | T---------------------GTCTTTC--CTTGTGTTACTTCT-GTGCAG------TGG---AGAGTGGCAGGACCCAAGTGGGC---ACTG-----G--------CAGACGGACCAAGGCAACC------TGCC--CTCCCAC---CCTCCAGATCCTGCACGCCAATGTGGTGGTATACAGCTACCACTACCTCCTGGACCCCAAGATTGCTG | |
| dipOrd1 | Unknown | ttccctttgtgcct-tgtctgtgtctctt--tctgtgtgtctTTT-CTGGACCTGTGTCCG---GTC-----AGGCCTGGTGTGGAC---ACTG-----G--------AAGGCAGACCAGGGTCACC------AT--TTT--CTCC---TTGTCAGATTCTTCACGCCAATGTAGTGGTTTACAGCTACCACTACCTCCTGGACCCCAAGATTGCTG | |
| hetGal2 | Unknown | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | Unknown | CTGTCTT---------GGCTTTGTCCCTT--CATCTGTG-CCTCG-CTCCACCTGTGTTGG---AGGGAGTCAGGT-----GTGGGGGCGACCG-----GCTC--A----GGCTGACCCGGACTG-C------TGCCATCTCCCAC---CCCCCAGATCCTGCACGCCA-TGTGGTGGTCTACAGCTACCACTATCTCCTGGACCCCAAGATCGCCG | |
| oryCun2 | Unknown | TGGCCTCTCTGC-----------------------------------------------GG---A------------------GGGA---GCGAGGAGG-CCCCGAGG-------------CCCAGCTGACGCTGGC--TCCCG-----GCCCCAGATCCTGCACGCCAACGTGGTGGTCTACAGCTACCACTACCTCCTGGACCCCAAGATCGCCG | |
| rn5 | chr1:81564960-81565134 + | rno-mir-343 | CCATCTTTCCACCCATTGCTGTGTCTGTC--TTTGTGGCACCTTC-ATGGACCTGGAGTAG---AGTGG-----GTGTGGCGGGGGG---AGCA---GGGCCC--AGG-------------GCAACC------TC--TCC--CTCCGTGTGCCCAGATCCTGCATGCCAACGTGGTGGTTTACAGCTACCACTACCTCCTGGACCCCAAGATCGCAG |
| speTri2 | JH393516:1825427-1825590 - | C-ATC--------------TGTCTCTGTCTCCTTGTGTC-CTTGGCCTCCGCCTGTGAGGCAAGAATGTGACA-----GCGGAGGGC---ACCAGGAA-GGCT--AGA-------------GCACCC------TGTC--CTCCT-----CCCCCAGATCCTGCACGCCAACGTGGTGGTTTACAGCTACCACTACCTCCTGGACCCCAAGATAGCAG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 05/01/2015 at 12:47 AM