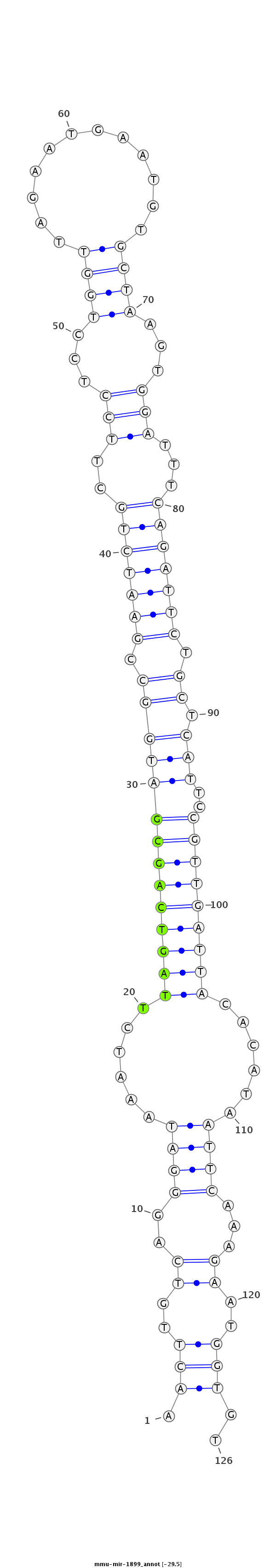
ID:mmu-mir-1899 |
Coordinate:chr9:8246786-8246881 + |
Confidence:Known |
Type:Unknown |
[View on miRBase] [View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -30.0 | -29.7 | -29.5 |
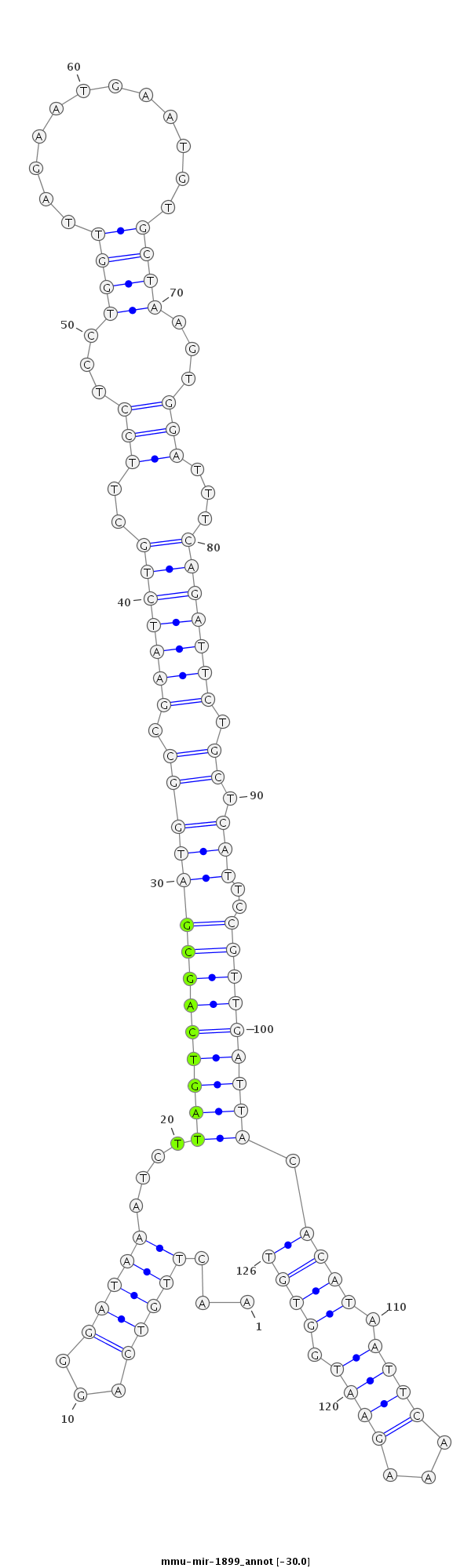 |
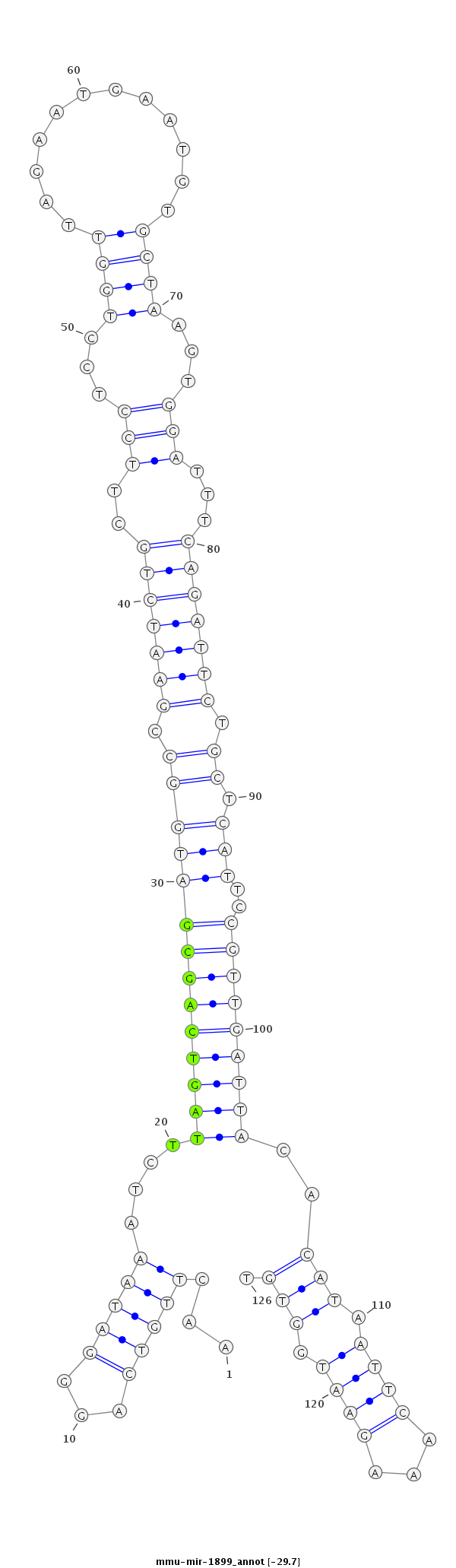 |
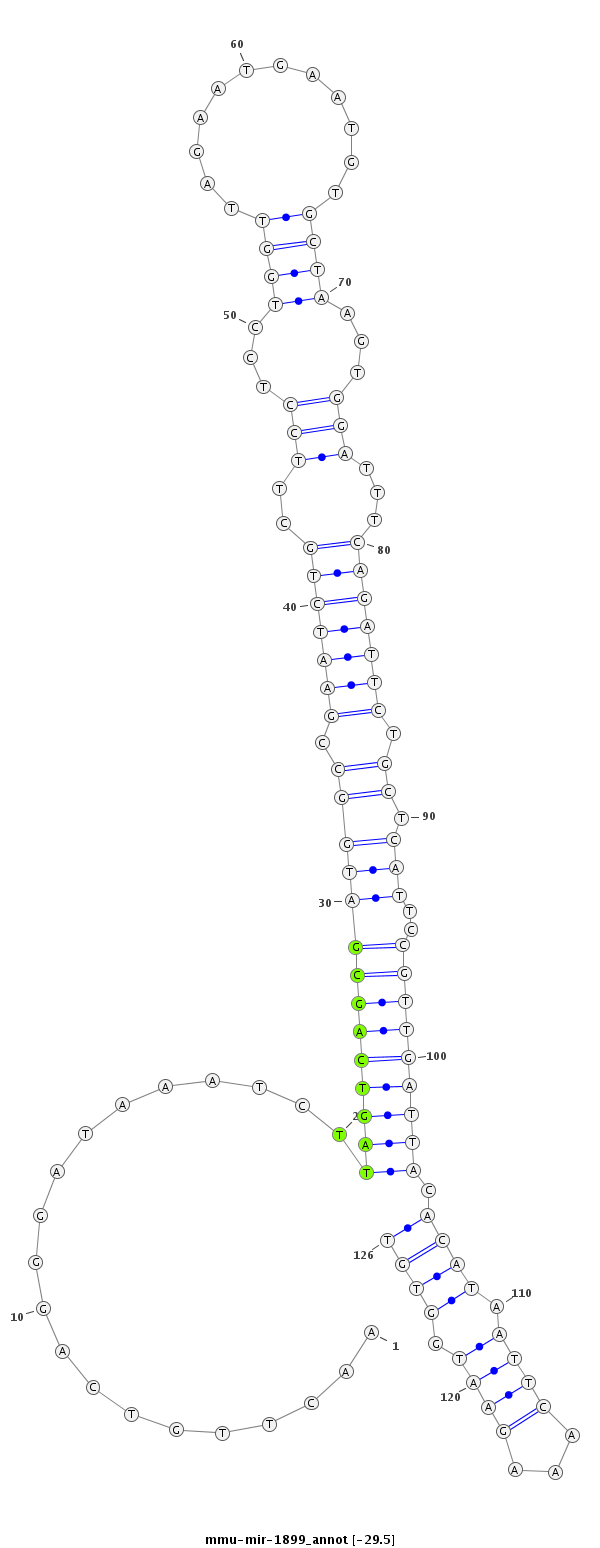 |
|
TGTCTAATTATCAGCTTCCCACTTAGCAGACTGGAAACTTGTCAGGGATAAATCTTAGTCAGCGATGGCCGAATCTGCTTCCTCCTGGTTAGAATGAATGTGCTAAGTGGATTTCAGATTCTGCTCATTCCGTTGATTACACATAATTCAAAGAATGGTGTTTATGCACCATCCCACTCTGAATTGACTACAAGCC
***********************************.(((..((..((((......((((((((((((((.(((((((..(((...((((............))))...)))...))))))).)).)))..)))))))))......))))...))..)))..*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR039185 fibroblast |
SRR037902 testes |
SRR095855BC5 heart |
SRR042466 spleen |
SRR391849 embryo |
SRR525238 Blood |
SRR345200 brain |
SRR345203 brain |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................................TCCCGCTCTGAATTGACT....... | 18 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........TCAGGTTCCCACTTAGCAG....................................................................................................................................................................... | 19 | 1 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................TTAGTCAGCG.................................................................................................................................... | 10 | 0 | 20 | 0.10 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................TCAGCGATGGCCGAGT.......................................................................................................................... | 16 | 1 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................AGCATGCTGGAAACTTGTTAGG...................................................................................................................................................... | 22 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................TCAGCGATGGCCGAGTTT........................................................................................................................ | 18 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................GCGAAGGCCGAATC......................................................................................................................... | 14 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................TATTAGTCAGCGACGGC............................................................................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................CAGCGATGGCCG............................................................................................................................. | 12 | 0 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
|
ACAGATTAATAGTCGAAGGGTGAATCGTCTGACCTTTGAACAGTCCCTATTTAGAATCAGTCGCTACCGGCTTAGACGAAGGAGGACCAATCTTACTTACACGATTCACCTAAAGTCTAAGACGAGTAAGGCAACTAATGTGTATTAAGTTTCTTACCACAAATACGTGGTAGGGTGAGACTTAACTGATGTTCGG
***********************************.(((..((..((((......((((((((((((((.(((((((..(((...((((............))))...)))...))))))).)).)))..)))))))))......))))...))..)))..*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR345201 brain |
SRR345198 brain |
SRR345197 brain |
SRR345199 brain |
SRR065055 jejunum |
SRR345203 brain |
SRR345196 brain |
SRR037924 embryo |
SRR345207 brain |
SRR039153 muscle |
SRR095855BC5 heart |
SRR095855BC8 heart |
SRR345200 brain |
SRR345205 brain |
SRR391846 embryo |
SRR391847 embryo |
SRR553583 cerebellum |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................CAGTCGCTGCCGGCTT........................................................................................................................... | 16 | 1 | 3 | 0.67 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCGCCTAAAGTCTACGACGAG...................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTCACCTAAAGTCTAACACGAT...................................................................... | 22 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................AAGTTTAGGACGAGTACGGCAAC............................................................. | 23 | 3 | 5 | 0.40 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCAGCTAAAGTCTAACACGAT...................................................................... | 21 | 3 | 20 | 0.35 | 7 | 3 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 |
| .........................................................................................................TCAGCTAAAGTCTAAGACGAT...................................................................... | 21 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................AAGTCTAAGACGAGTA.................................................................... | 16 | 0 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCGGCTAAAGTCTACGACGAG...................................................................... | 21 | 3 | 20 | 0.20 | 4 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTCGCCTAAAGTCTAACACGAT...................................................................... | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCACCTAAAGTCTAACACGAT...................................................................... | 21 | 2 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCGGCTAAAGTCTAACACGAG...................................................................... | 21 | 3 | 20 | 0.15 | 3 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTCAGCTAAAGTCTAACACGAT...................................................................... | 22 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCAGCTAAAGTCTACCACGAG...................................................................... | 21 | 3 | 17 | 0.12 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCAGCTAAAGCCTAAGACGAT...................................................................... | 21 | 3 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTCGGCTAAAGTCTAACACGAG...................................................................... | 22 | 3 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTCGGCTAAAGTCTAAGACGAT...................................................................... | 22 | 3 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................AAAGTCTAAGACGAG...................................................................... | 15 | 0 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCCGCTAAAGTCTAACACGAG...................................................................... | 21 | 3 | 19 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................GTCTAAGCCGCCTAAGGCA............................................................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TGAGCTAAAGTCTAACACGAG...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCGGCTAAAGTCTAAAACGAG...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................ACGTGGTAGGGT.................... | 12 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................GTATTAAGTTGCCTACCA..................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .........................................................................................................TTGGCTAAAGTCTAAGACGAG...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCGGCTAAGGTCTAAGACGAG...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTCTCCTAAAGTCTAACACGAT...................................................................... | 22 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................ACCTTTGAGCAGGCCCTA................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................GTATGGCAACTAATG........................................................ | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................TCAGCTAAAGTCTACGACGAA...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCCGCTAAAGTCTAAGACGAT...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TTGCCTAAAGTCTAAGACGAT...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................ACGATTCGGCTAAAGTCTA............................................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TCGCCTAAAGTCCACGACGAG...................................................................... | 21 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................CGCAGGAGGACCAATC........................................................................................................ | 16 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| mm10 | chr9:8246736-8246931 + | mmu-mir-1899 | TGTCTAATTATCAGCTTCCCACT-TAGCAGACTGGAAACTTGTCAGGGATAAATCTTAGTCAGCGATGGCCGAATCTGCTTCCTCCTGGTTAGAATGAATGTGCTAAGTGGATTTCAGATTCTGCTCATTCCGTTGATTACACATAA--TTCAAAGA-A-TGGTGTTTATGCACCATCCCACTCTGAATTGACTACAAGCC |
| cavPor3 | scaffold_19:5880511-5880703 + | TAGAAAATCAGAAGCTTCCCACTTCAGCAGACTGAAAACTCATCAGAG--AAATCTTAGTCAGTTTGAGGAGAATCATCTT---CCTAGCTAAACTGCATGTGTTAAGTGGATTTCATATTCTGTTCATTCACTTGATTATACATAA--CACAAAAA-AGTGGAAGTCATGTACAATCCACATTTAAATCAACTACAAGCA | |
| dipOrd1 | scaffold_12842:12901-13097 - | TAGAAAATGATAAGTGTCCCACTTCAGCACACTGGAGACCCCTCAGAGATACATCTTAATCAGTGCTGGCAGAGTCATCTT---CTTGGCTAAACCACATGTGTCAGTTGGATTTCACATTCTGTTTATTTACTTGATTATATGCAGCACACAAAAA-AGGAGCAGTCGTGCACAATCCAAATTTTAATCAGCTCTAAGTG | |
| hetGal2 | Unknown | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| ochPri2 | scaffold_85501:4457-4647 + | TAGAAAATTATAAGCTTCCCACTTTAGCAAACTGAAAACTCATCAGCA--AAATCTTAG----TGCTGGCAGAATCATCTTCCTCCTGGTTTAACTGCATGTGTTAAGTGGAT-TCATATTCTGTTCATTCCCTTGATTGTACATAA--CACAAAGA-AGTGGCTGTCGCATACAATCCAAATTTGAATAAACTAGAAGCA | |
| oryCun2 | chr1:114982400-114982593 - | TAGAAAATTACAAGCTTCCCACTTTAGCAAACTGAAAACTCATCAGAGAGAAATCTTAG----TGCTGGCAGAATCATCTTCCTCCTGGTTAAACTGCATGCGTTAAGTAGATTTCATATTCTGTTCATTCACTTGATTGTACATAA--CACAAAAA-CGTGGCAGTCATGTACAATCCAAATTTGAATCGACTAGAAGCA | |
| rn5 | chr8:6460943-6461139 - | TGTCTAATTACCAGCTTCCCACTTTAGCAGACTGGAAACTCATCAGAGATAAATCTTAGTCAGTGCTGGCAAAATCTGCTTCCTCCTGGTTAGACTGAATGTGCTAAGTGGATTTCAGATTCTGCTCATTCCGTTGATTACGCATAA--TACAAAGA-A-TGGTGCTCATGCAGCATCCCACTCTGAACTGACTACAAGCC | |
| speTri2 | JH393361:6005999-6006193 - | AAGAAAATTATAAGCTTCCCACTTTAGCAGACTGAAAACTCATCAGAGATAAATCTTAG----TGCTGGCAGAATCATCTTCCTCCTGGTTAAACTGCATGTGTTAAGTGGATTTCATATTCTGTTCATTCACTTGATTATACATAA--CACAAAAAAAGTGGCAGTCATGTACTATCCAAATTTGAATCGACTATTAGCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mm10 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| cavPor3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dipOrd1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| hetGal2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ochPri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| oryCun2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| rn5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| speTri2 |
|
Generated: 05/01/2015 at 01:38 AM