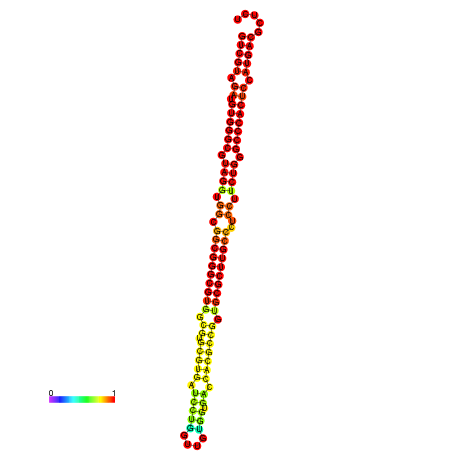
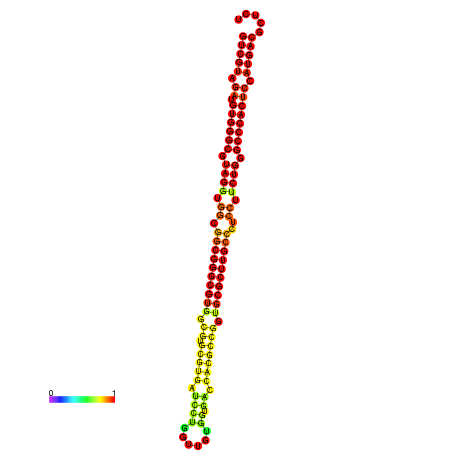
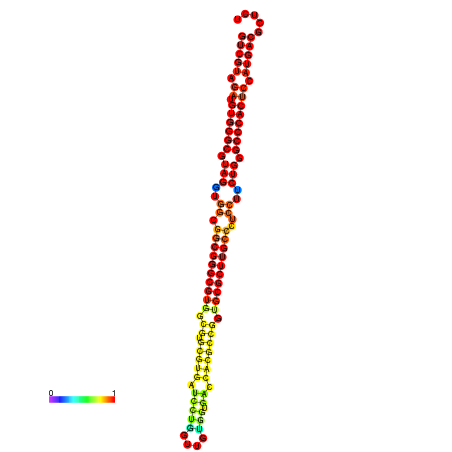
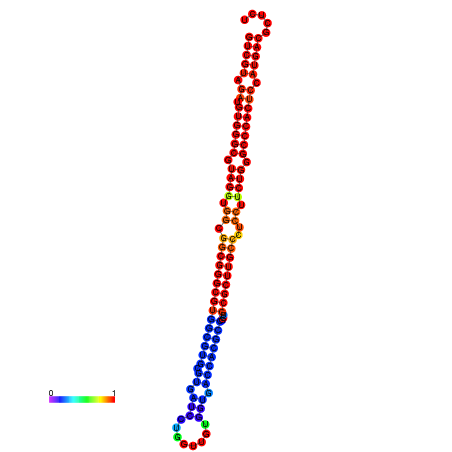
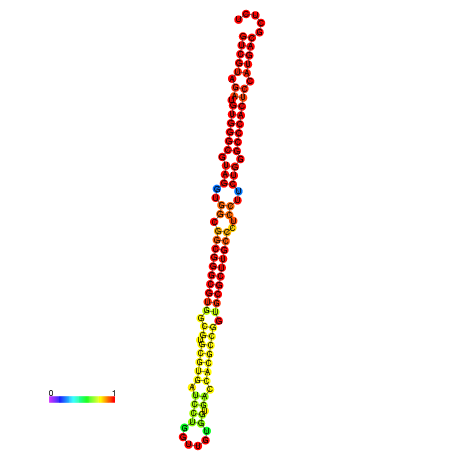
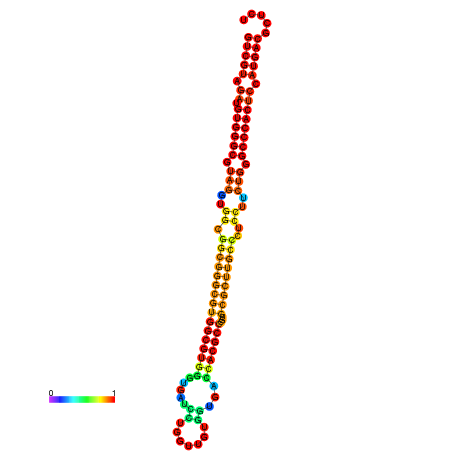
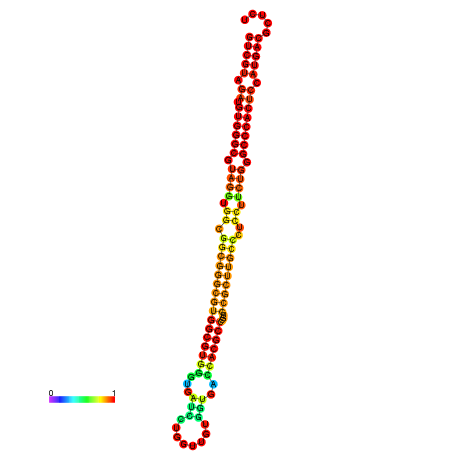
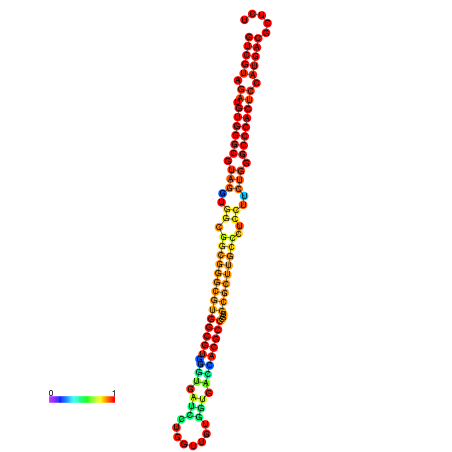
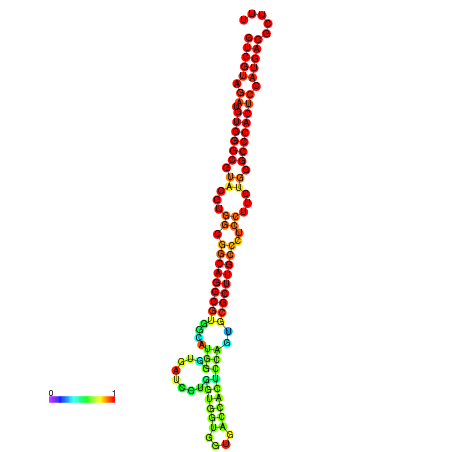
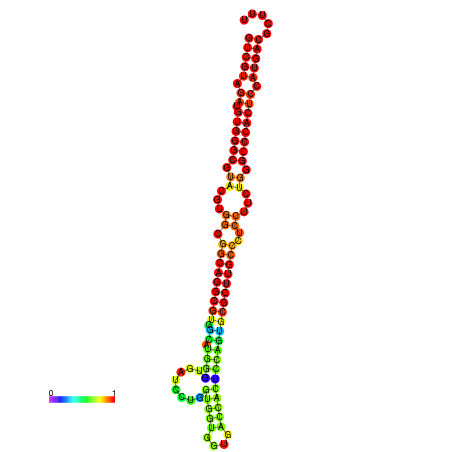
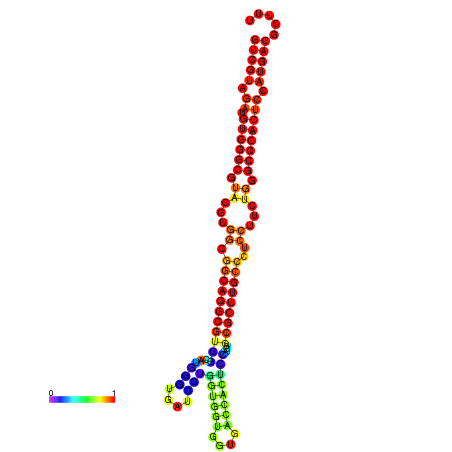
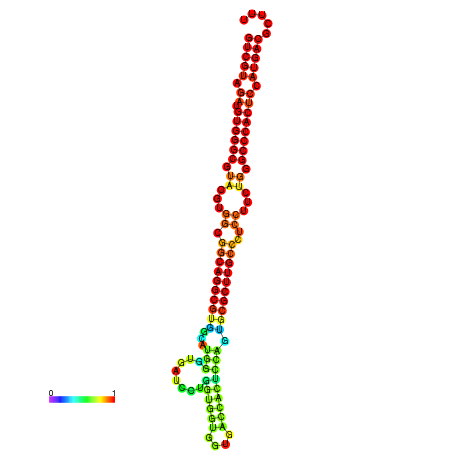
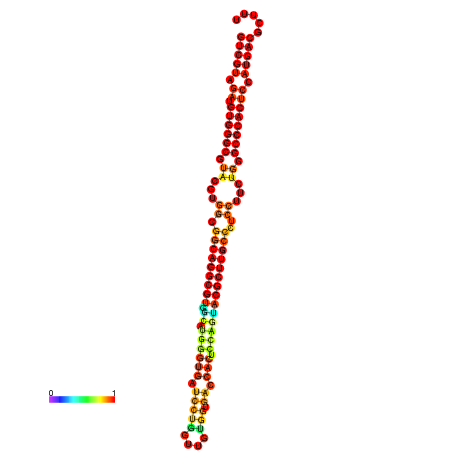
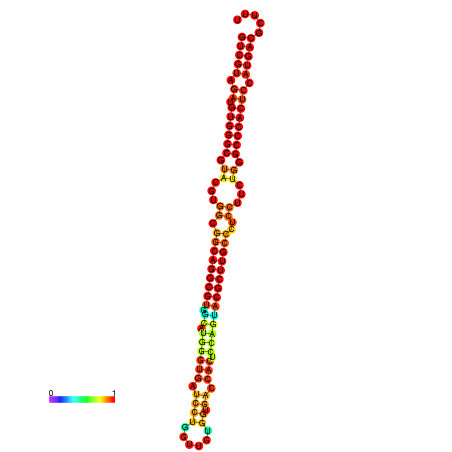
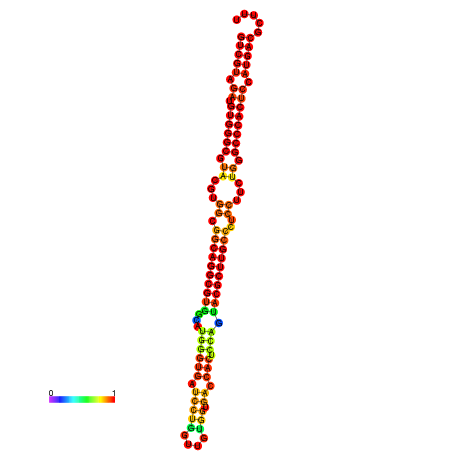
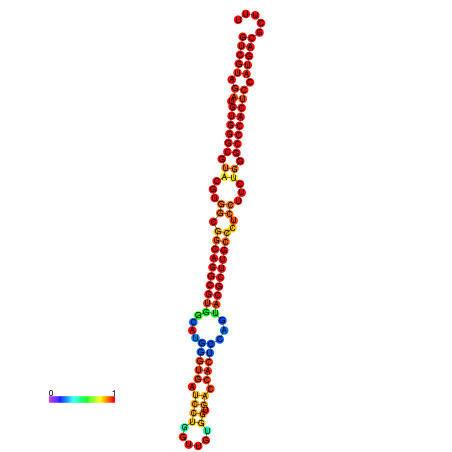
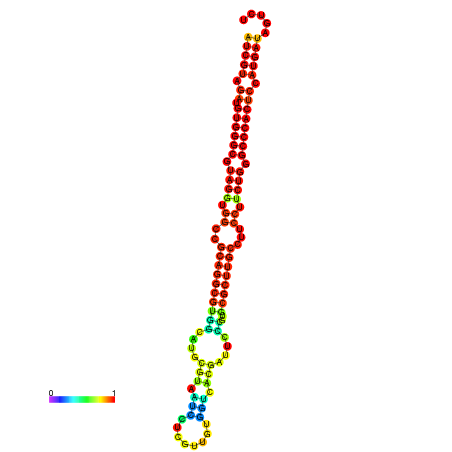
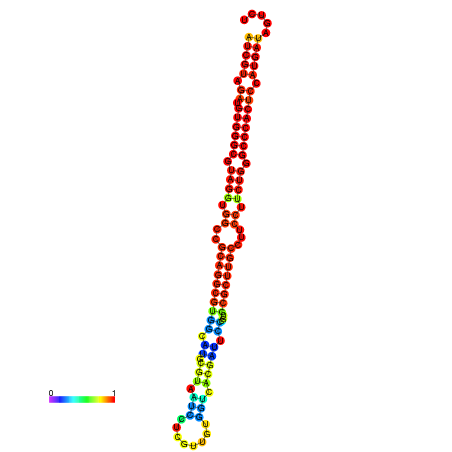
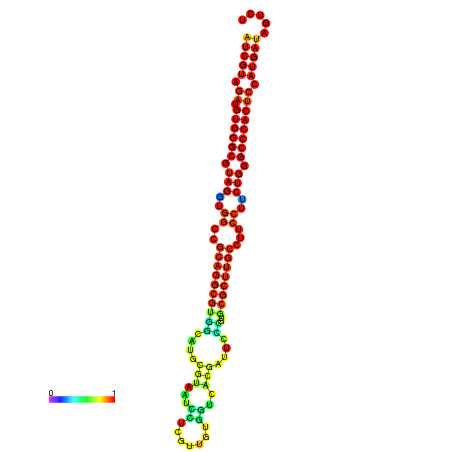
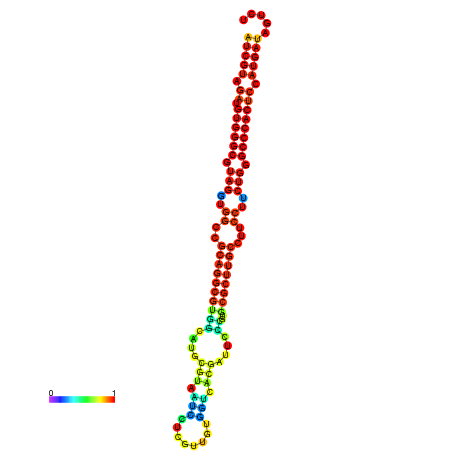
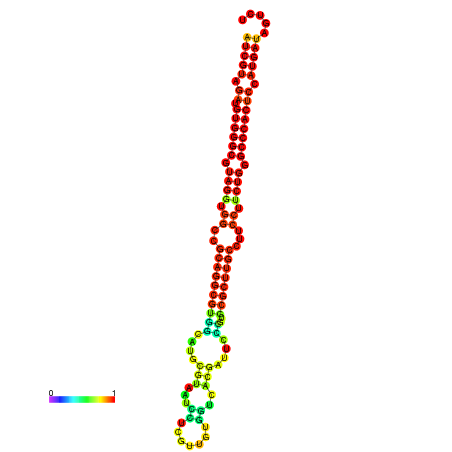
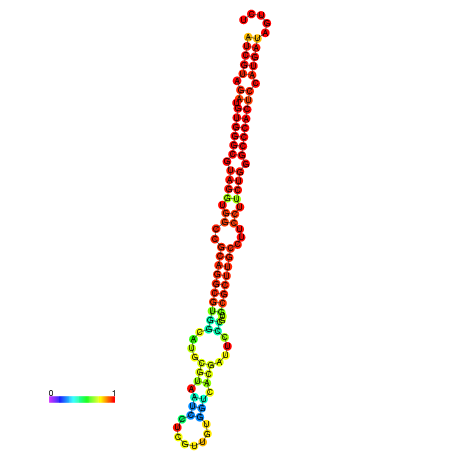
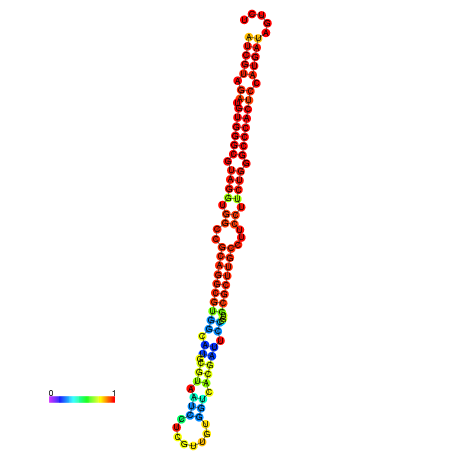
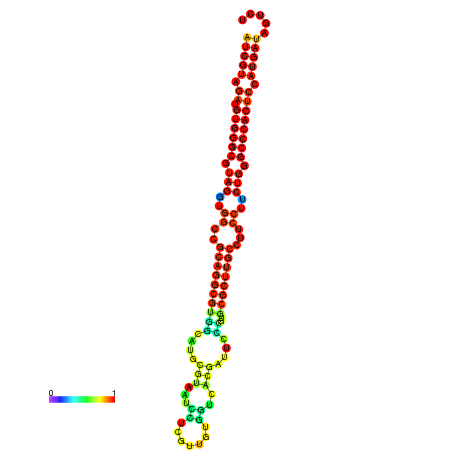
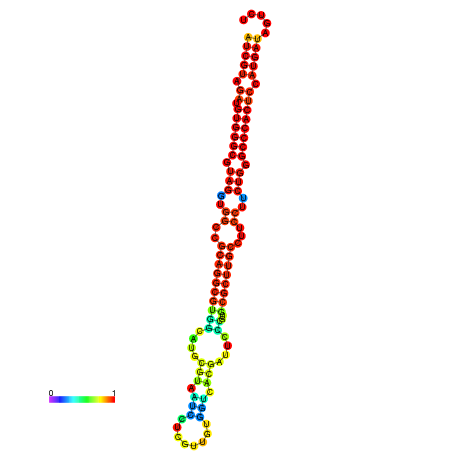
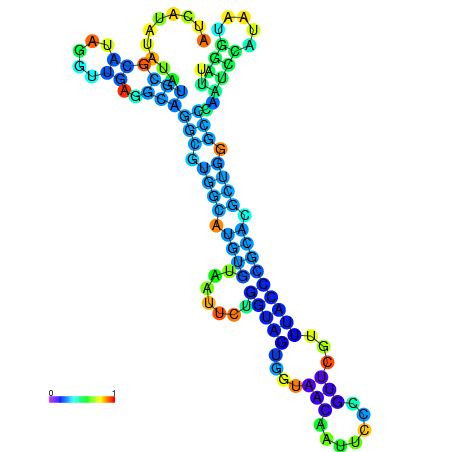
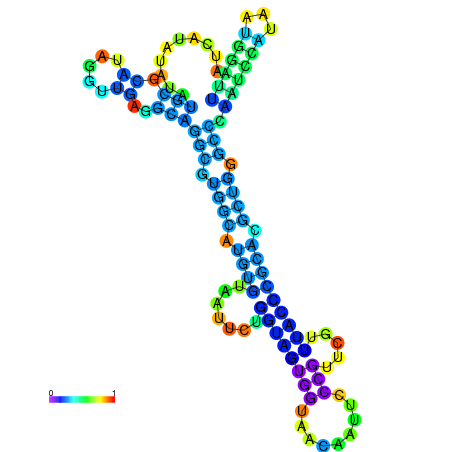
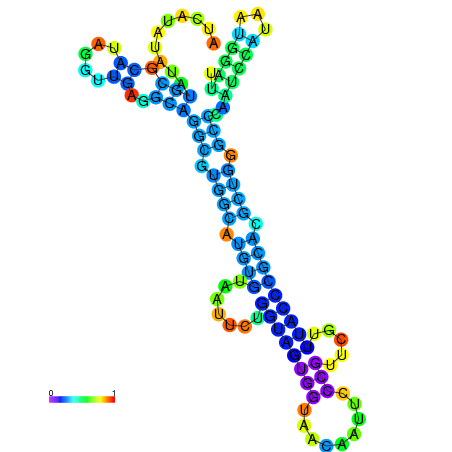
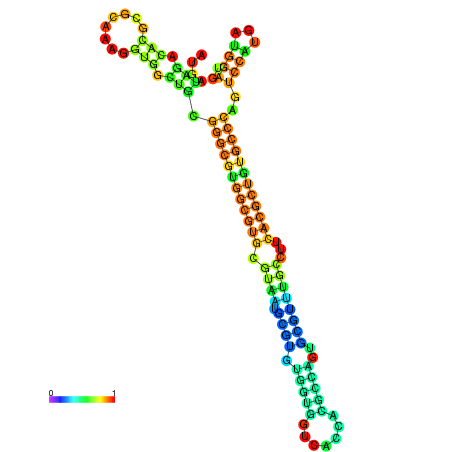
| dm3 |
chr3R:26876804-26876940 - |
GCACTCCCAGTCCCGGTCGTAGATGTGGGCGTAGGTGGCGGCGGGCGTGGCGTGCGTGATCCTGGTTGTGGTGACCACGCCGGTGCGCTTGCCCTCCTTCTGGGCCCACTCCATGACGCTCTGGACCCTGCCTTGCT---------------- |
| droSim1 |
chr3R:26523820-26523956 - |
GCACTCCCAGTCGCGGTCGTAGATGTGGGCGTAGGTGGCGGCGGGCGTGGCGTGGGTGATCCTGGTTGTGGTGACCACGCCCGTGCGCTTGCCCTCCTTCTGGGCCCACTCCATGACGCTCTGGACCCTGCCTTGTT---------------- |
| droSec1 |
super_4:5701489-5701625 - |
GCACTCCCAGTCGCGGTCGTAGATGTGGGCGTAGGTGGCGGCGGGCGTGGCGTGTGTGATCCTGGTTGTGGTGACCACGCCGGTGCGCTTGCCCTCCTTCTGGGCCCACTCCATGACGCTCTGGACCCTGCCTTGCT---------------- |
| droYak2 |
chr3R:27815996-27816132 - |
GCACTCCCAATCCCGGTCGTAGATGTGGGCGTACGTGGCGGCAGGCGTGGCATGGGTGATCCTGGTGGTGGTGACCACTCCAGTGCGCTTGCCCTCCTTCTGGGCCCACTCCATGACGCTTTGGACCCTACCTTGCT---------------- |
| droEre2 |
scaffold_4820:1105506-1105642 + |
GCACTCCCAATCCCGGTCGTAGATGTGGGCGTACGTGGCGGCAGGCGTGGCATGGGTGATCCTGGTTGTGGTGACCACTCCAGTACGCTTGCCCTCCTTCTGGGCCCACTCCATGACGCTTTGGACCCTGCCCTGCT---------------- |
| droAna3 |
scaffold_12911:4046748-4046884 + |
GCACTCCCAATCCCGATCGTAGATGTGGGCGTAGGTTGCAGCTGGCGTGGCGTGGGTAATCCGAGTGGTGGTCACTATTCCAGTTCGCTTCTTCTCCTTTTGGGCCCACTCCATGACGCTCTGAACGCGTCCTTGCT---------------- |
| dp4 |
chr2:271882-272018 + |
GCACTCCCAGTCGCGATCGTAGATGTGGGCGTAGGTGGCCGCAGGCGTGGCATGCGTAATCCTCGTTGTGGTCACGATTCCCGTGCGCTTGCCTTCCTTCTGGGCCCACTCCATGATAGTCTCAATCCGGCCCAGCT---------------- |
| droPer1 |
super_7:3056347-3056483 + |
GCACTCCCAGTCGCGATCGTAGATGTGGGCGTAGGTGGCCGCAGGCGTGGCATGCGTAATCCTCGTTGTGGTCACGATTCCCGTGCGCTTGCCTTCCTTCTGGGCCCACTCCATGATAGTCTCAATCCGGCCCAGCT---------------- |
| droWil1 |
scaffold_181108:531799-531935 - |
GCATTCCCAGTCCCGATCATATATATGCGCATAGGTTGAGGCAGGCGTGGCATGTGTAATTCTGGTAGTGGTAACAATTCCCGTTCGTTTACCCGCACGCTGGGCCCAATCCATAATGGATTCCACCCGACCCTGTT---------------- |
| droVir3 |
scaffold_12855:1470098-1470234 + |
GCACTCCCAGTCCCGATGATAGACACGCGCAAAGGTGGCTGCGGGCGTGGCGTGCGTAATGCGTGTGGTGGTCACCACGCCAGTGCGTTTGCCTTCACGCTGTGCCCAGTCCATGATGGAGTCGAGTCGACCCTGGC---------------- |
| droMoj3 |
scaffold_6540:27213622-27213758 + |
GCACTCCCAGTCCCGATGATAGATCCTTGCATAGGTGGCCGCAGGTGTGGCGTGTGTTATACGCGTGGTAGTCACGACGCCGGTCCGCTTGCCCTCACGCTGTGCCCAGTCCATGATGGAGTCGAGTCGACCCTGGC---------------- |
| droGri2 |
scaffold_15203:6653367-6653519 - |
GCACTCCCACCGCCGATCGGCGACATGGGCGTAGAGGGCAGCGGGTGTGGCATGGGTTACCCGCGTTGTGGTCACAAATCCAGTGCGCATGCCATCCACCTGCGCCCAGTTGAATATGTTCTGGACGTGATGTTCCTCCTTGAGAGAGGCACT |