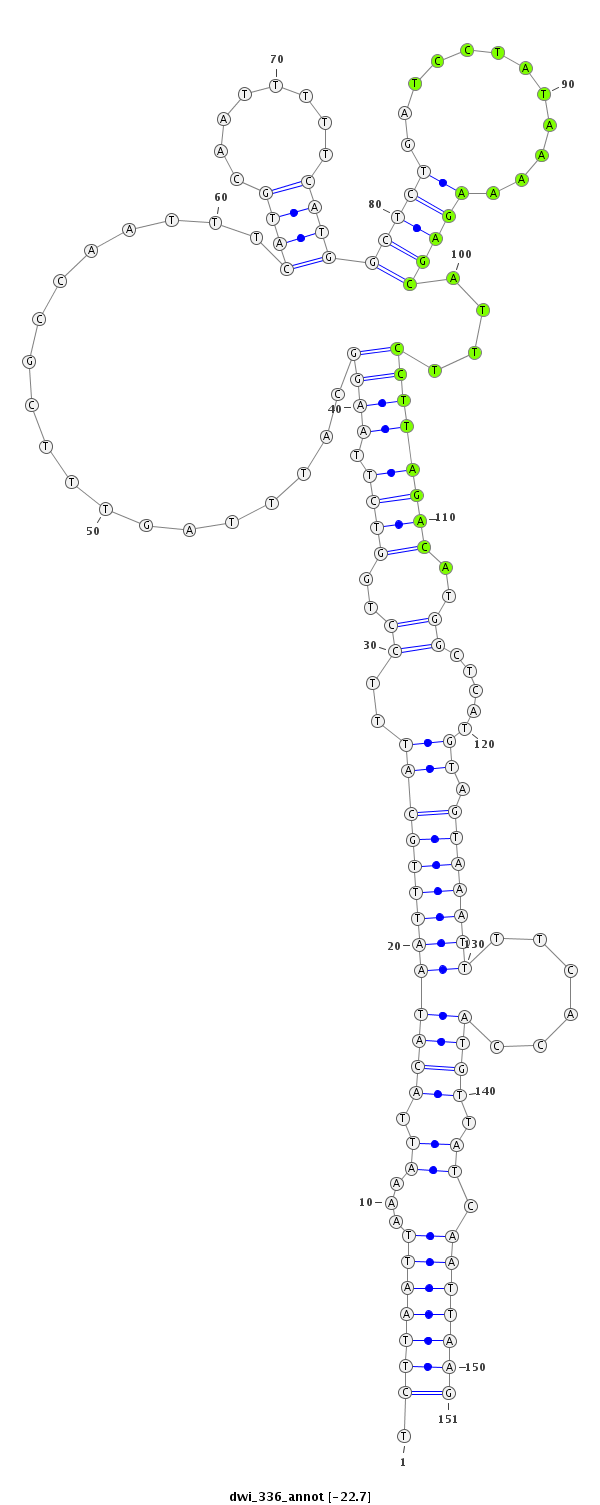
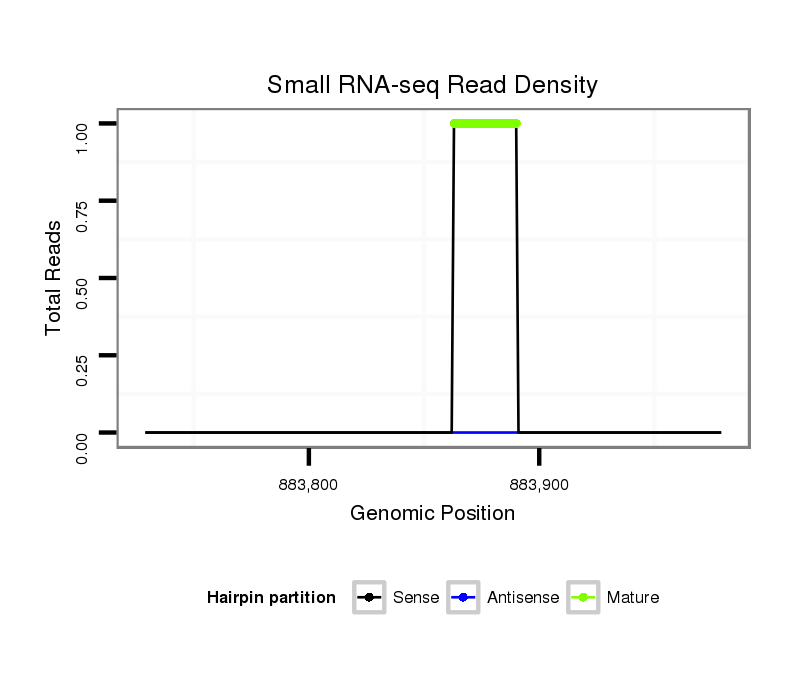
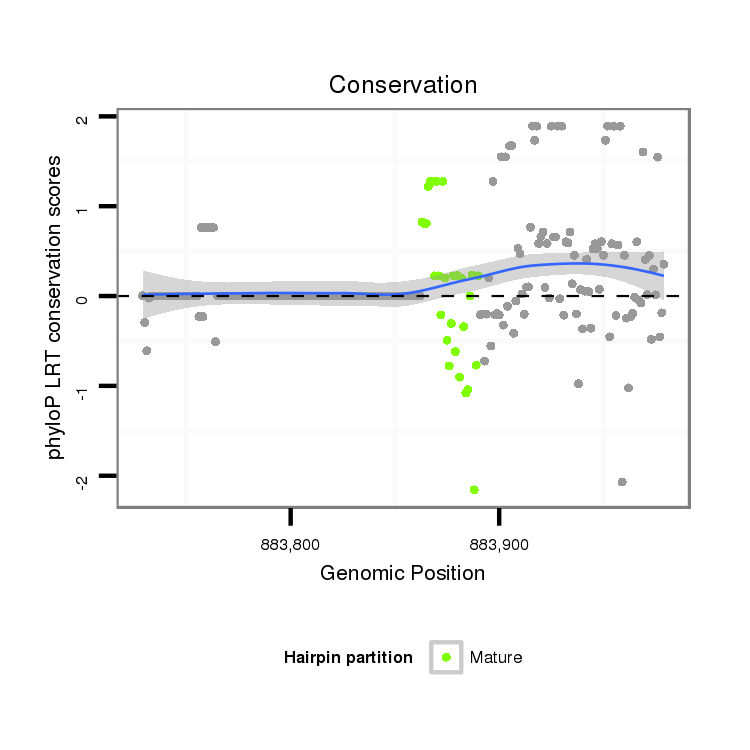
ID:dwi_336 |
Coordinate:scf2_1100000004382:883779-883929 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -22.7 | -22.5 | -22.5 |
 |
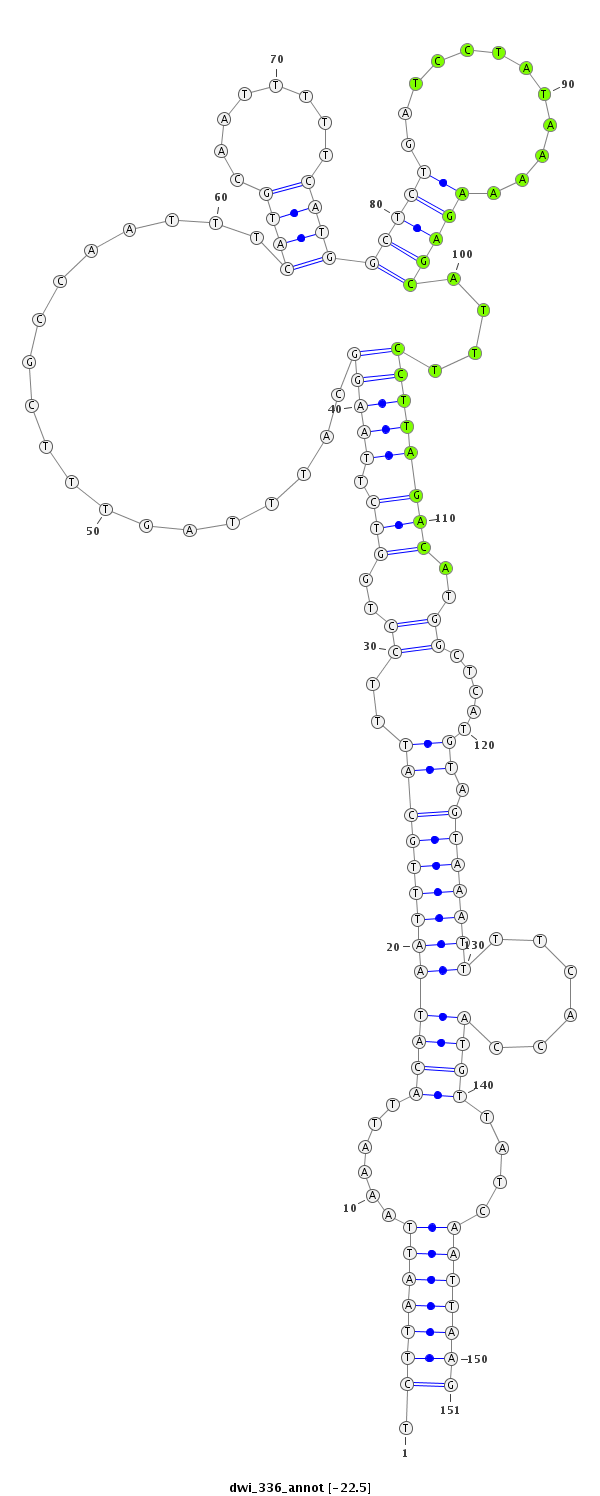 |
 |
CDS [Dwil\GK10737-cds]; exon [dwil_GLEANR_10938:2]; intron [Dwil\GK10737-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CATATACTAAATAAAGTATCCGCCCTGTTAACAAAGATAAGAAATATAAATCTTAATTAAAATTACATAATTTGCATTTCCTGGTCTTAAGGCATTTAGTTTCGCCAATTTCATGCAATTTTTCATGGCTCTGATCCTATAAAAAGAGCATTTCCTTAGACATGGCTCATGTAGTAAATTTTCACCATGTTATCAATTAAGAGTCTTGTTCCTGTTCTTGGCCTGCTACTTGTCCTGTCTGGCCATGGCTT **************************************************.(((((((...((.(((((((((((((..((..((((.((((...................((((........))))(((((............)))))....))))))))..)).....)).)))))))......)))).)).)))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V119 head |
M045 female body |
V118 embryo |
M020 head |
V117 male body |
|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................TATTGCATTTAGTTTCGCCA................................................................................................................................................ | 20 | 2 | 6 | 2.33 | 14 | 7 | 3 | 3 | 1 | 0 |
| ......................................................................................................................................TCCTATAAAAAGAGCATTTCCTTAGACA......................................................................................... | 28 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................TTCATGGCTCTCATCCTATAAAAAGAG....................................................................................................... | 27 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................CTACTTGTCCTGTCTGGCC....... | 19 | 0 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 |
| .........................................................................................ATGCATTTAGTTTCGCCAA............................................................................................................................................... | 19 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................TAAGAGTCTTGTTCCTGTTCT................................. | 21 | 0 | 4 | 0.50 | 2 | 0 | 0 | 0 | 0 | 2 |
| ..........................................................................................TGCATTTAGTTTCGCCCA............................................................................................................................................... | 18 | 2 | 20 | 0.45 | 9 | 4 | 2 | 3 | 0 | 0 |
| ..........................................................................................TGCATTTAGTTTCGCCAA............................................................................................................................................... | 18 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................................................................................................GTTCCTGTTCTTGGCCTG.......................... | 18 | 0 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................................................................................................................................................TAAGAGTCTTGTTCCTGTTCTTGGCC............................ | 26 | 0 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................................................................................................................AGAGTCTTGTTCCTGTTCTTG............................... | 21 | 0 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................CACTTTGCATTTCCTGGTCATA.................................................................................................................................................................. | 22 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................TGTTGGCCTGCTAATTGT.................. | 18 | 2 | 16 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................ACTGCATTTAGTTTCGCCCA............................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 |
|
GTATATGATTTATTTCATAGGCGGGACAATTGTTTCTATTCTTTATATTTAGAATTAATTTTAATGTATTAAACGTAAAGGACCAGAATTCCGTAAATCAAAGCGGTTAAAGTACGTTAAAAAGTACCGAGACTAGGATATTTTTCTCGTAAAGGAATCTGTACCGAGTACATCATTTAAAAGTGGTACAATAGTTAATTCTCAGAACAAGGACAAGAACCGGACGATGAACAGGACAGACCGGTACCGAA
**************************************************.(((((((...((.(((((((((((((..((..((((.((((...................((((........))))(((((............)))))....))))))))..)).....)).)))))))......)))).)).)))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M020 head |
V119 head |
V117 male body |
V118 embryo |
M045 female body |
|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................................................................................................CGGACGATGAACAGGACAGAC.......... | 21 | 0 | 3 | 0.67 | 2 | 1 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................................................AAGGACAAGAACCGGACGATG...................... | 21 | 0 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................CGGACGATGAACAGGACAG............ | 19 | 0 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................GGACAAGAACCGGACGATGA..................... | 20 | 0 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................CAGAATTCCGTAAATCAAA..................................................................................................................................................... | 19 | 0 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................CCGGACGATGAACAGGACAGAC.......... | 22 | 0 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................TTCTCAGAACAAGGACAAGA................................. | 20 | 0 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................................CTGGTAAAGGAAACTGTA........................................................................................ | 18 | 2 | 14 | 0.21 | 3 | 0 | 2 | 0 | 0 | 1 |
| .......................................................................................................................................................................................................TCTCAGAACAAGGACAAGAAC............................... | 21 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................ACTGGGCGACGAACAGGAC.............. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................................................................................................................................AGGGCAAGAACCGTACGAC....................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droWil2 | scf2_1100000004382:883729-883979 + | dwi_336 | CATATACTAAATAAAGTATCCGCCCTGTTAACAAAGATAAGAAATATAAATCTTAATTAAAATTACATAATTTGCATTTCCTGGTCTTAAGGCATTTAGTTTCGCCAATTTCATGCAATTTTTCATGGCTCTGATCCTATAAAAAGAGCATTTCCTTAGACATGGCTCATGTAGTAAATTTTCACCATGTTATCAATTAAGAGTCTTGTTCCTGTTCTTGGCCTGCTACTTGTCCTG---TCTGGCCATGGCTT-------------- |
| droVir3 | scaffold_12875:2778187-2778269 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTCAAGTTCTGAAATGTCTTCGATCTACAAGCTACCGACTGCGCTGGTGCTGTTTCTGCTCACCATCGTTGGCCCTGGTGT-------------- | |
| droAna3 | scaffold_13266:14031423-14031485 - | A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCATGTCTTCGGTTAACAGTCTAGTGTTTCCCCTGATCCTGCTCCTTGCCTCGGCTTCGGTT---------------------- | |
| droBip1 | scf7180000396759:2107788-2107864 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACCATGTCTTTGGTTAACAGTCTAGTGTTTCCTCTGATCCTGCTCGTTGCATCGACTTCGGTTTTTGGCCAGAGTA--------A | |
| droKik1 | scf7180000302470:960422-960526 + | CC---------------------------------------------------------------------------------------------------------------------------------------TATAAATAGGCAACACCCCC-ACCACAGTCAACACACTCAACCTTTGTCATGTCTCAGCTTAAGACTACTGCAACACCTCTGATCCTCCTCCTGGCTGTGGCTT--------------------------- | |
| droFic1 | scf7180000453851:762836-762913 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACCCTTGCCATGTCTTCGGTTAACAATCTAGCCAGTCCTCTGGTCCTCCTGCTCGTCGCCACCTCGGTCCTCGGCGA-------------- | |
| droEug1 | scf7180000409672:2603247-2603378 - | CC---------------------------------------------------------------------------------------------------------------------------------------TATAAATAGAGAATAACAGA-ACGACATTCAACACACTCAACCTTTGCCATGTCTTCGGTTAACAATCTAGTAAAACTTTCGATCCTACTCCTCGTGGTGACTTTTGTTCACTGTGAAATGGATATGGCCG | |
| dm3 | chr2R:12559502-12559629 + | CCAG----------------------------------------------------------------------------------------------------------------------------------TCCTATAAATAGAGAACAACTCC-ACCACAATCGACACACTCAACCTTTGCCATGTCTTCGGTTAACAATCTAGTGAAACTATCGATCCTGCTCCTGGCTGTGACTTTTGTTCACAGCGATATGG--------- | |
| droSim2 | 2r:13231903-13232030 + | CCAG----------------------------------------------------------------------------------------------------------------------------------TCCTATAAATAGAGAACAACTGC-AGCACAATCGACACACTCAACCTTTGCCATGTCTTCGGTTAACAATCTAGTGAAACTATCGATCCTGCTCCTAGCTGTGACTTTTGTTCACAGTGATATGG--------- | |
| droSec2 | scaffold_1:10057492-10057617 + | CCAG----------------------------------------------------------------------------------------------------------------------------------TCCTATAAATAGAGAACAACTGC-AGCACAATCGACACACTCAACCTTTGCCATGTCTTCGGTTAACAATCTAGTGAAACTATCGATCCTGCTCCTAGCTGTGACTTTTGTTCACAGTGATAT----------- | |
| droYak3 | v2_chrUn_258:43749-43809 - | CTTG-----------------------ATTACAAAC------------------------------------------------------------------------------------------------------ATGAGAAAGATGTATTTGT-AATGAGGCCAAATAATTTAAATTTCCCAG--------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/17/2015 at 10:47 PM