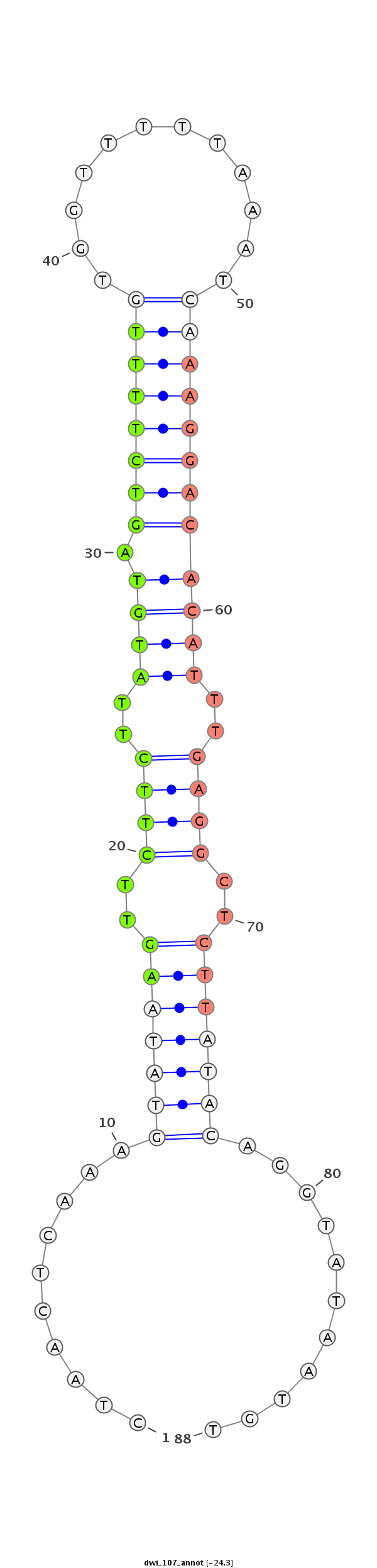
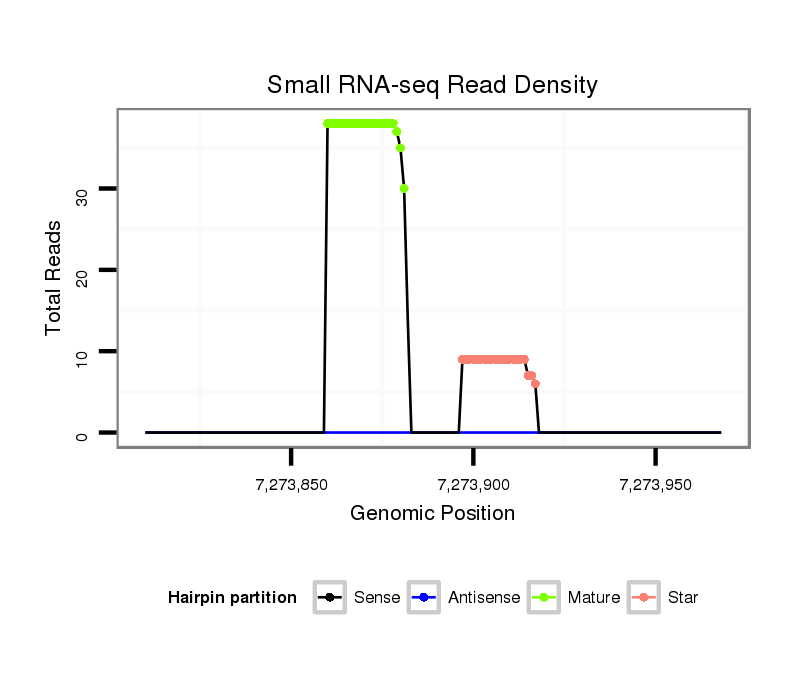
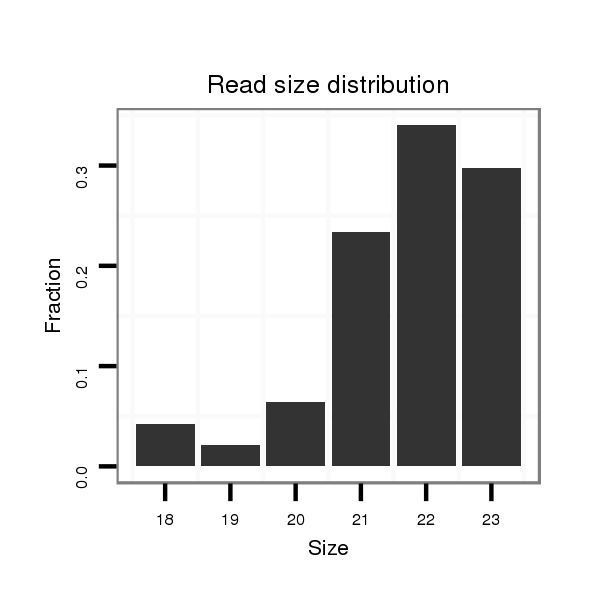
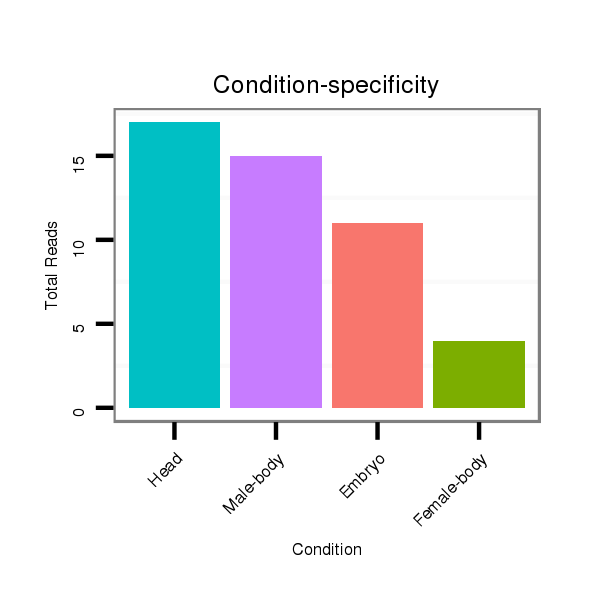
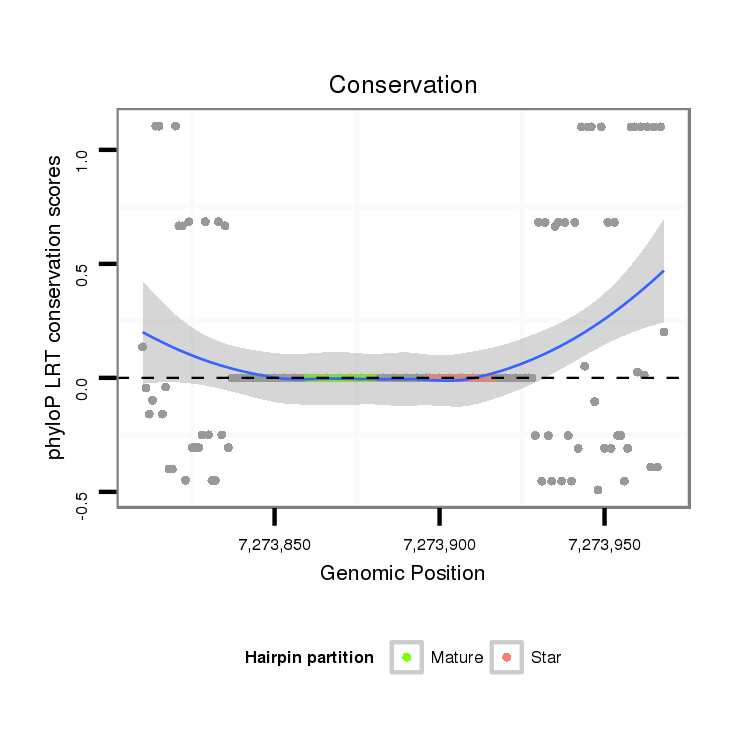
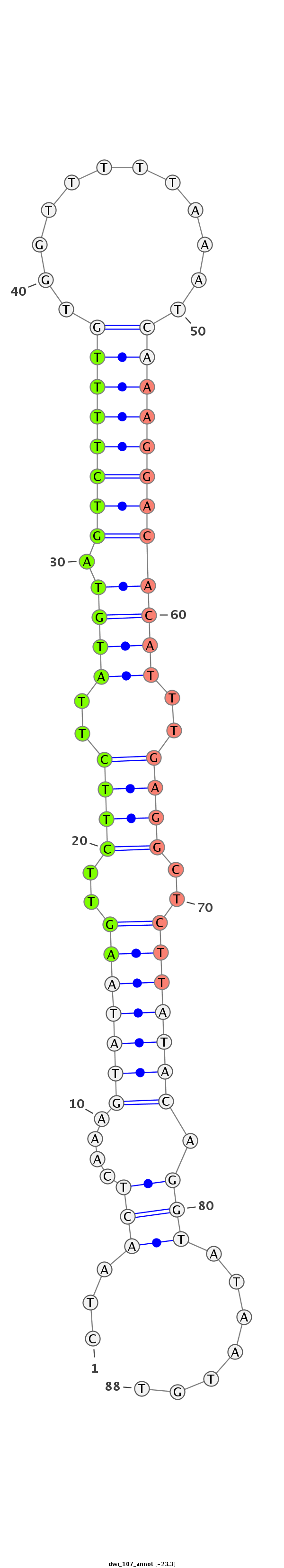
ID:dwi_107 |
Coordinate:scf2_1100000004902:7273860-7273918 + |
Confidence:confident |
Class:Testes-restricted |
Genomic Locale:intergenic |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -23.3 |
 |
intergenic
No Repeatable elements found
|
CTAATTACAATGGATTAAATCTTTCGAAATAGTTACTAACTCAAAGTATAAGTTCTTCTTATGTAGTCTTTTGTGGTTTTTAAATCAAAGGACACATTTGAGGCTCTTATACAGGTATAATGTTTCTGTTCTATGTTTTAATTACAATTAATTACAGAT
***********************************..........(((((((..((((..((((.((((((((............))))))))))))..))))..)))))))...........************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | M020 head |
V117 male body |
V118 embryo |
M045 female body |
V119 head |
|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................AGTTCTTCTTATGTAGTCTTTT....................................................................................... | 22 | 0 | 1 | 16.00 | 16 | 2 | 9 | 3 | 2 | 0 |
| ..................................................AGTTCTTCTTATGTAGTCTTTTG...................................................................................... | 23 | 0 | 1 | 14.00 | 14 | 1 | 2 | 8 | 0 | 3 |
| .......................................................................................AAGGACACATTTGAGGCTCTT................................................... | 21 | 0 | 1 | 6.00 | 6 | 5 | 1 | 0 | 0 | 0 |
| ..................................................AGTTCTTCTTATGTAGTCTTT........................................................................................ | 21 | 0 | 1 | 5.00 | 5 | 1 | 2 | 0 | 2 | 0 |
| .......................................................................................AAGGACACATTTGAGGCT...................................................... | 18 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ..................................................AGTTCTTCTTATGTAGTCTT......................................................................................... | 20 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 |
| .......................................................................................AAGGACACATTTGAGGCCCTT................................................... | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................GAGGACACATTTGAGGCTCT.................................................... | 20 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................AAGGACACATTTAAGGCT...................................................... | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................TTAAGGACACATTTGAGGCTCTT................................................... | 23 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..................................................CGTTCTTCTTATGTAGTCTTTT....................................................................................... | 22 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................AGTTCTTCTTATGTAGTCTTTCC...................................................................................... | 23 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................AAGGACACATTTGAGGCTCT.................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..................................................AGTTCTTCTTATGTAGTCT.......................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................ATTAGAATTATTTACAGAT | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
|
GATTAATGTTACCTAATTTAGAAAGCTTTATCAATGATTGAGTTTCATATTCAAGAAGAATACATCAGAAAACACCAAAAATTTAGTTTCCTGTGTAAACTCCGAGAATATGTCCATATTACAAAGACAAGATACAAAATTAATGTTAATTAATGTCTA
************************************..........(((((((..((((..((((.((((((((............))))))))))))..))))..)))))))...........*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total |
|---|
| Species | Coordinate | ID | Type | Alignment |
|---|---|---|---|---|
| droWil2 | scf2_1100000004902:7273810-7273968 + | dwi_107 | confident | CT--AATTACAATGGATTAAATCTTTCGAAATAGTTACTAACTCAAAGTATAAGTTCTTCTTATGTAGTCTTTTGTGGTTTTTAAATCAAAGGACACATTTGAGGCTCTTATACAGGTATAATGTTTCTGTTCTATGTTTTAATTACAATT-AATTACAGAT |
| droVir3 | scaffold_13047:3233409-3233436 + | TA--TGTTTCCCTGGCTATTGTTGGTTGT------------------------------------------------------------------------------------------------------------------------------------T | ||
| droGri2 | scaffold_14853:5075805-5075846 - | TA-----------------------------------------------------------------------------------------------------------------------GTTTCGCTTTCATTTGTTCCATTAATGCAT-AATTAAATAT | ||
| dm3 | chr3R:10784360-10784391 + | TTTTAATTAAAAT--------------------------------------------------------------------------------------------------------------------------TATTTAA--------TAAGTAAAATAC |
| Species | Read alignment | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droWil2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 10/20/2015 at 06:56 PM