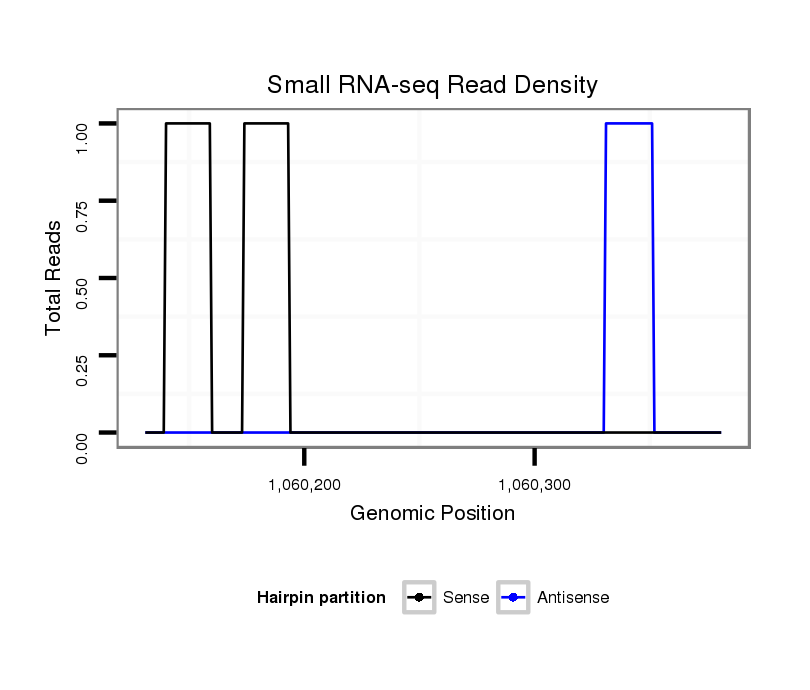
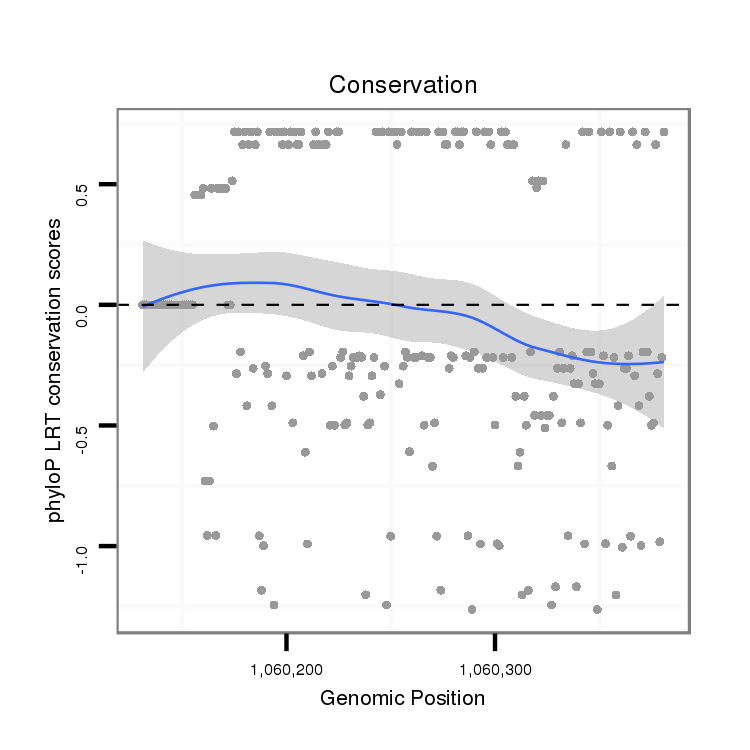
ID:dvi_9984 |
Coordinate:scaffold_12963:1060181-1060331 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ14623-cds]; exon [dvir_GLEANR_1468:5]; intron [Dvir\GJ14623-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CACAATCCTCATGCAGCCAAGGCACGGCGTTCATTCTATTTGCATAACGAAGATGAAAGTAATGTAACTCCAGAGCAACTGCGTCGTCGTAAAATTCTGCATTATGTGTACCTAGTTCTTAAGCTAGCGACATTTATTGGATTTTGGTTGTTCTTTACCGTATTTATGTTACTCACGCCACCGAATGAACATATCAGTAAGCTGATACACGTTGAGTCCCAAAAACCGTATATTATGGTGGTCAATGAGGA **************************************************.......(((((((((((...((((((((((((....)))(((((((.....((.((((..((((((....))))))..)))).))..)))))))))))))))).....))...)))))))))............................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060659 Argentina_testes_total |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
SRR060668 160x9_males_carcasses_total |
V047 embryo |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................ATAACGAAGATGAAAGTAAT............................................................................................................................................................................................ | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........CATGCAGCCAAGGCACGGCG.............................................................................................................................................................................................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................ATGTAACTCCAGAGCAAT............................................................................................................................................................................ | 18 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGCTGATACAAGATGAGT.................................. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................GGTCTTAAGGTAGCGAAATT..................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................GTCCTAAGCTAGCGATATT..................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GTGTTAGGAGTACGTCGGTTCCGTGCCGCAAGTAAGATAAACGTATTGCTTCTACTTTCATTACATTGAGGTCTCGTTGACGCAGCAGCATTTTAAGACGTAATACACATGGATCAAGAATTCGATCGCTGTAAATAACCTAAAACCAACAAGAAATGGCATAAATACAATGAGTGCGGTGGCTTACTTGTATAGTCATTCGACTATGTGCAACTCAGGGTTTTTGGCATATAATACCACCAGTTACTCCT
**************************************************.......(((((((((((...((((((((((((....)))(((((((.....((.((((..((((((....))))))..)))).))..)))))))))))))))).....))...)))))))))............................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060681 Argx9_testes_total |
V116 male body |
SRR060684 140x9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................................................................CGACTATGTGCAACTCAGGGT.............................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................CGTAAGACCCAGGGATCAA...................................................................................................................................... | 19 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................TCAAGCATTCGATGGCAGT....................................................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................GCATTTTAAGACGAAGTATA................................................................................................................................................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| ................................................................................................................................................ACCAACTAGAACTGGCAAA........................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:1060131-1060381 + | dvi_9984 | CACAATCCTCATGCAGCCAAGGCACGGCGTTCATTCTATTTGCATAACGAAGATGAAAGTAATGTAACTCCAGAGCAACTGCGTCGTCGTAAAATTCTGCATTATGTGTACCTAGTTCTTAAGCTAGCGACATTTATTGGATTTTGGTTGTTCTTTACCGTATTTATGTTACTCACGCCACCGAATGAACATATCAGTAAGCTGATACACGT---------TGAGTCCCAAAAACCGTATATTATGGTGGTCAATGAGGA |
| droMoj3 | scaffold_6500:27791387-27791609 + | -------------------------GGCGTGGCTCGTATTT---TGACGATGAAGAGCTCGAATTAACTCCAGAGCAAGAGCGTCGCCGTCGCATTCGGCGTTATATAGCCTTAATCATAAAGATGGTCATATTTCTTATCATGTGGATATTCTTTGTGGTTAATATGTGTATTACGTCAAGAACGG---------TTGTGCAGGAAAGAGTTGTGGCCTTAGAGCAGAAATCAATAAAGTATTTGGATGTCACGGGAAA | |
| droGri2 | scaffold_15126:2385731-2385938 - | -------------------------------------------ATGATGATGATGATGATGAACTAACTACACAGCAGCGAAGTCGCCGTAAAATATTCAAAATCGGCTCACTAATTGTCAAGCTAACGACTTTCATAGTCCTCTGGTCGTTCTTCTCAATAGTTTTGATCTTCACGCCCACCCACAAGCACAATGCGCAAGTGTTGCCCCT---------GAAACCACAGCAAACCAAAATCGTTGAAATTCAGGGTGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/17/2015 at 05:08 AM