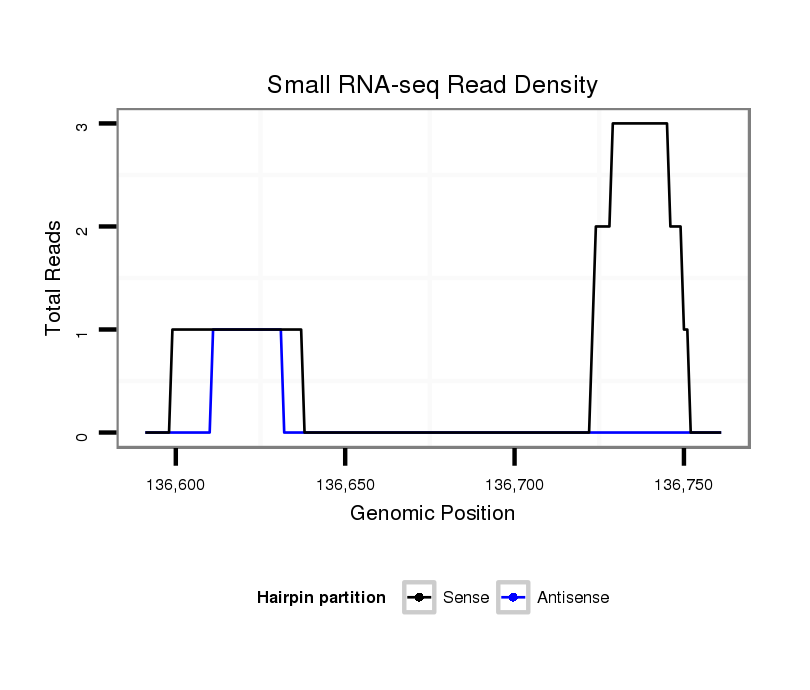
ID:dvi_986 |
Coordinate:scaffold_12472:136641-136711 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ18533-cds]; exon [dvir_GLEANR_3331:1]; exon [dvir_GLEANR_3331:2]; CDS [Dvir\GJ18533-cds]; intron [Dvir\GJ18533-in]
No Repeatable elements found
| ----------------------------######################-----------------------------------------------------------------------################################################## TGAGCCGAGCTTTCTAACTGCTCAAAATATGGATATGGAGGCTGAAAAAGGTATTACACAAAAAAGAGGGGAAGAAAACCTTAGCGTTAGCGTGCTTATGCGTCCACATTTTGTTTTCTAGGCGAAGCGTTCAGCAAGGAACCGGATGGTGACGCGGCTCTGACATCGGAT **************************************************..............((((((.((((........((((.((....)).)))).......)))).))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060665 9_females_carcasses_total |
M061 embryo |
SRR060659 Argentina_testes_total |
SRR060669 160x9_females_carcasses_total |
SRR060675 140x9_ovaries_total |
V047 embryo |
V053 head |
V116 male body |
M027 male body |
SRR060682 9x140_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........GCTTTCTAACTGCTCAAAAT............................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................TACACAAAAAAGAGGGGAAGTA............................................................................................... | 22 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................CAAGGAACCGGATGCTGA................... | 18 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................GAACCGGATGGTGACGCGGCTCT.......... | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................GCAAGGAACCGGATGGTGACGCGGCT............ | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................AGCAAGGAACCGGATGGTGACGC................ | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................ATCAACAAGGAACCGGATGCTGA................... | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................ATGGATATGGAGGCTGAAA............................................................................................................................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................AAGGAACCGGATGCTGAGG................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................AACCTTTCAGCACGGAACC............................ | 19 | 3 | 12 | 0.33 | 4 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 1 |
| ........................AAATATGGATATGGTGGT................................................................................................................................. | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................CAAACCTTAGCATTAGCGC.............................................................................. | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................................................................AAACCTTAGCATTAGCGCA............................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
ACTCGGCTCGAAAGATTGACGAGTTTTATACCTATACCTCCGACTTTTTCCATAATGTGTTTTTTCTCCCCTTCTTTTGGAATCGCAATCGCACGAATACGCAGGTGTAAAACAAAAGATCCGCTTCGCAAGTCGTTCCTTGGCCTACCACTGCGCCGAGACTGTAGCCTA
**************************************************..............((((((.((((........((((.((....)).)))).......)))).))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
V053 head |
SRR060665 9_females_carcasses_total |
SRR060662 9x160_0-2h_embryos_total |
SRR1106730 embryo_16-30h |
|---|---|---|---|---|---|---|---|---|---|---|
| ....................GAGTTTTATACCTATACCTCC.................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................TCGTTCCTAGGTCTACCACTAC................. | 22 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................TGAAATCGCACTGGCACGAA.......................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 |
| ....GGCTCAAAGGATGGACGAG.................................................................................................................................................... | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 |
| .......................................................................................................................TCCGCTTGCCAAGTAGTTC................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12472:136591-136761 + | dvi_986 | TGAGCCGAGCTTTCTAA---CTGCTCAAAATATGGATATGGAGGCTGAAAAAGGTATTACACAAAAAAGAGGGGAAGAAAACCTTAGCGTTAGCGTGCTTATGCGTCCACATTTTGTTTTCTAGGCGAAGCGTTCAGCAAGGAACCGGATGGTGA------------------CGCGGCTCTGACATCGGAT |
| droMoj3 | scaffold_6308:1417192-1417371 - | TGTACCAAATTTTCTAAAAACT--TTCAAGTATGGATATGGGGGCTGAAAAAGGTATTGTACAAAAAAATGGTGGATAAA----CAGCGTTAGCGTGCTTATGCGTCTACACTTTGTGTTCTAGGCGAAGCGTTCAACAAAGAGCCGGACGGCGGCAAAGTCACGGACGTGGTCGCGGCCTTGCC------C |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
Generated: 05/17/2015 at 05:00 AM