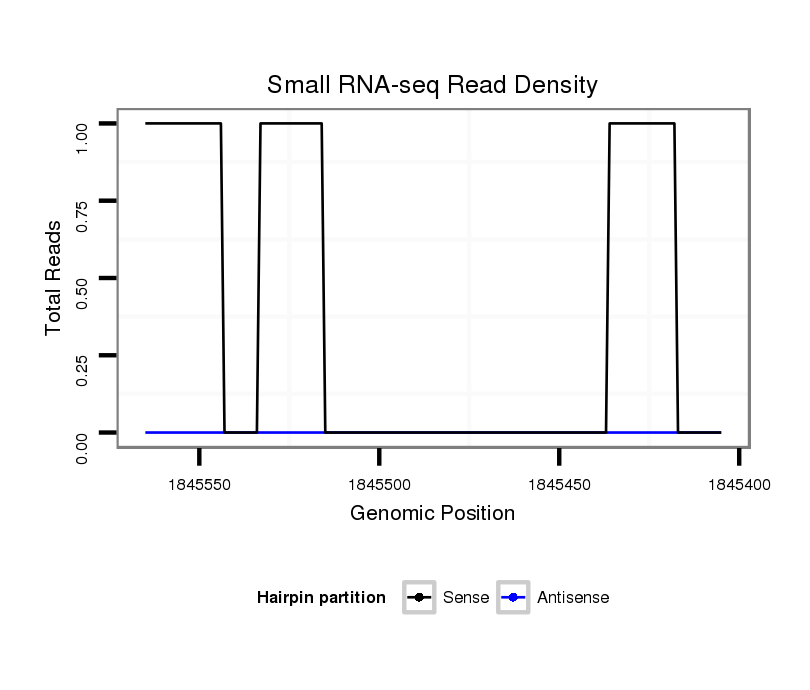
ID:dvi_9523 |
Coordinate:scaffold_12932:1845455-1845515 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ14714-cds]; exon [dvir_GLEANR_14953:2]; exon [dvir_GLEANR_14953:1]; CDS [Dvir\GJ14714-cds]; intron [Dvir\GJ14714-in]
No Repeatable elements found
| ##################################################-------------------------------------------------------------################################################## GGTGCGAAGAGGCACAACGGATGACGAGAGAAATCCGCTGGAGATTTTGGGTGAGCTGCTTTTTAGATATGGTGCATGCCGTACAGATAGAGAGCATTCTTGTATTTATAGATATAACCGATGAGACGCAAACAGCTGAATTAGAGGAGGAGATCATGGAC **************************************************..(((.(((((((((..((((((....))))))....))))))))))))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060663 160_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
SRR060659 Argentina_testes_total |
SRR060661 160x9_0-2h_embryos_total |
V116 male body |
SRR060666 160_males_carcasses_total |
M047 female body |
SRR060671 9x160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| GGTGCGAAGAGGCACAACGGAT........................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................ATCCGCTGGAGATTTTGG............................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................AAACAGCTGAATTAGAGGA............. | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................CTGGAGATTTTGGGTGAAAT........................................................................................................ | 20 | 2 | 4 | 0.50 | 2 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................GCTGAAAGAGAGAAGGAGATCA..... | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................ATCTGGAGATTTTGGGTGA........................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................GGAGATTTTGGGTGCTCT........................................................................................................ | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................GACAAATCCGCTGAAGAT.................................................................................................................... | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................GCTGAAAGAGAGAAGGAGATC...... | 21 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................................................CATTCTTGGATTTATTGA................................................. | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
CCACGCTTCTCCGTGTTGCCTACTGCTCTCTTTAGGCGACCTCTAAAACCCACTCGACGAAAAATCTATACCACGTACGGCATGTCTATCTCTCGTAAGAACATAAATATCTATATTGGCTACTCTGCGTTTGTCGACTTAATCTCCTCCTCTAGTACCTG
**************************************************..(((.(((((((((..((((((....))))))....))))))))))))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|
| ...........CGTGTTGCCTGGTGCACTCT.................................................................................................................................. | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 |
| ................................................CCCACTCGACGACACATATA............................................................................................. | 20 | 3 | 18 | 0.06 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12932:1845405-1845565 - | dvi_9523 | GGTGC------GAAGAGGCACAACGGATGACGAGAGAAATCCGCTGGAGATTTTGGGTGAGCTGCTTT------TTAGATATGGTGCATGCCGTACAGATAGAGAGCATTCTTGTATTTATAGATATAACCGATGAGACGCAAACAGCTGAATTAGAGGAGGAGATCATGGAC |
| droMoj3 | scaffold_6473:5405803-5405953 - | GACTC------CAAAAGTTG---CGAATGCTGAATCAAGTCCGTTGGAGATTTTGGGTAAGCAGCTCT------ATA-ATATTGTCGATGCACGGTATATA------ACTCTCACATTTACAGACGATGACGAAGCGAAGCCAACACTGGTTCTCAACGAAGACACCGGGGAA | |
| droGri2 | scaffold_14862:11160-11287 - | GATACTTGAGCTAAAAGCCATAAAGAATGACGAAACAAATCCGCTGTAGACTTCAGGTGAGTTGCCCTGATGATTCAAAAATGTTGG------AACACATAAAAGCCATTACTTTATTCATAGAC---------------------------------------ATGGGTAAC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/17/2015 at 05:00 AM