
ID:dvi_952 |
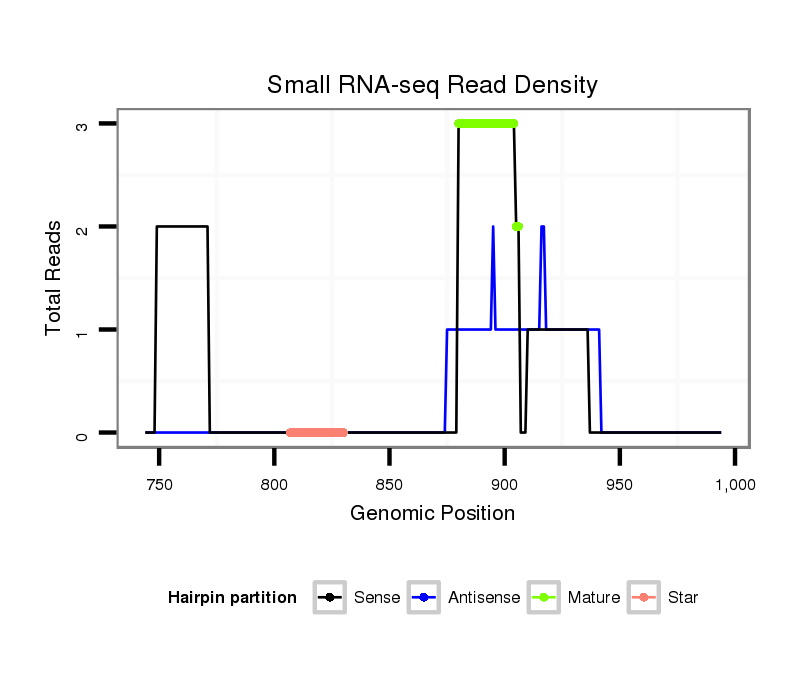
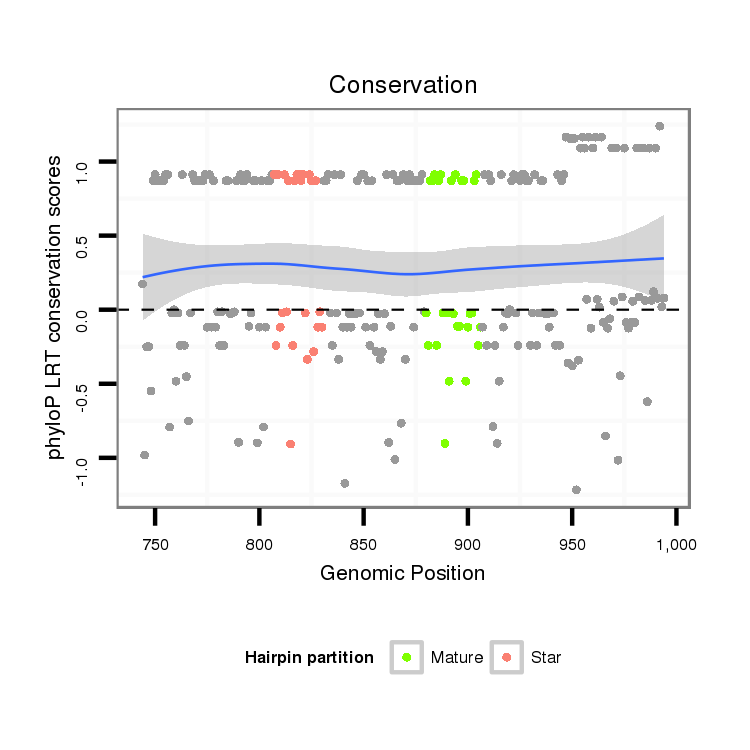
Coordinate:scaffold_12177:794-944 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -25.5 | -25.4 | -25.2 |
 |
 |
 |
CDS [Dvir\GJ22498-cds]; exon [dvir_GLEANR_7800:2]; intron [Dvir\GJ22498-in]
No Repeatable elements found
| mature | star |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GTATTCACCCGAAGACTTCTGTTTTGGGCCAATTGTGCCGCCATGGCTGGACACCTTGGAGGATCAATTCGGGGTAGATGTCCGAGACCAACATCAAATTTTAGATTTCGGTCTGGAACCAGCGATCGTCTGCCAGTCGGACGTTATTGAAGGGCTTTTCAGTTTCTCACTAATTAGCTTCGATGGCATCTGGTATTACAGGTTCGGCAGCACCTGCAGAATACCCTCTGGCAGAATGCCGGAGGTGCTGG ***************************************************************....((((((((((((((((.(((((...(((........)))...)))))))).........))))))))..)))))....(((((((....)))))))**************************************************************************************** |
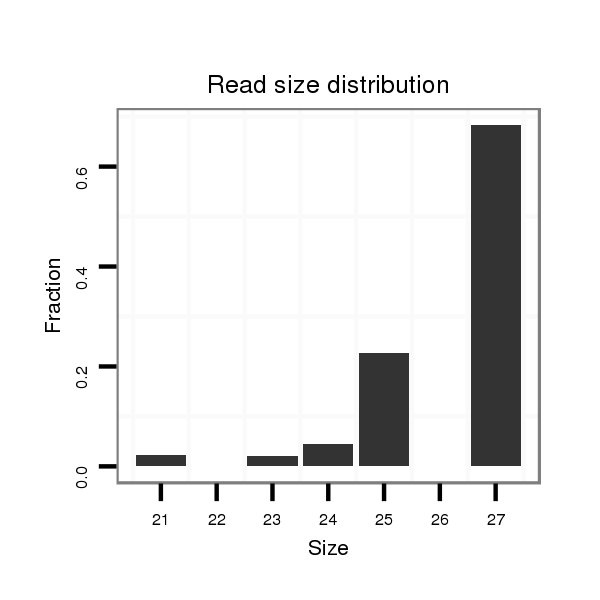
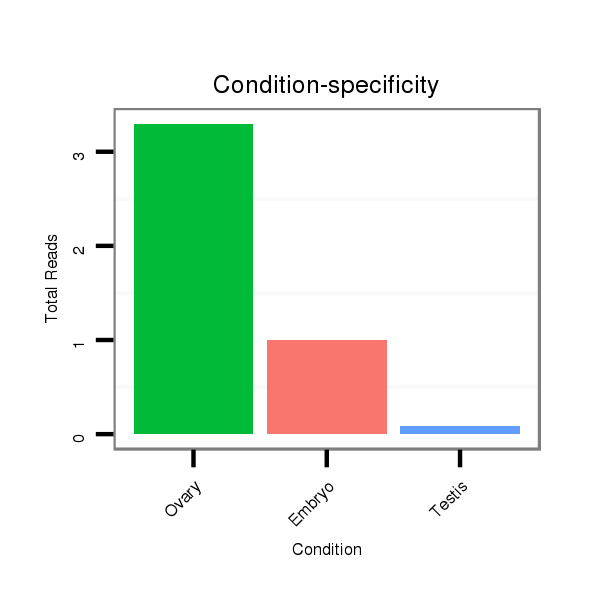
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060670 9_testes_total |
SRR060673 9_ovaries_total |
SRR060658 140_ovaries_total |
M047 female body |
SRR060654 160x9_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060675 140x9_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060674 9x140_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060683 160_testes_total |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....CACCCGAAGACTTCTGTTTTGGG............................................................................................................................................................................................................................... | 23 | 0 | 1 | 2.00 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................TCGGACGTTATTGAAGGGCTTTTCAGT........................................................................................ | 27 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................TCACTAATTAGCTTCGATGGCATCTGG.......................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................TCGGACGTTATTGAAGGGCTTTTCA.......................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................TCCGGTATTACAGGTTCGGCAGCACC..................................... | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................GCTGAATGCAGGAGGTGCTGG | 21 | 2 | 3 | 0.67 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................TGGCAGGAGACCTTGGAGG............................................................................................................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................TGGCGTCCGGTATTACAGGTTCGG............................................ | 24 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................AGAATACCGGAGGTGATG. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................TGTACCAGTTGTGCCGCCA................................................................................................................................................................................................................ | 19 | 3 | 20 | 0.20 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 2 | 0 | 1 |
| ...............................................................TCAATTCGGGGTAGATGTCCGAGA.................................................................................................................................................................... | 24 | 0 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................ACATCAAATTTGAGGTTTCGA............................................................................................................................................ | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................AGCGATCGTCTGCCAGTCGGA.............................................................................................................. | 21 | 0 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................AGGATCAAATCGGGGTTGT............................................................................................................................................................................. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................GAACCAGCGATCGTCTGCCAGTC................................................................................................................. | 23 | 0 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .....................TTTTGGGTCGATTGGGCCGC.................................................................................................................................................................................................................. | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GTTGGCATCTGGTGTTACC................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
CATAAGTGGGCTTCTGAAGACAAAACCCGGTTAACACGGCGGTACCGACCTGTGGAACCTCCTAGTTAAGCCCCATCTACAGGCTCTGGTTGTAGTTTAAAATCTAAAGCCAGACCTTGGTCGCTAGCAGACGGTCAGCCTGCAATAACTTCCCGAAAAGTCAAAGAGTGATTAATCGAAGCTACCGTAGACCATAATGTCCAAGCCGTCGTGGACGTCTTATGGGAGACCGTCTTACGGCCTCCACGACC
****************************************************************************************....((((((((((((((((.(((((...(((........)))...)))))))).........))))))))..)))))....(((((((....)))))))*************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060673 9_ovaries_total |
SRR060677 Argx9_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
V053 head |
V116 male body |
M047 female body |
SRR060654 160x9_ovaries_total |
SRR060679 140x9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR1106727 larvae |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................AGAAGATGGTTAGCCTGCAAT......................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................CGGTCAGCCTGCAATAACTTC................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................TAATCGAAGCTACCGTAGACCATAAT..................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................CTGTCAGCCTGCAATAACTTC................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................CCCGAAAAGTCAAAGAGTGATTA............................................................................. | 23 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................TAGAAGATGGTTAGCCTGCAAT......................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................CGGCGGTACCGACCCCTGTAA.................................................................................................................................................................................................. | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................CGACAAGTGGAACCTCCTA........................................................................................................................................................................................... | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................GTAGGCCAGTATGTCCAAG.............................................. | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................................CCGTCTTAGGTCCTACACGAC. | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................CGTCTTAGGCGCTCCACGAC. | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................CTAAACTACCGTAGACCAT........................................................ | 19 | 2 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................GTCGCTAGCAGACGGTCAGCCTG............................................................................................................. | 23 | 0 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................GGTCGCTAGCAGACGGTCAGC................................................................................................................ | 21 | 0 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................TGGTTGTACTTTGAAAGCTA................................................................................................................................................. | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................TCTTGGTCGCTAGCAG......................................................................................................................... | 16 | 1 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................TTGGTCGCTAGCAGCCTG..................................................................................................................... | 18 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................CTGTGGAACCTGCTAGTGT....................................................................................................................................................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................................AGCTTCGTCGCTAGCAGCC....................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12177:744-994 + | dvi_952 | GTATTCACCCGAAGACTTCTGTTTTGGGCCAATTGTGCCGCCAT-------GGCTGGACACCTTGGAGGATCAATTCGGGGTAGATGTCCGAGACCAACATCAAATTTTAGATTTCGGTCTGGAACCAGCGATCGTCTGCCAGTCGGACGTTATTGAAG-GGCTTTTCAGTTTCTCACTAATTAGCTTCGATG-GCATCTGGTATTACAGGTTCGGCAGCACCTGCAGAATACCCTCTGGCAGA----------------ATGCCGGAGGTGC---TGG |
| droAna3 | scaffold_7028:1009-1074 + | GT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCGACGGCACCTGCAGAATACGCCCGCGCAGATGATAATTACG*ATACGTGCCGTCGTCGT---TCT | |
| droEle1 | scf7180000491223:12738-12753 + | ATGCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGACG-------------------------------------------------------ACTTCG | |
| droRho1 | scf7180000772938:1010-1270 + | GGATCCACCCGAATG-CCGTCTTCTGGGCCACTGGGAGTCCCTCGTCGACCAGATGGAAGCCCGGTCGGATGACCTTGGCGTAGACATCCGCACCCAGGACTACCGAGATGGTTGCCGGCAAGTAAAAACGTTTGTCGGCCAACGGGAGGTCTCCGTATCTGCCGACCACGGTGTCCGAGAGCT-CTCGGATGGGGGTGCGGATGCGGACGTTCGGCTCCACTTTCAAGACTACCTCTAGCTTG----------------AAGCCGTTGTCGCACTTCG | |
| droTak1 | scf7180000411255:10314-10574 + | GGATCCACCCGAAAG-TCGTCCGCTGGGCCACTGGGAGTCCCTCGTCGACCAGGTGGAAGCCAGGACGGATGACCTTGCCGTAGACGTCTGCACCCAGGACCACGGAGATGGTTGCCGGTAGATAGAACCGCTCGTCGGCCAACGGGAGGTCGCTGTATTTGCTGACCACGGTGTCAGCAAGCT-CTCGGATGGGGGTGCGGATGCGGACGTTTGGCTCCACCTTCAGAACCACCTCCAACTTG----------------AAGTCGTCGTCGCGCTTCG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/17/2015 at 04:43 AM