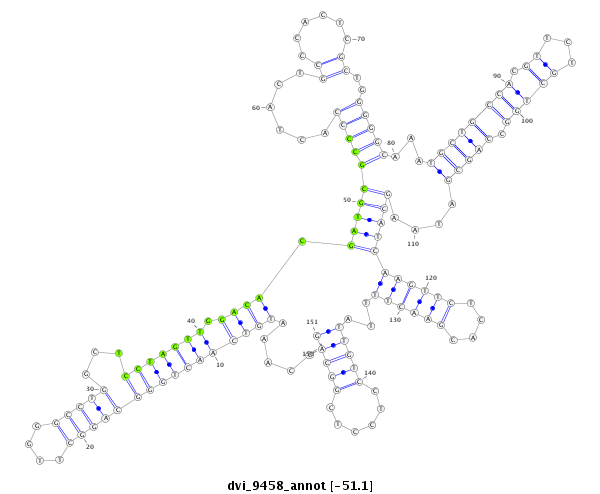
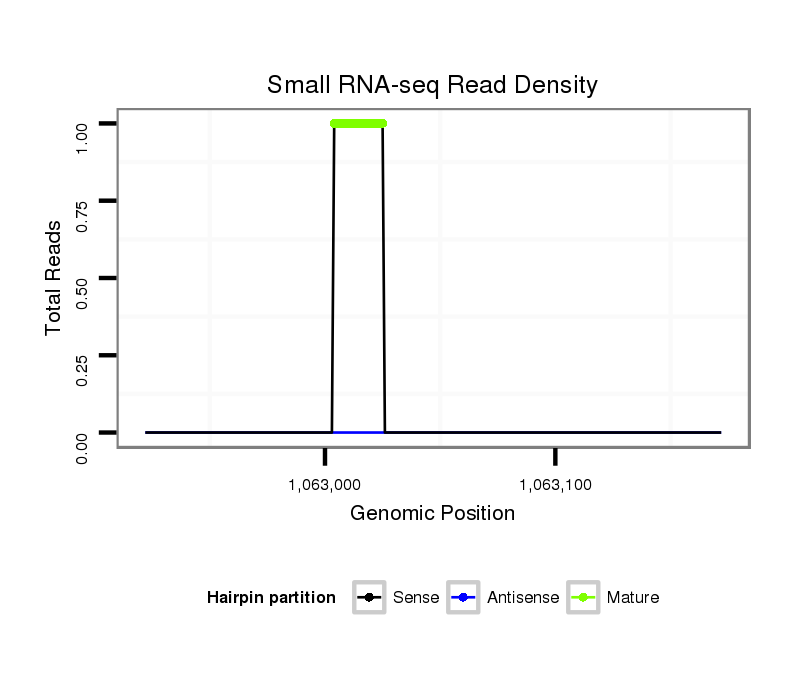
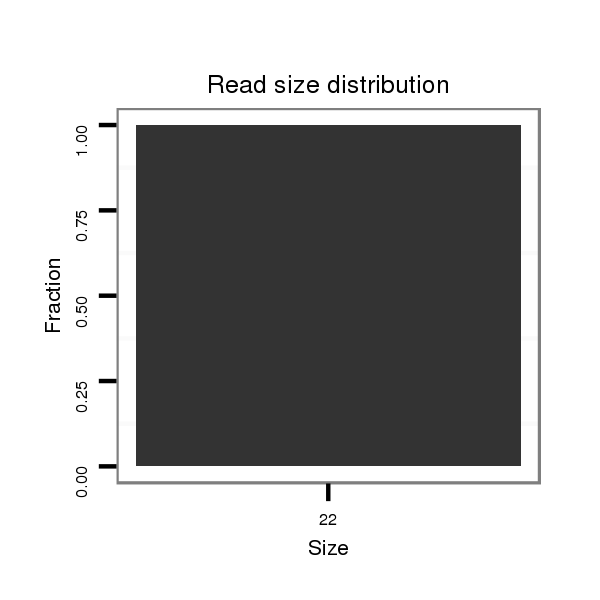
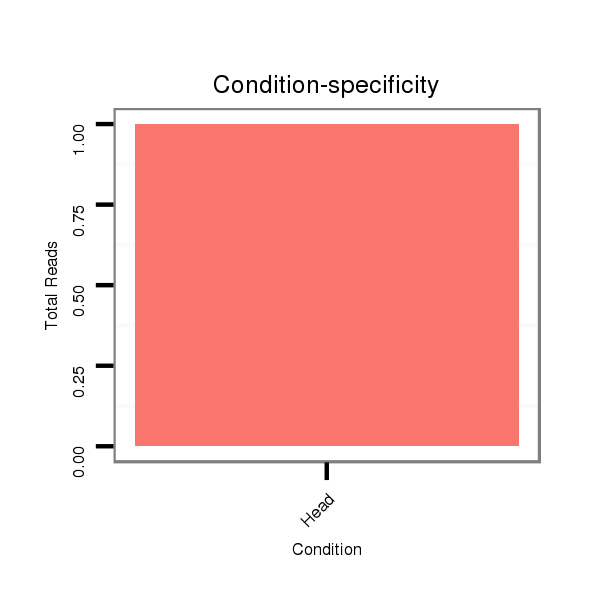
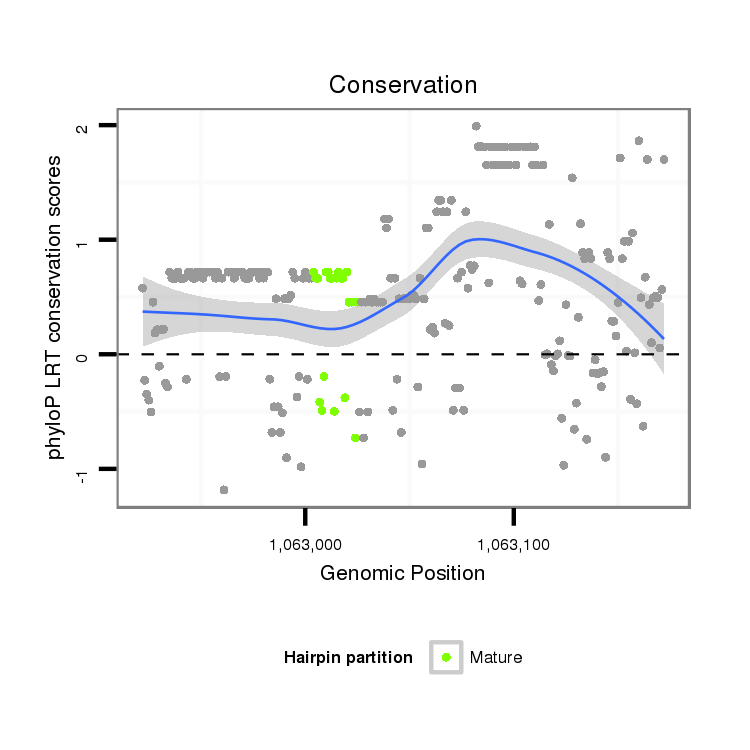
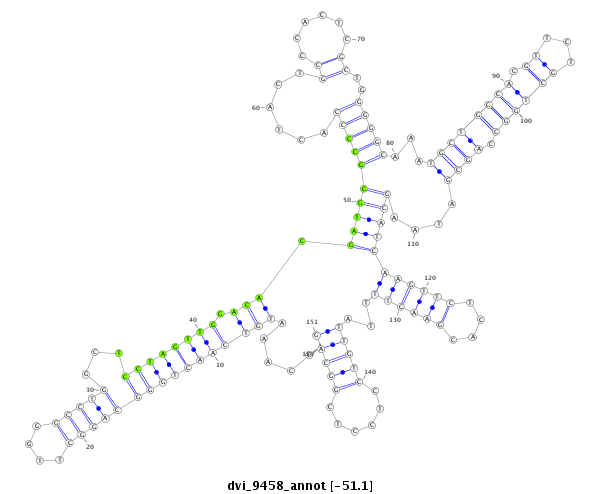
ID:dvi_9458 |
Coordinate:scaffold_12932:1062972-1063122 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -51.1 | -51.1 | -51.1 |
 |
 |
 |
CDS [Dvir\GJ14845-cds]; exon [dvir_GLEANR_15080:2]; intron [Dvir\GJ14845-in]; Antisense to intron [Dvir\GJ14741-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CTGGTCGGGCTTTTGCGTGGCATTCGAATGTTAATCAGGAGAATCAATTGCCAAATGTCAACTGGGCAGGCTTGGGCCTGGCTCCTAGTTGGACACGATGCGCCCCACTACTGCCCACTCGCTGGGGGCAAATGCTGCCACGTTCTGCTGGCCAGCGATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTCCTCGGCAGCAGTTGGAGCTGGAGTCGAGTCTGAGGCTGGAGTTGAAGCCGGGCGCCGC **************************************************.....((((((((((((((((....)))))...))))))).)))).((((((((((......((......))..)))))...((((((((.((...))))).)))))....)))))((((((....))))))...(((((......)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
M061 embryo |
M047 female body |
SRR060664 9_males_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................TCCTAGTTGGACACGATGCGCC................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................TCTGGGGGTGGAGTGGAAGC.......... | 20 | 3 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................TATAATGTTAATCAGGAGAC................................................................................................................................................................................................................ | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................ATAATGTTAATCAGGAGA................................................................................................................................................................................................................. | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............................................................CTGGGCAGCCTTGGGCGTGC.......................................................................................................................................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................CGGGCTTGGGGCAAATGCTG.................................................................................................................. | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................TGGCCAGCGATACCCAGCA.................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GACCAGCCCGAAAACGCACCGTAAGCTTACAATTAGTCCTCTTAGTTAACGGTTTACAGTTGACCCGTCCGAACCCGGACCGAGGATCAACCTGTGCTACGCGGGGTGATGACGGGTGAGCGACCCCCGTTTACGACGGTGCAAGACGACCGGTCGCTATTCGTAGTTCAAGAGTGCTTGAAAATAACAGGAGGAGCCGTCGTCAACCTCGACCTCAGCTCAGACTCCGACCTCAACTTCGGCCCGCGGCG
**************************************************.....((((((((((((((((....)))))...))))))).)))).((((((((((......((......))..)))))...((((((((.((...))))).)))))....)))))((((((....))))))...(((((......)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060676 9xArg_ovaries_total |
SRR060680 9xArg_testes_total |
SRR060667 160_females_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060678 9x140_testes_total |
V116 male body |
M028 head |
SRR060674 9x140_ovaries_total |
M027 male body |
SRR060687 9_0-2h_embryos_total |
SRR060675 140x9_ovaries_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR1106728 larvae |
SRR060660 Argentina_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................................................................ACCTCGCCCTCTGCTCAGA........................... | 19 | 2 | 2 | 3.50 | 7 | 3 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ACCTCGCCCTCTGCTCAG............................ | 18 | 2 | 3 | 1.00 | 3 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................GAGGTGAGGACGGATGAGC.................................................................................................................................. | 19 | 3 | 19 | 0.58 | 11 | 0 | 0 | 0 | 0 | 0 | 0 | 8 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 1 |
| .............................................................................................................................................................................................................ACCTCGCCCTCTGCTCAGAG.......................... | 20 | 3 | 8 | 0.38 | 3 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ACCTCGCCCTCTGCTCAGAT.......................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................CTTACGATTAGTCCTCTTC............................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................GGGTGAGGACGGATGAGC.................................................................................................................................. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ACCTCGCCCTCTGCTCAGAGT......................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ACCTCGTCCTCTGCTCAG............................ | 18 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ACCTCGCCCTTTGCTCAGA........................... | 19 | 3 | 14 | 0.14 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................................GGACGGATGAGCGACCCCG........................................................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................CCTCTCAGTTAACGGTG..................................................................................................................................................................................................... | 17 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................................................................TAGGAGCCGTCTTGAACCT.......................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12932:1062922-1063172 + | dvi_9458 | CTGG-TCGG----GCTTTTGCGTGGCATTCGAATGTTAATCAGGAGAATCAATTGCCAAATGTCAACTGGGCAGGC-------------------TTGGGCCTGG-----------------------------------CTCCTAGTTGGACACGATGCGC--CCCACTACTGCCCACTCGCTGGGGGCAAATGCTGC-----------C--ACGTTCTGCT-------------------------------------------------------------------GGC---CAGCGATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTCCTCGG---CAGC----AGTTGGAG------C------------------------------TGGAGTCGAGTCTGAGGCTGGAGTTGAAGCCGGGCGCCGC- |
| droMoj3 | scaffold_6473:14879879-14880206 + | CGATGCCGGAGCTGCCCCTGCGTGGCATTCGAATGTTAATCAGGCGAATCAATTGCCAAATGTCAAT-----------------------------TGAGTCTAGCTCTCGGGCGCCTGGCTAGCCCGGTCTGGTCTGGCCTCCACGTTGGCCACGATGCGACGCTCCCCACTGCCCACTCGCT--------ACGGTGC-----------CAGACGTTCTGCTAGCTGCTGCTGCTCCTTCTGCTGCTGCTGCTGCTGCTGCTGTTGCTGTTGCTGTTGCTGTTGCTGGTGGC---CAGCGATAAGCATCAAGTTCTCACGAACTCGTATTGTCCTCTTCGAGTT-G--CAGTTGCAGTTGG--------------------------------------------------------GAGCTGCAGTTGGGCGCCGC- | |
| droGri2 | scaffold_14853:4668771-4669051 - | CTTC---GA----CTCTCTGCGTGGCTTTCGAATGTTAATCAAGGAAATCAATTGCCAAATGTCAACGAGAATGCCTTGGCTTTGCGGACTTCCCTTGAAACTGG-----------------------------------CTCCACATTGGACACGCT----------------------CCCAGTGGGCAA-CG---------------T--TCGTTCTGCT-------------------------------------------------------------------CTCGTGAACCGATAAGCATCGAGTTCTCACGAACTTTTATTGTCGTCCTC---CTGT---CGTGGACGCT--AGACGCTGGCTCTGGCGCTGGCGCTGGCGCTGAAGTTGCAGCCAGATTTGAAGTTGGAGTTTTAGCTGGGCGCCGC- | |
| droPer2 | scaffold_1:7855388-7855435 + | G-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGGAGC-------------------------------------------------------------------------------------------TGGAGCTGGAGGTGGAGCTGGCGCTCAAGGTGGCCGCCGC- | |
| droAna3 | scaffold_13117:2881660-2881740 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTCAGCTAGCG-AGTCGAC-TACGACAAAGA--------------------------------------------------CT------GGAATTGCAAGTTGC- | |
| droBip1 | scf7180000396696:254933-255012 - | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATAAGCATCAAGTTCTCACGAACTTTTATTGTCCCCAACGACCA-A--GACTCGACGACAAAGA--------------------------------------------------CT------GGAATTGCAAGTTGC- | |
| droEle1 | scf7180000491044:1496521-1496608 - | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTCAA------GAGG----AGACGGAGAAGACGCTGGC--------TGGC----------------------------AACCT------GGAATTGCAACTGCCA | |
| droRho1 | scf7180000779588:504568-504686 + | CCGC---TG----CCC-----------------------------------------------------------------------------------------------------------------------------------------------------------------ACT-----------------GCGGCATTGTCTGC--AAGTTCAGTT----------------------------------------------------------------------------CGATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTCAA------GAGG----AGACGGCGAAGACGC------------TGGC----------------------------AACCT------GGAATTGCAACTGGC- | |
| droTak1 | scf7180000415381:258920-258957 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTC------------------------------------------------------------------------------------------------------ | |
| dm3 | chrX:7113718-7113791 + | CCGC---TG----CCC-----------------------------------------------------------------------------------------------------------------------------------------------------------------ACT-----------------GCGGCATTGTCTGC--AAGTTCAGTT----------------------------------------------------------------------------CGATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTC------------------------------------------------------------------------------------------------------ | |
| droSim2 | x:6694770-6694887 + | CCGC---TG----CCC-----------------------------------------------------------------------------------------------------------------------------------------------------------------ACT-----------------GCGGCATTGTCTGC--AAGTTCAGTT----------------------------------------------------------------------------CGATAAGCATCAAGTTCTCACGAACTTTTATTGTCCTCAA------GAC------GACGGCGAAGACGC------------TGGA----------------------------AACCT------GGAACTGCAACTGGCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
Generated: 05/17/2015 at 04:24 AM