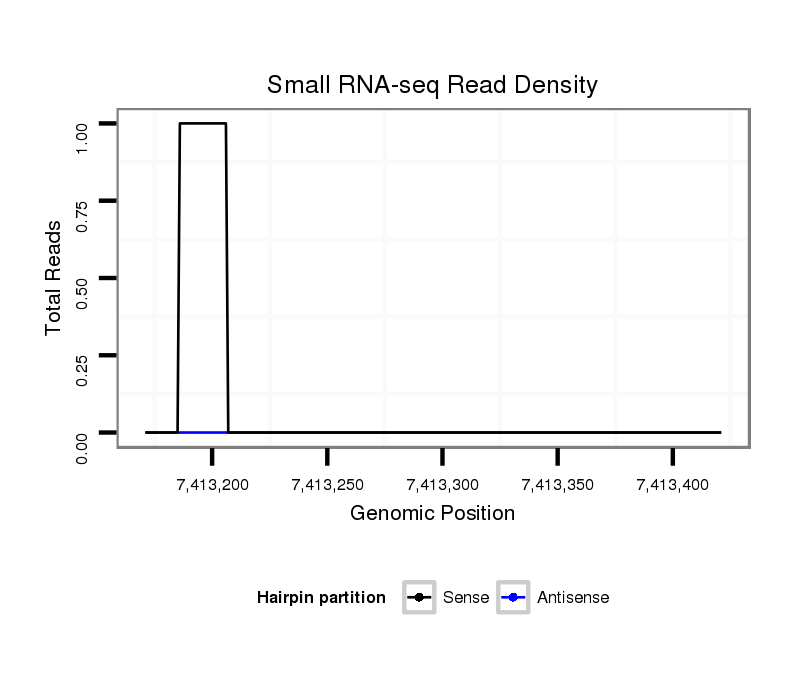
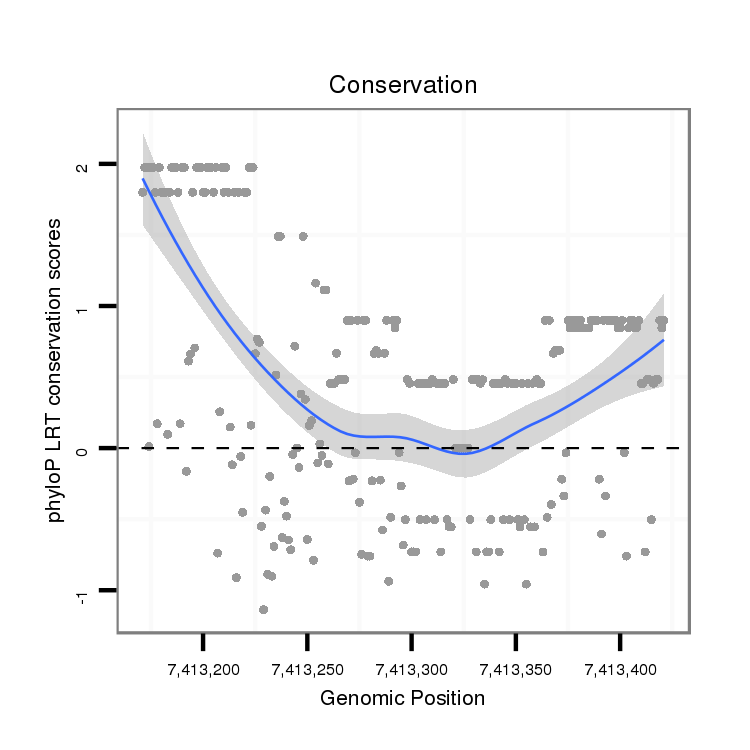
ID:dvi_9266 |
Coordinate:scaffold_12928:7413221-7413371 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ17060-cds]; exon [dvir_GLEANR_17561:1]; intron [Dvir\GJ17060-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GAATTTCCTGCGCCAAACATTCAACGTTACGAAACAATTGACGGCACAAGGTAAGTTTTAACAAAAATCCCTATTTAAAACATTGCGTTGCGGCTATTTATTCAACTTCGAGTCCCGTGTTCTATATACATACGCGCCCACGCGCCTAATTGCCAACCATGTGAGGCAGCCGGTAGCCGCTTGCGCACATCCATTTCCAATTGGTGACTGTGCGCATTTTGTGTTTATCCAAGGATCCTGCGTTCCATTGT **************************************************....(((((((.............)))))))..(((((.(((((((((.((((......))))...((((.....))))....((((....))))....(((((..........)))))..)))))))))..)))))..............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
M047 female body |
SRR060663 160_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
V047 embryo |
SRR060674 9x140_ovaries_total |
SRR060671 9x160_males_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
M028 head |
SRR060668 160x9_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060664 9_males_carcasses_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............AACATTCAACGTTACGAAACA....................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................CGAGGCAGCCGGTACCCGCCT..................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................TAGCCAAGGATCCTGCATT....... | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................CTAATTAAAACATAGCGTTGC................................................................................................................................................................ | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................ACGAAACAATTGACGGCACAAGC........................................................................................................................................................................................................ | 23 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................TTAAAACATAGCATTGCG............................................................................................................................................................... | 18 | 2 | 18 | 0.72 | 13 | 0 | 1 | 0 | 0 | 0 | 0 | 3 | 3 | 0 | 0 | 2 | 2 | 0 | 0 | 1 | 1 |
| ................................................................................................................................................................................................AGTGCCAATTGGTGACTGT........................................ | 19 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................CGACACAAGGTAAATTTTA............................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................ATTTATTCAGGTTCGACTCC........................................................................................................................................ | 20 | 3 | 13 | 0.15 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................ATTTAAAACATAGCATTGC................................................................................................................................................................ | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................CTACTTAAAACATAGCATTGC................................................................................................................................................................ | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................GAAGGCACAAGGGAAGTCT................................................................................................................................................................................................. | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......CTGCGCCGAAGATGCAACG................................................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
CTTAAAGGACGCGGTTTGTAAGTTGCAATGCTTTGTTAACTGCCGTGTTCCATTCAAAATTGTTTTTAGGGATAAATTTTGTAACGCAACGCCGATAAATAAGTTGAAGCTCAGGGCACAAGATATATGTATGCGCGGGTGCGCGGATTAACGGTTGGTACACTCCGTCGGCCATCGGCGAACGCGTGTAGGTAAAGGTTAACCACTGACACGCGTAAAACACAAATAGGTTCCTAGGACGCAAGGTAACA
**************************************************....(((((((.............)))))))..(((((.(((((((((.((((......))))...((((.....))))....((((....))))....(((((..........)))))..)))))))))..)))))..............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060662 9x160_0-2h_embryos_total |
M047 female body |
SRR060687 9_0-2h_embryos_total |
SRR060659 Argentina_testes_total |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................AACGCGTGGTGGTAAAGGTT................................................... | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................GCGAACTCGCGTAGGTAAA....................................................... | 19 | 2 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................GTGTAGGAAAAGGTGAACC............................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................ACCGCGTCGGCCATCGGGGA...................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................................................................GTGCGAACTCGTGTAGGTAAA....................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................AGATATATGAGTGAGCGGGT............................................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................................ACCGCGTCGGCCATCGGC........................................................................ | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................GAACTCGCGTAGGTAAAAGT.................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................GATGTAAAATTGTTTTTAGGG.................................................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .........................................................................................................................................................................................GTGTAGGGAAAGGTGAATC............................................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................TGCGAACTCGCGTAGGTAAA....................................................... | 20 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:7413171-7413421 + | dvi_9266 | GAATTTCCTGCGCCAAACATTCAACGTTACGAAACAATTGACGGCACAAGGTAAGTTTTA-ACAAAAATCCCTATTT--AAA-----ACATTGCGTTGCGGCTATTTATTCAACTTCG-------------------------------------AGTCCCGTGTTCTATATACATACGCGCCCACGCGCCTAATTGCCAACCATGTGAGGCAGCCGGTAGCCGCTT--------GCGCACATCCATTTCCAATT-GGTGACTGTGCGCATTT-T---------------------GTGTTTATCCAAGGATCCTGCGTTCCATTGT------ |
| droMoj3 | scaffold_6473:2636797-2637042 - | GAATTTCCTGCGCCAAACATTCAACGTTACGAAACAGTTGACGACCCAGGGTAAGTTCGGCCCGC-AAGCTTAAT-C-GAAACTTATATA---TGTTCCGG-TATTTGTCCATATTGCAG------------------------------------------TTGTCTAAGCACCGCCACGTCCATGCTCCCTTT-------TATTTGACTAGGCCTGCAGTCGCCAATCAACGGGTCCTCTTCCCTGT-----CAAGTGACTGTGCGCATTTTCT--------------------CTATTTATCCAACGATCCTGCTTTTCATTGT------ | |
| droGri2 | scaffold_15081:3294868-3294940 - | GAACTTCTTGCGTCAAACATTCAACGTTACGAAACAATTGACGGCCCAAGGTAAACG-------AAAAATCCAAT-T-AGAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XL_group1a:3513714-3513938 - | GAACTTCCTGCGGCAAACATTCAGCATTACGAAACAATTGACGGCGCAGGGTAAGTTTG-CCTGC-AAGTTGGC--CAAAAACT-----G-TC--------C----TATCTATTTTGCAGAGGGTCCCTTCAGACAGCCCCTTCAAGATCGGTCTGGTCTGGTCTTCTGTT---------------------------------------------------------------------------TTTACAATC-AATGACTGTGCGCATTT-CTCCCCCTCGCCGTCTGTGCAAGTATTTATCCAGCGATCCT---------TGTCGCGTC | |
| droPer2 | scaffold_11:55156-55385 - | GAACTTCCTGCGGCAAACATTCAGCATTACGAAACAATTGACGGCGCAGGGTAAGTTTG-CCTGC-AAGTTGGC--CAAAAACTTGACTG-TC--------C----TATCTATTTTGCAGAGGGTCCCTCCAGACAGCCCCTTCAGGATCGGTCTGGTCTGGTCTTCTGTT---------------------------------------------------------------------------TTTACAATC-AATGACTGTGCGCATTT-CTCCCCCTCGACGTCTGTGCAAGTATTTATCCAGCGATCCT---------TGTCGCGTC | |
| droKik1 | scf7180000302258:468812-468872 - | GAACTTCTTGCGGCAAACTTTCGGCATTACGAAACAATTGACGACGCTGGGTGAGTTTTA-A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453927:135125-135185 - | GAATTTCCTGCGGCAAACTTTCGGCATTACGAAACACCTGACTTCGCTTGGTAAGTTCGA-A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491011:644760-644816 - | GAATTTCCTGCGGCAAACTTTTGGCATTACGAAACAACTGACTTCGCAGGGTGAGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000779506:436905-436997 - | GAATTTCCTGCGGCAAACTTTGGGCATTACGAAACAGTTGACTTCACAAGGTGAGTTCA-CCTA--AAATAAGT--TTAAAACTAAAGTAGTGCTTTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415151:473167-473260 + | GAATTTCCTGCGGCAAACCTTGGGCATTACGAAACAGTTGACTTCGCAGGGTAAGTTTAG-ACTA-AAACAAAT--TAAAAGCTCATATATTGCGTTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409277:19333-19389 - | GAATTTCCTGCGGCAAACTTTCGGCATTACGAAACAGTTGACATCCCGGGGTAAGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/16/2015 at 09:38 PM