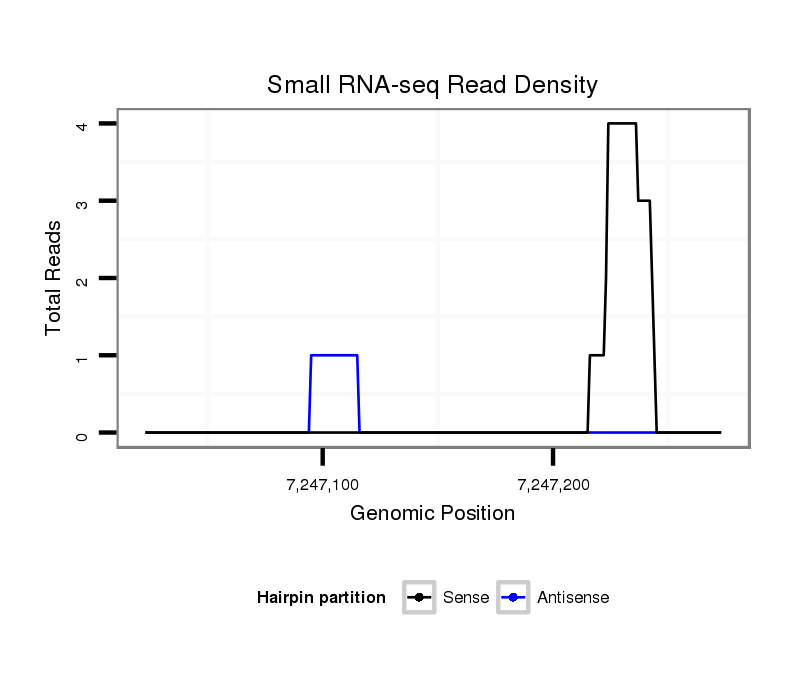
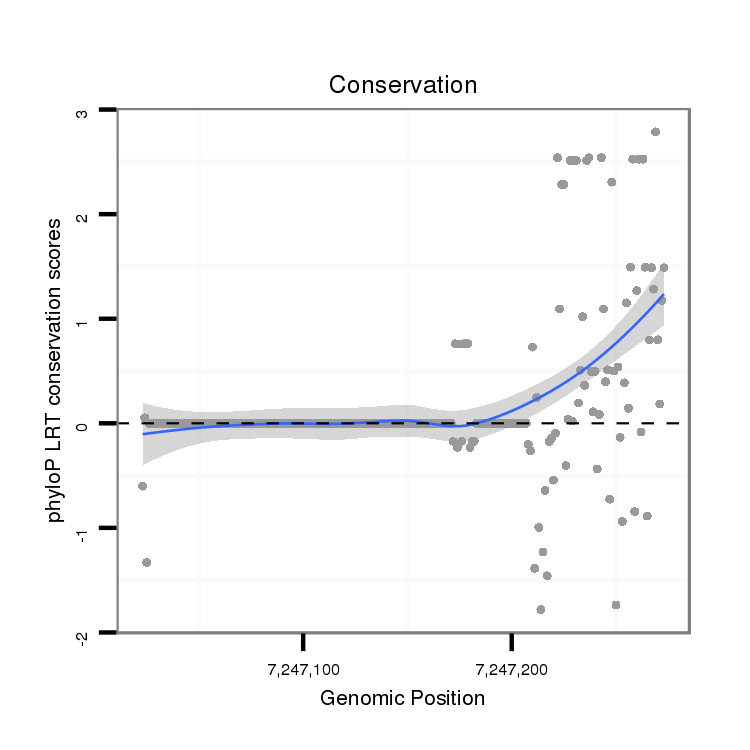
ID:dvi_9233 |
Coordinate:scaffold_12928:7247073-7247223 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_17553:3]; CDS [Dvir\GJ17051-cds]; intron [Dvir\GJ17051-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CTGAAACTTGCTTTGGAAGCTGTACGTAAACAACATTTAGAAGCATGCAATCGTTTATCGATTTTTAAAATCGATAAATCGCATTTAACCAAGTCGCATTCATCGGCGAGTTTCCCGTGCAATTGAGCGAAGTCGTCGCTGTTTATAGCCTTCTTAATGGAAATAGCCTTTTTCTTCTACCGCCCCCCCATTGACTCGCAGGCCCTCAACGCGAAGAGCATGCATCGTGGCCCCACCGCGCTCATCTATGA **************************************************..((((((((((((...)))))))))))).((((......((.((((........)))).))....))))......((((.((((..(((((((....((........)))))))))........................))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060672 9x160_females_carcasses_total |
SRR1106729 mixed whole adult body |
SRR060659 Argentina_testes_total |
SRR060667 160_females_carcasses_total |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
SRR060666 160_males_carcasses_total |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
M028 head |
SRR060665 9_females_carcasses_total |
SRR060663 160_0-2h_embryos_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........TCGGGAAGCTGTACGTAAGC............................................................................................................................................................................................................................ | 20 | 3 | 4 | 1.00 | 4 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................ACTCGCAGGCCCTCAACGCGA..................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GGCCCTCAACGCGAAGAGCAT.............................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................GCCCTCAACGCGAAGAGCA............................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................GCCCTCAACGCGAAGAGCATG............................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................TCTAGGCTTCGTAATGGAAATA...................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............GGAAGCTGTACGTAAGC............................................................................................................................................................................................................................ | 17 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TTTGGGAGCTGTACGTAA.............................................................................................................................................................................................................................. | 18 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................AAGAATGCAATCGGTTATCGAG............................................................................................................................................................................................. | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................AGAATGCCCTCAACGCGAAGA.................................. | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............GGGGAAGCTGTACGTAAGC............................................................................................................................................................................................................................ | 19 | 3 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................TTTTAATGGAAATAACTTTTTTC............................................................................. | 23 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................................TATAGCCTTCATAGTGGCA......................................................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................TACGTAAACAACATATAA................................................................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ............................................................................................................................GAGCGAAGTCGATGGTGT............................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
GACTTTGAACGAAACCTTCGACATGCATTTGTTGTAAATCTTCGTACGTTAGCAAATAGCTAAAAATTTTAGCTATTTAGCGTAAATTGGTTCAGCGTAAGTAGCCGCTCAAAGGGCACGTTAACTCGCTTCAGCAGCGACAAATATCGGAAGAATTACCTTTATCGGAAAAAGAAGATGGCGGGGGGGTAACTGAGCGTCCGGGAGTTGCGCTTCTCGTACGTAGCACCGGGGTGGCGCGAGTAGATACT
**************************************************..((((((((((((...)))))))))))).((((......((.((((........)))).))....))))......((((.((((..(((((((....((........)))))))))........................))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060671 9x160_males_carcasses_total |
SRR060689 160x9_testes_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|
| ........................................................................CTATTTAGCGTAAATTGGTTC.............................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ........ACGGAACCTTCGACATGCATT.............................................................................................................................................................................................................................. | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 |
| .........CGTAACCTTCGACATTCA................................................................................................................................................................................................................................ | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:7247023-7247273 + | dvi_9233 | CTGAAACTTGCTTTGGAAGCTGTACGTAAACAACATTTAGAAGCATGCAATCGTTTATCGATTTTTAAAATCGATAAATCGCATTTAACCAAGTCGCATTCATCGGCGAGTTTCCCGTGCAATTGAGCGAAGTCGTCGCTGTTTATAGCCTTCTTAATGGAAATAGCCTTTTTCTTCTACCGCCCCCCCAT-TG----------ACTC-------GCAGGCCCTCAACGCGAAGAGCATGCATCGTGGCCCCACCGCGCTCATCTATGA |
| droMoj3 | scaffold_6473:2825404-2825460 - | CTG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGCTGTCAATGCCAAGAGCATGCATCGCGGAGCCACAGCGCTCATCTATGA | |
| droGri2 | scaffold_15081:3460882-3460959 - | TTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TT-AAT-CTACCACCACTCGGCT-------GCAGGCCATGAATGCTAAGAATATGCAGCGTGGGGCAACAGCGCTCATCTATGA | |
| droWil2 | scf2_1100000004515:1842384-1842435 + | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCTGTCAATGCCAAAGGCATGCTACGTGGCCCCACAGCCCTGATCTTTGA | |
| dp5 | XL_group1e:12168486-12168502 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACGGCTCTGGTCTACCA | |
| droPer2 | scaffold_14:1440638-1440654 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACGGCTCTGGTCTACCA | |
| droAna3 | scaffold_13117:1453819-1453873 - | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGCAGTCAATGCCAAGGGCCTGGTCCGGAGACCCACGGCCCTCATCTATGA | |
| droBip1 | scf7180000396425:662068-662136 + | TCT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCGCC-TC----------ATCC-------GCAGGCAGTTAATGCCAAGGGTCTGGTCCGGAAACCCACGGCCCTTATCTACGA | |
| droKik1 | scf7180000302816:81238-81291 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGCCATGAATGCCAAGGGCTTGGTGCAAAGACCCACGGCCCTGATCTATGA | |
| droFic1 | scf7180000454038:465331-465382 + | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCTGTCAATTGCAAGGGTCTGGTGCGCAAGCCCACGGCCCTCATCTACGA | |
| droEle1 | scf7180000491023:1369010-1369061 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCTGTCAATTGCAAGGATCTGGTGCGAAAACCCACAGCCCTGATCTACGA | |
| droRho1 | scf7180000777097:264217-264273 - | CCC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAGGCTGTCAATTGCAAGGGTCTGGAGCGCAGGCCGACGGCCCTGATCTACGA | |
| droBia1 | scf7180000299226:162204-162271 - | CCC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TC-TAA-CC----------ACCT-------CCAGGCCGTCAACTGCAAGGGCCTGGTGCGGAAACCCACGGCCCTGATCTACGA | |
| droTak1 | scf7180000415381:847124-847143 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCACGGCCCTCATCTACGA | |
| droEug1 | scf7180000409533:369415-369469 + | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATAGGCTGTCAATTGCAAGGGCCTGGTACGGAAACCCACTGCTCTGATCTACGA | |
| dm3 | chrX:9526987-9527039 + | AGT--------------------------------------------------------------------------------------------------------------------------------------------------TTACCTAAAAA---------------------------------------------------------AT-------------ATTGTCTTTTCCCTCCGCTTTCCCCCCATTGTTTCGC | |
| droSim2 | x:14392757-14392808 + | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCTGTCAACAGCAAGAGTATGGTGCGGAAACCCACGGCACTGATCTACGA | |
| droSec2 | scaffold_56:116979-117030 + | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCTGTCAACAGCAAGAGTATGGTGCGGAAACCCACGGCACTGATCTACGA | |
| droYak3 | X:9429317-9429383 + | CCC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGTGA----------TCTT-------TCAGGCTGTCAATTGCAAGGGTCTGGTGCGGAAACCCACTGCCCTGATCTACGA | |
| droEre2 | scaffold_4690:11791992-11792065 - | TCT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TT--AA-TC----------ATCTCCGACCAAAAGGCTGTCAATTGCAAGGGTCTGGTGCGGAAACCCACGGCCCTGATCTACGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 09:04 PM