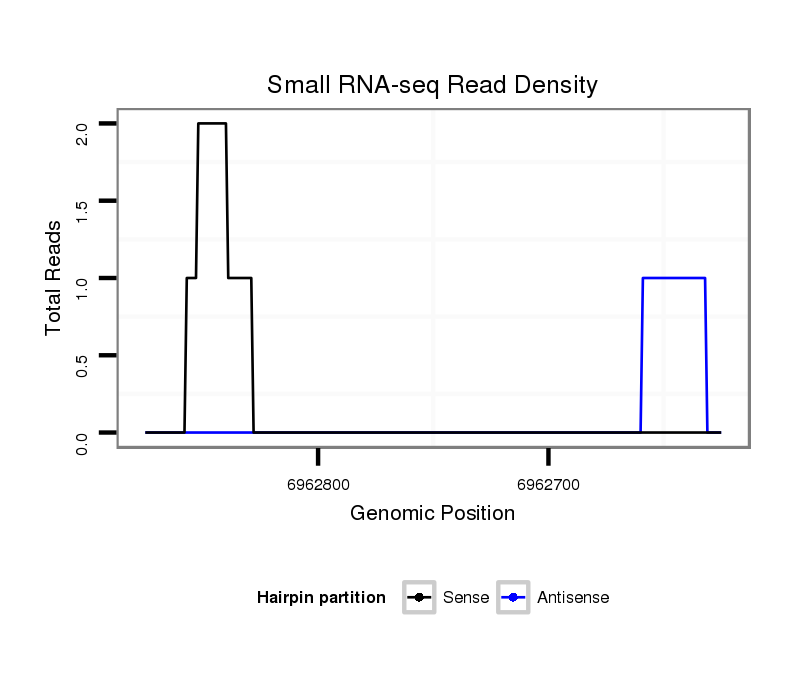
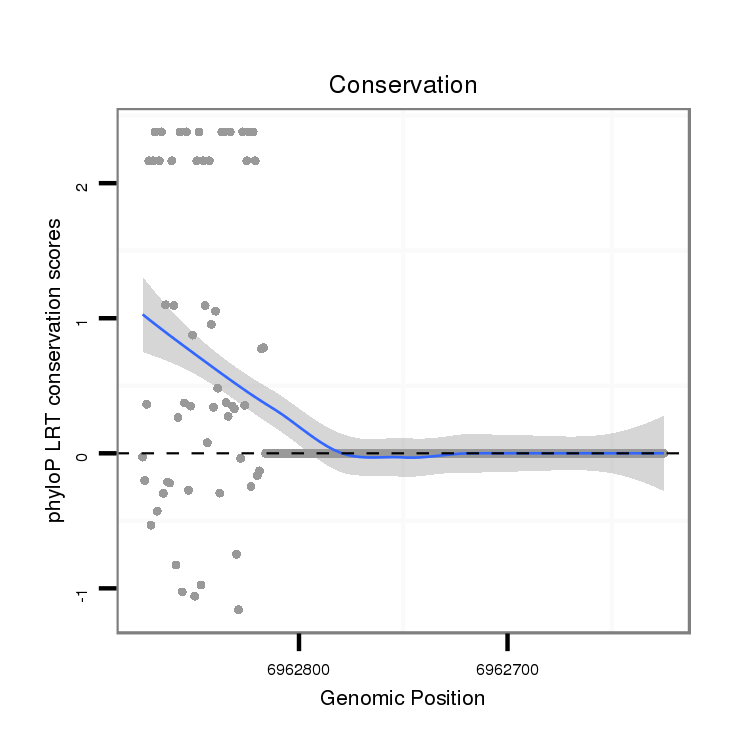
ID:dvi_9166 |
Coordinate:scaffold_12928:6962675-6962825 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_16912:1]; CDS [Dvir\GJ16408-cds]; intron [Dvir\GJ16408-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCGCCGAGGTCTGCGATTTGCATGAACTAGTTCGCAAATATCAAAGTAAGGTAAGAGGTGGAGTCATACGAGTATTCGACTAGCAGATACCTTAACTACAGCATTGAGCAGCTGTCGTTGTTATTCTATTACATCCCCTAGCTAGACTCTATGCGAATAGCTTTCCTGCCTCACTGGGCTGTCTGTGGTCCGATCTTTATGAAATGTTTTAGGGCACACGAGAGAGTGTTCATACCCGGTCGCAAGGCTCG **************************************************.((((((((..................(((((.(((((((((........(((..(((.(((((((((......((((..............))))......)))).)))))))).)))......))).)))))))))))..)))))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
M027 male body |
SRR060665 9_females_carcasses_total |
SRR060657 140_testes_total |
M028 head |
SRR060672 9x160_females_carcasses_total |
SRR060658 140_ovaries_total |
V116 male body |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................................................................................................................................ATAGCCGGTCGCAAGACT.. | 18 | 2 | 3 | 1.67 | 5 | 0 | 3 | 0 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................GAACTAGTTCGCAAATATCAAAGT............................................................................................................................................................................................................ | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................TGCATGAACTAGTTCGCA....................................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................CAGGTACCTTAACTACAGGTT................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................AATAGCCGGTCGCAAGACT.. | 19 | 3 | 11 | 0.45 | 5 | 0 | 2 | 0 | 0 | 0 | 1 | 2 | 0 | 0 |
| .......................................................................................................................................................................................................................................ATAGCCGGTCGCAAGACTG. | 19 | 3 | 12 | 0.33 | 4 | 0 | 3 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................CAAATAGCTTCCCTGCCTCA.............................................................................. | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................AGTGTTCATGCCAGGTCGTA....... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................TAGGGTAAGAGGAGGAGTGA......................................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................CGATACCTTTACTAAAGCA.................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CGCGGCTCCAGACGCTAAACGTACTTGATCAAGCGTTTATAGTTTCATTCCATTCTCCACCTCAGTATGCTCATAAGCTGATCGTCTATGGAATTGATGTCGTAACTCGTCGACAGCAACAATAAGATAATGTAGGGGATCGATCTGAGATACGCTTATCGAAAGGACGGAGTGACCCGACAGACACCAGGCTAGAAATACTTTACAAAATCCCGTGTGCTCTCTCACAAGTATGGGCCAGCGTTCCGAGC
**************************************************.((((((((..................(((((.(((((((((........(((..(((.(((((((((......((((..............))))......)))).)))))))).)))......))).)))))))))))..)))))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060683 160_testes_total |
SRR060660 Argentina_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060678 9x140_testes_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................TTACTTGATCAAGCGTTTATAG................................................................................................................................................................................................................. | 22 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................ATTTACTTGATCAAGCGTTTATAG................................................................................................................................................................................................................. | 24 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GTGCTCTCTCACAAGTATGGGCCAGCGT....... | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........ACGCTAATCGTACTTGTTC............................................................................................................................................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................................................................................................................................................CTCTGAGAAGTATGGGCCCG.......... | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................ATACTTTACAAAATCTCGAGT................................. | 21 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................................................................ACACTCACCAGGTTAGAAAT.................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:6962625-6962875 - | dvi_9166 | GCGCCGAGGTCTGCGATTTGCATGAACTAGTTCGCAAATATCAAAGTAAGGTAAGAGGTGGAGTCATACGAGTATTCGACTAGCAGATACCTTAACTACAGCATTGAGCAGCTGTCGTTGTTATTCTATTACATCCCCTAGCTAGACTCTATGCGAATAGCTTTCCTGCCTCACTGGGCTGTCTGTGGTCCGATCTTTATGAAATGTTTTAGGGCACACGAGAGAGTGTTCATACCCGGTCGCAAGGCTCG |
| droMoj3 | scaffold_6328:2633728-2633782 - | GCGCCGAGGTCTGTGATTTGCATGAGCTGGTTCGCAAATATCAAATCAAGGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_14853:3627835-3627889 + | GTTCTGAAGTTTGCGAGCTGAACGAACTGGTTCGCAAATATGAATCAAAGGTAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004963:2149050-2149104 + | GTGCCGAAGTGTACGGCCTGCATACCCTGGCCCATAAATATAAGGAGCATGTAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000395915:7010-7064 - | AGGCTGAAGTCTTTGATCTTCACACCCTGGTGCGACGCTACCAGGATTATGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453901:252519-252573 + | AGGCCGAGGTTTACGATCTTCACGCTCTCGTTCGCAAATATCAGGATCATGTAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491011:2161626-2161680 + | AGGCTGAGGTTTGCGACCTTAACACTCTAGTTCGCACTTACCAGGAAGATGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000779334:465234-465288 + | AGGCCGAGGTCTGCGACCTACACACTCTAGTTCGCAAATACCAGGATCATGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000301703:544109-544163 + | GAGCCGAAGTCTGCGAACTTCACACCCTGGTACGCAAGTACCAGGCTCACGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415394:1212521-1212575 + | GAGCCGAAGTCTGTGACCTCCACACTCTGGTTCGCAAGTACCAGGCTCATGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409076:212580-212634 - | GGTCAGAGGTCGGGGATTTGCACACGCTTGTTCGCAAATACCAGGCTCATGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:3915859-3915917 - | AGGCCGAAGTCTACGATCTACAGACGCTGGTTCGCAAGTACCAAGCCCATGTAAGTAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droSec2 | scaffold_4:2504562-2504620 + | AGGCTGAAGTCTACGATCTACAGACGCTGGTTCGCAAGTACCAAGCCCATGTAAGTAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droYak3 | X:4219905-4219959 + | GGGCCGAAGTCTGCGATCTGCACACGCTAGTTCGCAAGTACCAAGTTCATGTAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4690:1592467-1592521 - | GGGCCGAAGTCTGCGATCTGCATACGCTAGTTCGCAAGTACCAAGCCCATGTAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:16 PM