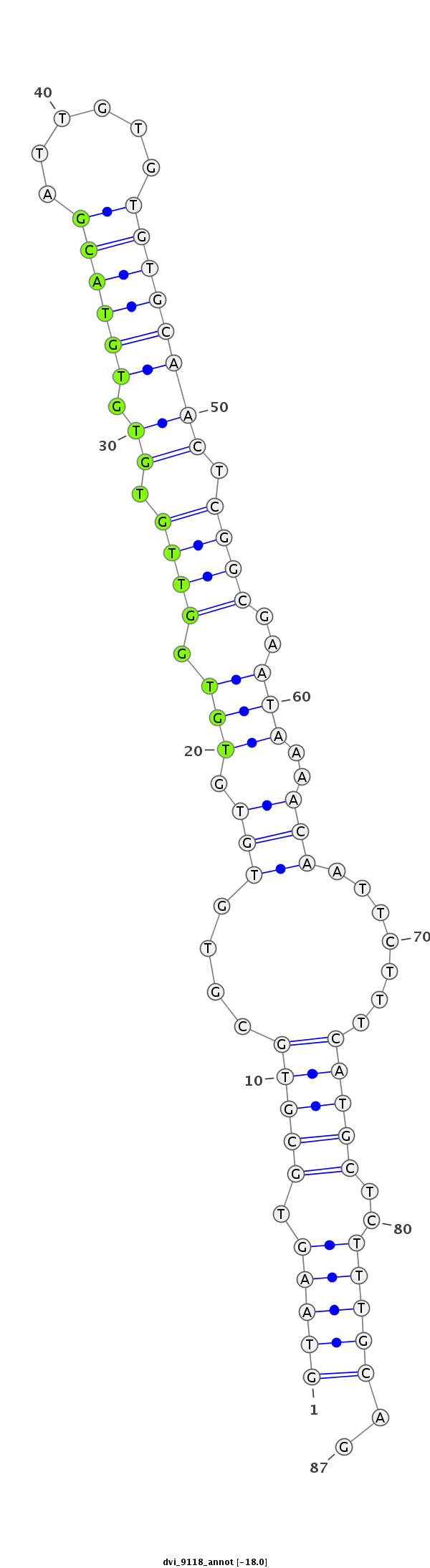
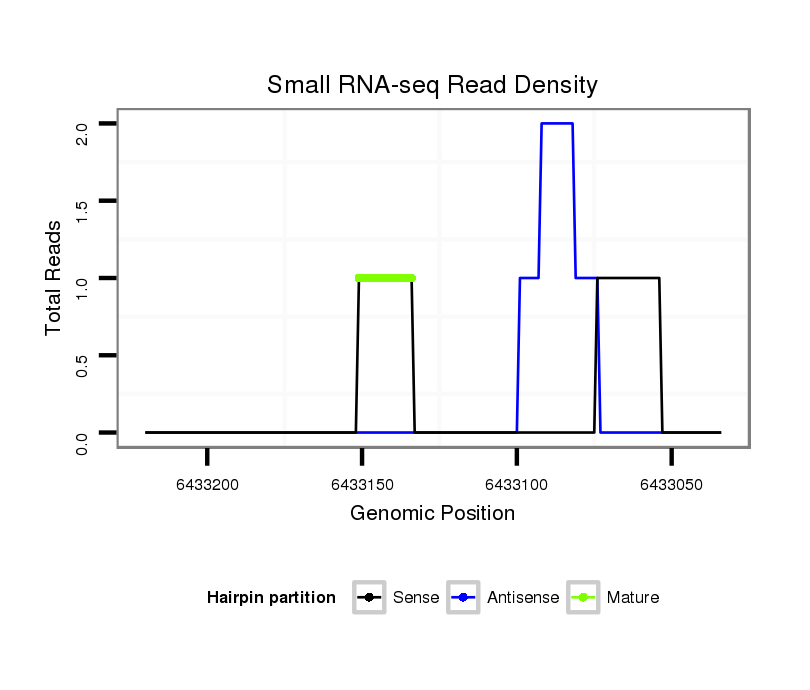
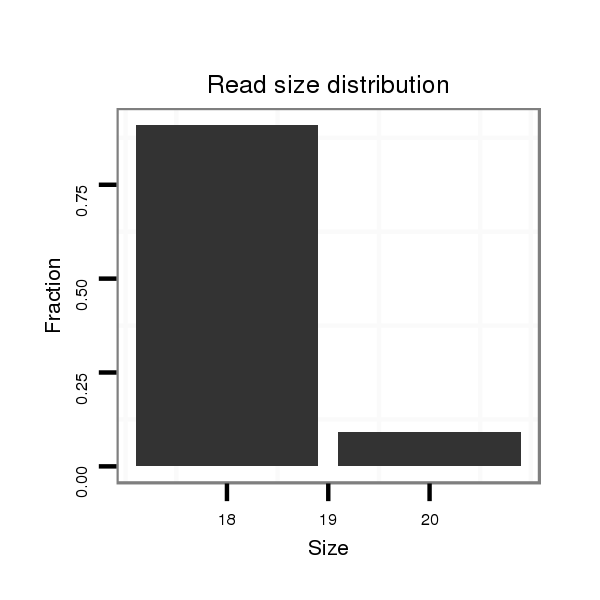
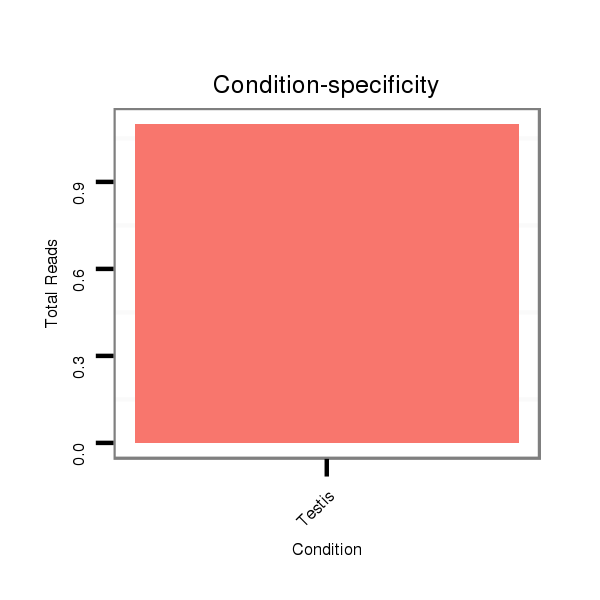
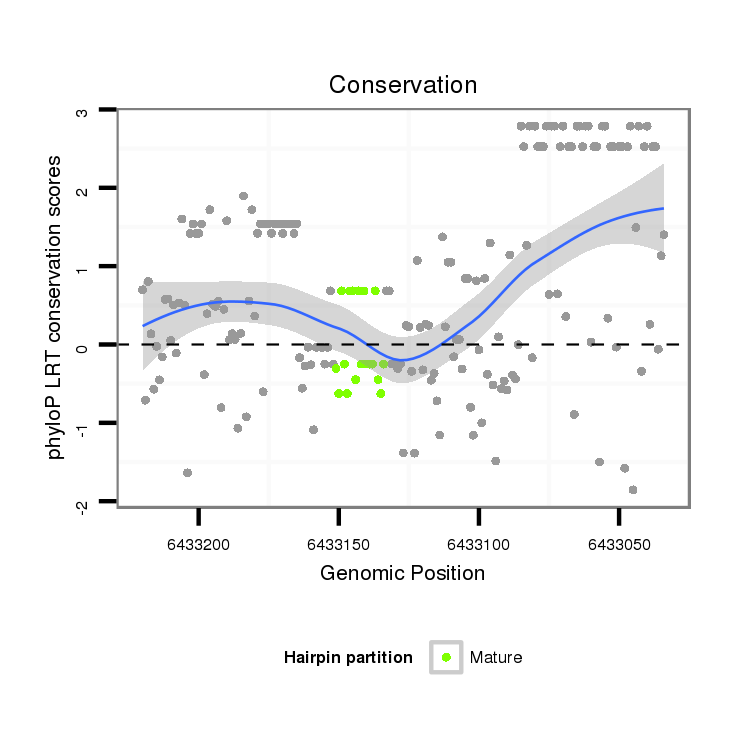
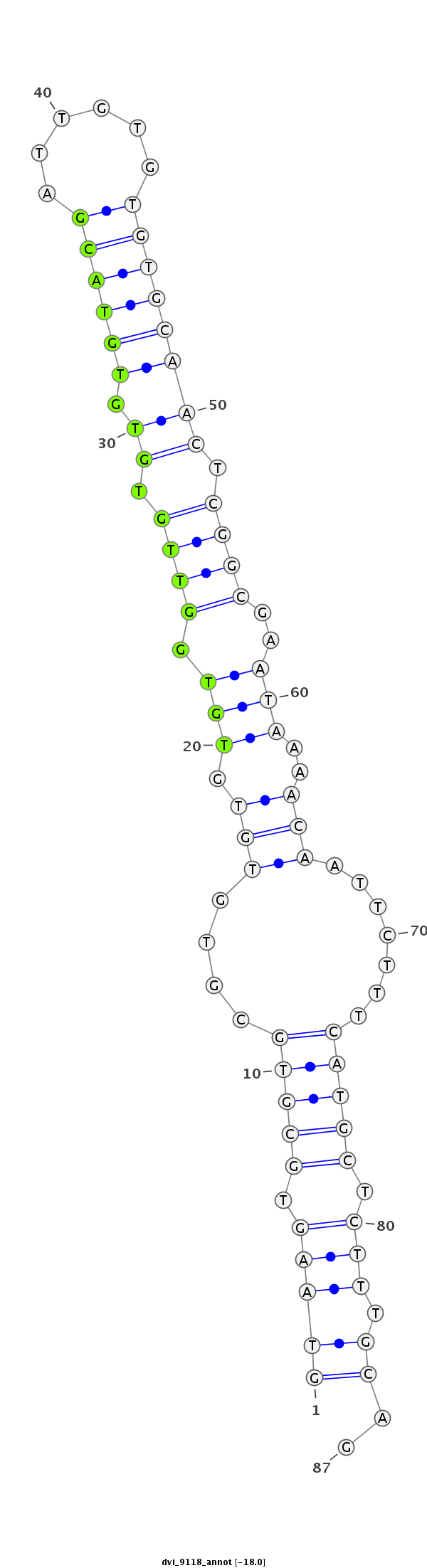
ID:dvi_9118 |
Coordinate:scaffold_12928:6433084-6433170 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -18.0 | -18.0 | -17.7 |
 |
 |
 |
CDS [Dvir\GJ16431-cds]; exon [dvir_GLEANR_16935:2]; exon [dvir_GLEANR_16935:1]; CDS [Dvir\GJ16431-cds]; intron [Dvir\GJ16431-in]
| Name | Class | Family | Strand |
| (CA)n | Simple_repeat | Simple_repeat | + |
| ##################################################---------------------------------------------------------------------------------------################################################## TGCGCGACAAGCCAGCTGAGGACATTGTGGCCAAGCTATACGATATGTTTGTAAGTGCGTGCGTGTGTGTGTGGTTGTGTGTGTACGATTGTGTGTGCAACTCGGCGAATAAAACAATTCTTTCATGCTCTTTGCAGGTATGGGATTTCGATCCCATGATACCCTTTCCGCCCGTCATTGAACCGGA **************************************************(((((.(((((....(((.(((.((((.((.((((((......)))))))).))))..)))..))).......)))))..)))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060657 140_testes_total |
SRR060655 9x160_testes_total |
SRR060666 160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060658 140_ovaries_total |
SRR060669 160x9_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060660 Argentina_ovaries_total |
SRR060664 9_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................GCTCCTTGTAGGTAGGGGA.......................................... | 19 | 3 | 7 | 9.86 | 69 | 27 | 5 | 0 | 7 | 5 | 5 | 5 | 3 | 1 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 0 | 0 |
| ..................................................................................................................................................TTCGATCCCATGATACCCTTT.................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................TGTGGTTGTGTGTGTACG.................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................TGCTCCTTGTAGGTAGGGGA.......................................... | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................CGGACATGGTGGACAAGCTAT.................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................GGATTTCGATCACCTGAT........................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................GCTCGTTGTAGGTAGGGGA.......................................... | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................GTGTGTGTGGTTGTGTGTGT....................................................................................................... | 20 | 0 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 |
| .............................................................................................................................................GGGATTTCGCGCCCAT.............................. | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................................................................................GCTCATTGTGGGTATGGGA.......................................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
ACGCGCTGTTCGGTCGACTCCTGTAACACCGGTTCGATATGCTATACAAACATTCACGCACGCACACACACACCAACACACACATGCTAACACACACGTTGAGCCGCTTATTTTGTTAAGAAAGTACGAGAAACGTCCATACCCTAAAGCTAGGGTACTATGGGAAAGGCGGGCAGTAACTTGGCCT
**************************************************(((((.(((((....(((.(((.((((.((.((((((......)))))))).))))..)))..))).......)))))..)))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060667 160_females_carcasses_total |
M047 female body |
M028 head |
SRR060685 9xArg_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................AAGTACGAGAAACGTCCA................................................ | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................AGAAACGTCCATACCCTAA........................................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................GTCCATACTGTAGAGCTAG.................................. | 19 | 3 | 8 | 0.25 | 2 | 0 | 0 | 2 | 0 | 0 |
| ................................................AACGTTCACGCACGCACACACA..................................................................................................................... | 22 | 1 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................................AAGCTACGGTATTATGGGTA..................... | 20 | 3 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................AAGCTTGGGTACAATGGC....................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| ....................................................TTCTCGCACGCACACACACAC.................................................................................................................. | 21 | 1 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:6433034-6433220 - | dvi_9118 | TGCGCGACAAGCCAGCTGAGGACATTGTGGCCAAGCTATACGATATGTTTGTAAGTGCGTGCGTGTG----------TGTGTGGTTGTGTGTGTACGATTGTG---------------------------------------------------TGTGCA---ACTCGGCGAA-------------------------------------TAAAACAATTCTTTCATGC-----TCTTTGCAGGTATGGGATTTCGATCCCATGATACCCTTTCCGCCCGTCATTGAACCGGA |
| droMoj3 | scaffold_6328:2108110-2108268 - | TGCGCGGCAAGCGAGCCGAGGATGTGGTAGCCACGCTCTACGACATGTTTGTAAGTGTGTGGGTGCG----------------------------------------------------------------------------------------------------------------------------CGTGTGTGTGGCGACAGTATATAA----------AGATCCCTCCACTTTCAGGTTTGGGACTTCGATCCGATGATACCGTTTCCGCCCGTCATTGAGCCGGA | |
| droGri2 | scaffold_14853:7429591-7429643 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTGTGGGATTTCGATCCCATGATACCATTTCCACCGGTCATTGAGCCGGA | |
| droWil2 | scf2_1100000004401:1244345-1244579 + | TACGTAGCATACGCGCGGAGGATATTGTTGCTGCACTCTATGATATGTTTGTAAGTATATCTACACAACCTAAAAAGTAACTACTTTTATACATCGAATCAAATCAGCCATTTAAAAAGACAATATTTTTTGGAATGTTATATTTTAAGCTTTAAATTGACAAATCTTTACAA--------------------TCT--------------------------------------GTGTCTTAGATTTGGGACTTTGATCCTATGATCCCTTTTCCGCCAGTGATAGAACCTCC | |
| dp5 | XL_group1e:11831902-11832060 + | TACGTCAGAAGCCAGCCGAGGATATTGTGGCTTCGCTTTACGACATGTTTGTAAGTATAT----------------------------------------------------------------------------------------------TGAAC--TGGATCCTTAATGAGTTGAGAGCTAATCTGGCGTT--------------------------------------GTTTGGCAGGTCTGGGACTTTGATCCCATGATACCATTTCCTCCGGTGATTGAGCCCGA | |
| droPer2 | scaffold_14:1782852-1783010 - | TACGTCAGAAGCCAGCCGAGGATATTGTGGCTTCACTTTACGACATGTTTGTAAGTACAT----------------------------------------------------------------------------------------------TGAAC--TGGATCCTTAATGAGTTGAGAGCTAATCTGGCGTT--------------------------------------GTTTGGCAGGTCTGGGACTTTGATCCCATGATACCATTTCCCCCGGTGATTGAGCCTGA | |
| droAna3 | scaffold_6016:3300-3456 - | TGCGGGGGAAGCCAGCAGAGGATATTGTCGCCTCCTTGTACGACATGTTTGTAAGTTCTG----------------------------------------------------------------------------------------------CGTGGAAGGATTCTAGGAA-------------------------------------TATGTGAATACCGTAAGCT-----TCTTCTTAGGTCTGGGACTTTGATCCGATGATCCCCTTCCCGCCAGTCATCGAGCCGGA | |
| droBip1 | scf7180000396033:11160-11314 - | TGCGGGAGAAGCCAGCGGAGGACATTGTCGCCTCCTTGTACGATATGTTTGTAAGTTCAA----------------------------------------------------------------------------------------------GGTGA--AGATTCTAGAAA-------------------------------------TATGTGAAACCCATAAGAT-----TCGTCTTAGGTCTGGGACTTCGATCCGATGATTCCCTTCCCGCCGGTTATTGAGCCGGA | |
| droKik1 | scf7180000299277:241610-241686 + | AGCTC-------------------TTACCTCTTCTTTCAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGGCAGGTCTGGGACTTTGATCCGATGATACCCTTCCCGCCGGTCATTGAGCCTGA | |
| droFic1 | scf7180000453901:602155-602241 + | AACTAGGCTAATCAGTC--------------TTACCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAACT-----TATTGACAGGTTTGGGACTTTGACCCAATGATACCCTTTCCGCCAGTCATCGAACCGGA | |
| droEle1 | scf7180000491006:2713529-2713594 - | TTTAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TC--------------------------------------TTTTTGAAGGTCTGGGACTTTGACCCGATGATACCTTTTCCCCCGGTGATCGAACCGGA | |
| droRho1 | scf7180000779334:857415-857476 + | TGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTTAAGGTCTGGGACTTTGATCCGATGATACCTTTTCCGCCGGTGATCGAACCGGA | |
| droBia1 | scf7180000302421:2651950-2652006 - | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGCAGGTCTGGGACTTTGACCCAATGATACCTTTTCCGCCAGTGATTGAACCGGA | |
| droTak1 | scf7180000414393:264213-264269 - | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGCAGGTCTGGGACTTTGATCCCATGATACCATTCCCGCCAGTCATCGAACCGGA | |
| droEug1 | scf7180000409553:112884-112938 - | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTAGGTCTGGGACTTTGATCCGATGATCCCTTTTCCGCCAGTCATTGAACCGGA | |
| dm3 | chrX:3864061-3864126 - | TTCTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TT--------------------------------------GTATTGCAGGTCTGGGACTTTGATCCGATGATACCATTTCCGCCCGTGGTCGAACCGGA | |
| droSim2 | x:3559537-3559592 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAGGTCTGGGACTTTGATCCGATGATACCATTTCCGCCCGTGATCGAACCGGA | |
| droSec2 | scaffold_4:2853713-2853768 + | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAGGTCTGGGACTTTGATCCGATGATACCATTTCCGCCCGTGATCGAACCGGA | |
| droYak3 | X:4559218-4559273 + | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAGGTCTGGGACTTTGATCCGATGATACCATTTCCGCCTGTGATCGAACCGGA | |
| droEre2 | scaffold_4690:1239162-1239217 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAGGTCTGGGACTTTGATCCGATGATACCATTTCCGCCCGTAATCGAACCGGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 07:59 PM