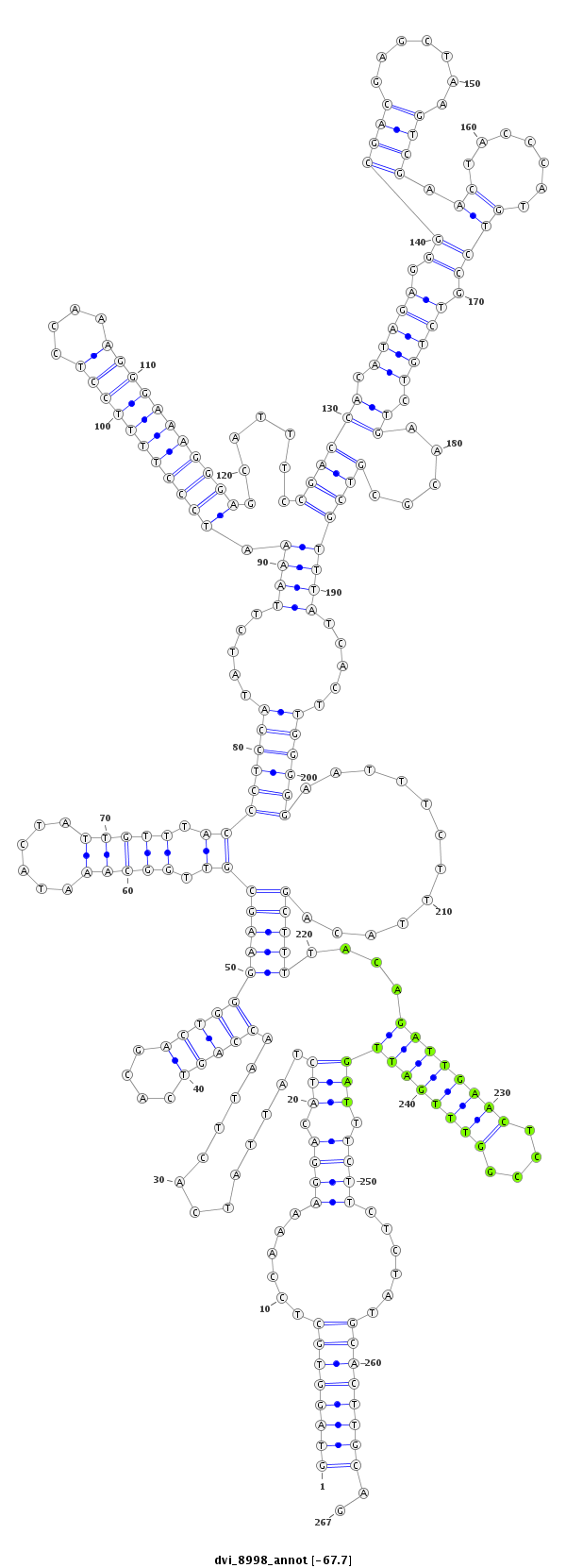
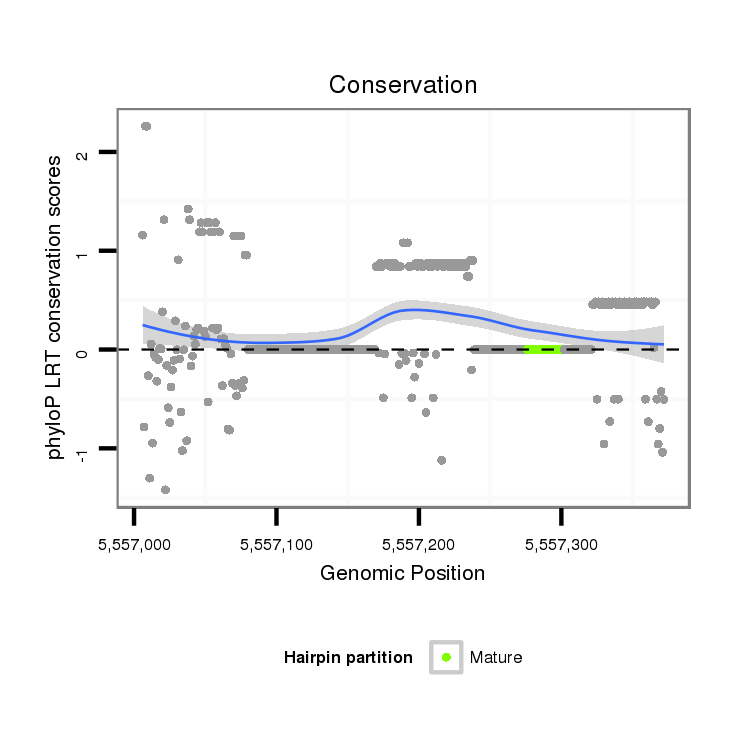
ID:dvi_8998 |
Coordinate:scaffold_12928:5557056-5557322 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -67.6 | -67.2 | -67.2 |
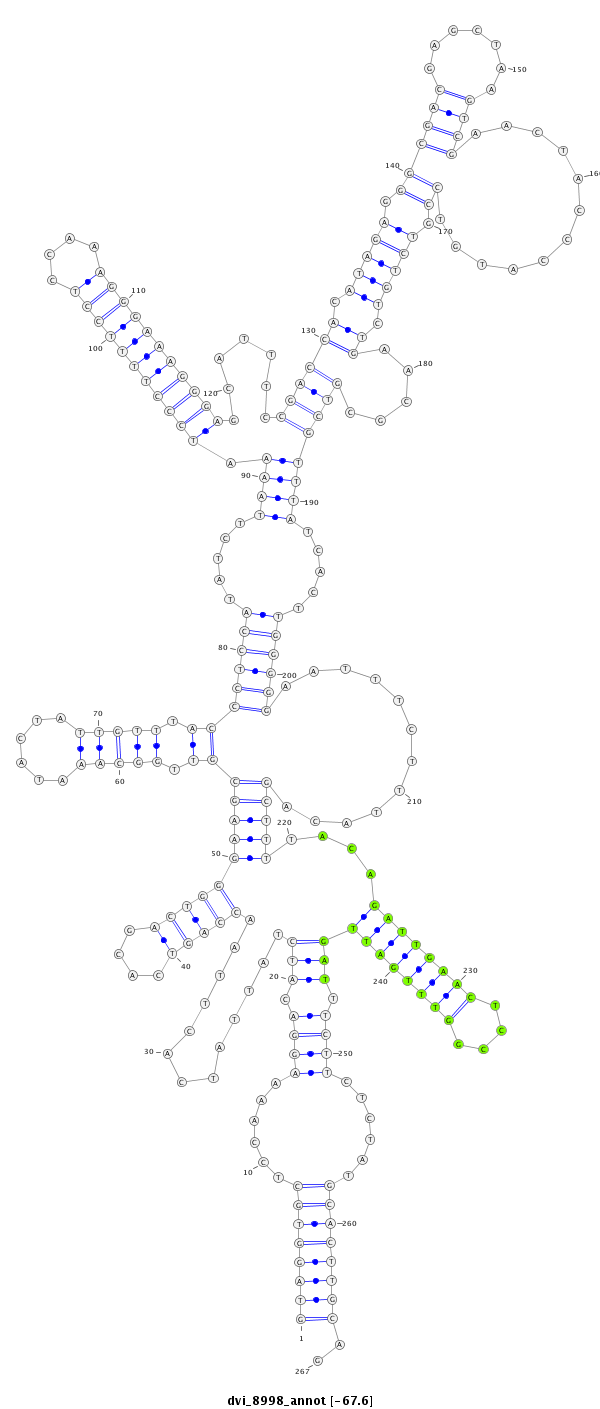 |
 |
 |
CDS [Dvir\GJ16964-cds]; CDS [Dvir\GJ16964-cds]; exon [dvir_GLEANR_17473:2]; exon [dvir_GLEANR_17473:3]; intron [Dvir\GJ16964-in]
| Name | Class | Family | Strand |
| Helitron-1N1_DVir | RC | Helitron | + |
| Homo6 | DNA | hAT-Pegasus | + |
| ##################################################---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## ACTTTATAACACTACCCAGCGAAAGCTACGGCAGTGACAGCAGCCTAAGCGTAGGTGCTCCAAAAGGACATCTATTATCACTTAACCAGTCACGACTGGGAAGCGTTGGCAAATACTATTGTTTACCCTCCATATCTTAAAATCCCTTTTCCTCCAAAGGGAAAGGGAGCATTTCCGACCACATAGAGGGCGACGAGCTAAGTCGAACTACCCATGTCCGTCTGTCTGAACGCGTCGTTTATCACTTGGGGGAATTTCTTTACAGCTTTTACAGATTGAACTCCGGTTTGATTGATTTCTTCTCTATGCACTTGCAGGATTGGACCGATGAGACTAGCAGCGAGGAGAGCTCACAATACGAAGAGCC **************************************************((((((((......((((.(((.............(((((....)))))(((((((.(((((......))))).))((((((.....((((.(((((((((((....))))))))))).......((((((.(((((.((((((.......)))).((.......)))).))))).)).....)))))))).....))))))............)))))....((((((((....))))))))))).))))......))))))))..************************************************** |
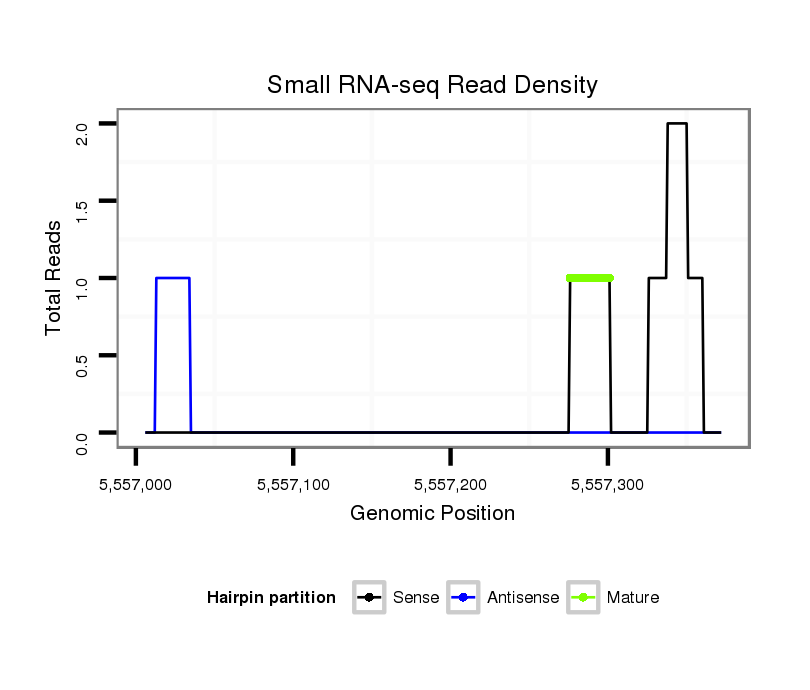
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060674 9x140_ovaries_total |
SRR060680 9xArg_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
V116 male body |
SRR060668 160x9_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060689 160x9_testes_total |
SRR060671 9x160_males_carcasses_total |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................CATTCGCGACTGGGAAGC....................................................................................................................................................................................................................................................................... | 18 | 2 | 4 | 1.25 | 5 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................................................................................................................................................................................................TGGACCGATGAGACTAGCAGCGAGG...................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................................................................................................................................ACTAGCAGCGAGGAGAGCTCACA............ | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................................................................................................................GAAGAGAGTAGCAGCGAGGA..................... | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................................................................................ACAGATTGAACTCCGGTTTGATTGAT....................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................AATCATTCGCGACTGGGAAGC....................................................................................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................GCAGGCTAAGCGTAGGTCC..................................................................................................................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................................................................ATGAACTACGGTTGGATTGA........................................................................ | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................................................................................TGAGATTAGCAGCGATGA..................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................................................................................................................................................................GAACGAACTCTGGTTTGATT.......................................................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................................................................AACGCCGTTCTGATTGATTT..................................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................................................................................................................................................................................................................................................................AGAAGAGAGTAGCAGCGAGG...................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................AGCGAGCATTTCCGGCAAC......................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
TGAAATATTGTGATGGGTCGCTTTCGATGCCGTCACTGTCGTCGGATTCGCATCCACGAGGTTTTCCTGTAGATAATAGTGAATTGGTCAGTGCTGACCCTTCGCAACCGTTTATGATAACAAATGGGAGGTATAGAATTTTAGGGAAAAGGAGGTTTCCCTTTCCCTCGTAAAGGCTGGTGTATCTCCCGCTGCTCGATTCAGCTTGATGGGTACAGGCAGACAGACTTGCGCAGCAAATAGTGAACCCCCTTAAAGAAATGTCGAAAATGTCTAACTTGAGGCCAAACTAACTAAAGAAGAGATACGTGAACGTCCTAACCTGGCTACTCTGATCGTCGCTCCTCTCGAGTGTTATGCTTCTCGG
**************************************************((((((((......((((.(((.............(((((....)))))(((((((.(((((......))))).))((((((.....((((.(((((((((((....))))))))))).......((((((.(((((.((((((.......)))).((.......)))).))))).)).....)))))))).....))))))............)))))....((((((((....))))))))))).))))......))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060683 160_testes_total |
SRR060678 9x140_testes_total |
V053 head |
SRR060681 Argx9_testes_total |
M047 female body |
SRR060677 Argx9_ovaries_total |
SRR060665 9_females_carcasses_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......TTGTGATGGGTCGCTTTCGATG.................................................................................................................................................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................ACCACTGGGAGATATAGAAT.................................................................................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............TAGGTCGGTTTCGATGCCGTG............................................................................................................................................................................................................................................................................................................................................. | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................GGTACAGGGAGACATACTTGCG...................................................................................................................................... | 22 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................................................................................................................GTCCAACCCTGGCAACTCT.................................. | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................GGTACAGGCAGACATATTTGC....................................................................................................................................... | 21 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................................................................................................................................................................................................................TGCTAACCTGCCTACGCT.................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................................................................................................................................................................................................................................................CGAACCCACTTAAAGAAGTG........................................................................................................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:5557006-5557372 + | dvi_8998 | ACTTTATAACACTACCCAGCGAAAGCTACGGCAGTGACAGCAGCCTAAGCGTAGGTGCTCCAAAAGG---ACATCTATTATCACTTAACCAGTCACGACTGGGAAGCGTTGGCAAATACTATTGTTTACCCTCCATATCTTAAAATCCCTTTTCCTCCAAAGGGAAAGGGAGCATTTCCGACCACATAGA-------------------GGGCGACGAGCTAAGTCGAACTACCCATGTCCGTCTGTCTGAACGCGTCGTTTATCACTTGGGGGAATTTCTTTACAGCTTTTACAGATTGAACTCCGGTTTGATTGATTTCTTCTCTATGCACTTGCAGGATTGGACCGATGAGACTAGCAGCGAGGAGAGCTCACAATACGAAGAGCC |
| droMoj3 | scaffold_6359:4078954-4079016 + | TGTTCCC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTAA----------------------------------------------------------------------------------------------------------------------------------------------------GGACTGGAGCGAGGAAACCAGCAGCGAGGAGAGCTCATACTACGAGCCAGA | |
| droGri2 | scaffold_15203:1444966-1445039 + | ATTTCGATAAAGAATCACTACTAAGCTCGGATAGCAACAGCAGCTTGAGCGTAGGTTCTTTTATATA---ACAATTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dp5 | XL_group1a:933983-933986 - | ATTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_26:1047124-1047127 - | ATTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12929:2955210-2955224 - | ATTTTGTTCCACCGC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000391693:31213-31216 - | ATTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302401:100429-100432 - | ATTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454038:608934-608982 - | ATTTCCTGGAACCACCAGACATGG------ATAGTTCGGACAGCTTAAGTGTGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491023:1221496-1221499 + | ACTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000777097:117022-117098 + | ACTTCCTCGATCCACCGGGGGATGACGATAACAGCTCGGACAGTTTGAGTGTGAGCATTTTAGGAACCCTATAATTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBia1 | scf7180000302193:3657044-3657109 + | AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------GGAAGTGTTTCCGACCCCATAAA-------------------GGATCACTAGCCAAGTCAATCTACCCATGTCCGTCTGTCTG------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415879:66472-66561 - | AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------GGAAGCGTTTCCGACCCCATAAAGTATATATATTCTTGATCAGGGTCACTAGCCGAGTCGATCTAGCCATGTCCGTCTGTCTGAACGC-------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409533:514878-514907 - | ACTTTTTGGACTCACCAGAGGATG------AAAGCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:14527832-14527906 - | ATTTCCTGGATCCGCCTGAGGATG------ACAGCTCGGACAGCTTAAGTGTGAGCATCTAGAGAACTCCAAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAC------------------------------------------------------------------------------------------------------------------------------A---TATC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
Generated: 05/17/2015 at 05:27 AM