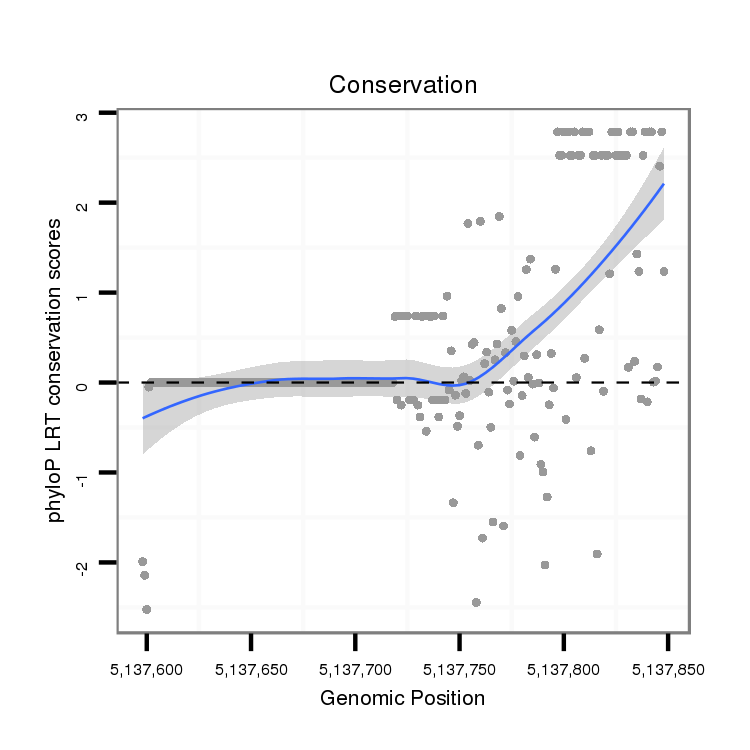
ID:dvi_8972 |
Coordinate:scaffold_12928:5137648-5137798 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ16953-cds]; exon [dvir_GLEANR_17461:4]; Antisense to exon [dvir_GLEANR_16998:1]; Antisense to CDS [Dvir\GJ16495-cds]; intron [Dvir\GJ16953-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GCTTCTTCACACAGCATATATACCCTTTTGGCACACTCTGTACAGGGTATATTAATGTGAATAGAAAAGTATAAGCCTTTGCCTTTATTTATAACGTAATGCACAGGCACACCCTGTGTACGGTTATACCCTAGCTCTGCAGCGAGAATGTGCTCCTATTAACGCTTCCTCTTCCTCTTCTTCCTCTTGTTGTTGTTGTAGGTCTGGTACCAGAATCGACGCGCCAAGTGGCGCAAGACGGAAAAATGCTG **************************************************.....((((((((((...(((........))).))))))))))......((((((((.....)))))))).(((((.....)))))(((((((((.((((.....................................)))).)))))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060668 160x9_males_carcasses_total |
M061 embryo |
SRR060667 160_females_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060673 9_ovaries_total |
SRR060659 Argentina_testes_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................CTTCCTCTTCCTCTTCTTCCTCTGT.............................................................. | 25 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................AATCGACGCGCCAAGTGGC................... | 19 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................TTCCGCTAGCTCTTCTTCCTCTT............................................................... | 23 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................CTCTTCCTCTTCTTCCTCTGTT............................................................. | 22 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................TGAATAGAAAGGTATTAGC............................................................................................................................................................................... | 19 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................................TTGTGGGAGGACTGGTACC........................................ | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................................................................TCTTCCTATTGTTGTTGTTG..................................................... | 20 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| --------------------------------------------------------------------------------------------------------------------------------------------------------------############################################################################################# CGAAGAAGTGTGTCGTATATATGGGAAAACCGTGTGAGACATGTCCCATATAATTACACTTATCTTTTCATATTCGGAAACGGAAATAAATATTGCATTACGTGTCCGTGTGGGACACATGCCAATATGGGATCGAGACGTCGCTCTTACACGAGGATAATTGCGAAGGAGAAGGAGAAGAAGGAGAACAACAACAACATCCAGACCATGGTCTTAGCTGCGCGGTTCACCGCGTTCTGCCTTTTTACGAC **************************************************.....((((((((((...(((........))).))))))))))......((((((((.....)))))))).(((((.....)))))(((((((((.((((.....................................)))).)))))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060658 140_ovaries_total |
SRR060673 9_ovaries_total |
SRR060679 140x9_testes_total |
SRR060677 Argx9_ovaries_total |
V053 head |
SRR060687 9_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
V116 male body |
M047 female body |
M061 embryo |
M027 male body |
SRR060657 140_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
SRR060674 9x140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................AGGAGAAGGAGAAGAAGGAAAA............................................................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................GTTCGTGTGGGACACAGGCC................................................................................................................................ | 20 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................AAAAAAATAGTGCATTACGT.................................................................................................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................AGGAAAAGAAGGAGACCAAAAACA....................................................... | 24 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................CGTGGTCTTAGCTGCAGGGT......................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................AAGAAGGAGAACAACAACAAC..................................................... | 21 | 0 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................AGAAGGAGAACAACAACAAC..................................................... | 20 | 0 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................GGTTCAACGCGGTCTACCTT........ | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................TAGGAGAAGGAGAAGAAGTAGA................................................................ | 22 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................AGAAGGAGAAGAAGGAGT................................................................ | 18 | 1 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................................................AGAAGGAGAAGTAGGAGA................................................................ | 18 | 1 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................GGGGACGGAGAAGAAGGAG................................................................. | 19 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................CTGGCAATTTGGGATCGA................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................AGAAGAAGAAGAACAACAACAACAT................................................... | 25 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................................................................................................................GAGAAGGAGAAGGAGAAGA...................................................................... | 19 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................AGAAGAAGGAGAAGAAGGAG................................................................. | 20 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................................................TCAAGGAGAAGGAGAAGAAG.................................................................... | 20 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:5137598-5137848 + | dvi_8972 | GCTTCTTCACACAGCATATATACCCTTTTGGCACACTCTGTACAGGGTATATTAATGTGAATAGAAAAGTATAAGCCTTTGCCTTTATTTATAACGTAATGCACAGGCACACCCTGTGTACGGTTATACCCTAGCTCTGCAGCGAGAATGTGCT-CCTATTAA---CGCTTCC--TCTT---CCTCTTCTTCCTCTTGTTGTT-GTTGTAGGTCTGGTACCAGAATCGACGCGCCAAGTGGCGCAAGACGGAAAAATGCTG |
| droMoj3 | scaffold_6359:3641598-3641701 + | GAG-----------------------------------------------------------------------------------------------------------------------------------------------AATCCGCTTGCTAATAAATTCTCTT-------T---GCTCTTCTCTC---TCTGGTT-ATTGCAGGTGTGGTACCAGAACCGTCGGGCCAAGTGGCGCAAGACCGAGAAGTGCTG | |
| droGri2 | scaffold_15252:6699316-6699401 + | CTTT-----------------------------------------------------------------------------------------------------------------------------------------------------C-ATTC---T---CTCTCTC--TCTTCCTGTCTT-------------C-C-TGTGCAGGTCTGGTTCCAGAATCGACGCGCCAAGTGGCGCAAGCAGGAGAAGG--TT | |
| droWil2 | scf2_1100000004909:11079970-11080062 + | AACA------------------------------------------------------------------------------------------------------------------------------------------------TTTTTT-ATTCATTT---CCATGT-T-TTTT---CTTTT-------------G-A-ATTGCAGGTCTGGTACCAGAATCGTCGGGCAAAGTGGCGTAAAACCGAAAAATGCTG | |
| dp5 | XL_group1a:8116949-8117043 + | GAA-------------------------------------------------------------------------------------------------------------------------------------------------TGTACC-ATTAACAG---CGCTCTG--TTTTGC---T-----------TGATATT-CTTCCAGGTCTGGTTCCAGAATCGCAGGGCCAAGTGGCGGAAGACCGAGAAGGTCTG | |
| droPer2 | scaffold_15:477701-477795 + | GAA-------------------------------------------------------------------------------------------------------------------------------------------------TGTACC-ATTAACAG---CGCTCTG--TTTTGC---T-----------TGATATT-CTTCCAGGTCTGGTTCCAGAATCGCAGGGCCAAGTGGCGGAAGACCGAGAAGGTCTG | |
| droAna3 | scaffold_13117:805924-805992 - | TCCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------C-------------------TGTTATC-CTTCCAGGTCTGGTACCAGAACCGCAGGGCCAAGTGGCGCAAGACGGAAAAGTGCTG | |
| droBip1 | scf7180000396412:717658-717732 - | CCTC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCCTCTCGTTATC-CTTTCAGGTCTGGTACCAGAACCGCAGGGCCAAGTGGCGCAAGACGGAAAAGTGCTG | |
| droKik1 | scf7180000302469:638410-638482 - | TCTT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------T---------------CTGTTGTGACC-TTTGCAGGTTTGGTACCAAAACCGACGGGCCAAGTGGCGCAAGACGGAAAAGTGCTG | |
| droFic1 | scf7180000454072:2605999-2606072 - | CTCC--------------------------------------------------------------------------------------------------------------------------------------------------------------------CTT--CCCT-------------------TTCCG-A-TCCGCAGGTCTGGTACCAGAACCGCCGGGCCAAGTGGCGCAAGACGGAGAAGTGCTG | |
| droEle1 | scf7180000491001:955544-955616 - | TCGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------T--TTT-----------------TCGTTGAT-CCTGCAGGTGTGGTACCAGAATCGACGGGCCAAGTGGCGGAAAACGGAGAAGTGCTG | |
| droRho1 | scf7180000779976:777967-778044 - | TTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCTT--CTTTT----T-------------TCTTGAT-CCTGCAGGTCTGGTACCAGAACCGTCGGGCCAAGTGGCGCAAAACGGAGAAGTGCTG | |
| droBia1 | scf7180000302069:320572-320639 - | CCCT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTTTTT-CCTGCAGGTGTGGTACCAGAACCGCCGGGCCAAGTGGCGCAAGACCGAGAAGTGCTG | |
| droTak1 | scf7180000415289:643855-643975 - | TAT----------------------------------------------------------------------------------------------------------------------GATAATATTTTTTCTGTGTAAAAAAACCTCGTT-ACTAATTT---CGCTTA-C-CTTT---GTTTT-------------GCTCCTTGCAGGTGTGGTACCAGAATCGCCGGGCCAAGTGGCGCAAAACGGAGAAGTGCTG | |
| droEug1 | scf7180000409095:541422-541501 + | TCCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-T---CTTCTA----TTCTCTTTGAC-CCTGCAGGTTTGGTACCAGAATCGACGGGCCAAGTGGCGCAAGACGGAGAAGTGCTG | |
| dm3 | chrX:5425428-5425518 - | AAT--------------------------------------------------------------------------------------------------------------------------------------------------GTAAT-ACTAATAG---CATATCG--TTTACC---T--------------TCTG-TTTGCAGGTGTGGTTCCAAAACCGACGCGCCAAGTGGCGCAAGACGGAGAAAGTCTG | |
| droSim2 | x:5139693-5139771 + | ATT--------------------------------------------------------------------------------------------------------------------------------------------------------------GG---CGCTT--CCTT-T---GTCCC-------------A-T-TCCGCAGGTGTGGTACCAGAACCGCCGGGCCAAGTGGCGCAAGACGGAGAAGTGCTG | |
| droSec2 | scaffold_4:1253131-1253209 - | ATT--------------------------------------------------------------------------------------------------------------------------------------------------------------TG---CGCTT--CCTT-T---GTCCC-------------A-T-TCCGCAGGTGTGGTACCAGAACCGCCGGGCCAAGTGGCGCAAGACGGAGAAGTGCTG | |
| droYak3 | X:2963913-2963993 - | ATT--------------------------------------------------------------------------------------------------------------------------------------------------------------GG---CGCATCC--TTTT---GTCCC-------------GCA-TCCGCAGGTGTGGTACCAGAATCGACGGGCCAAGTGGCGCAAGACGGAAAAGTGCTG | |
| droEre2 | scaffold_4690:2776271-2776332 - | TT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCC-CTTTCAGGTGTGGTTCCAAAATCGACGCGCCAAGTGGCGCAAGACGGAGAAAGTCTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 05:12 AM