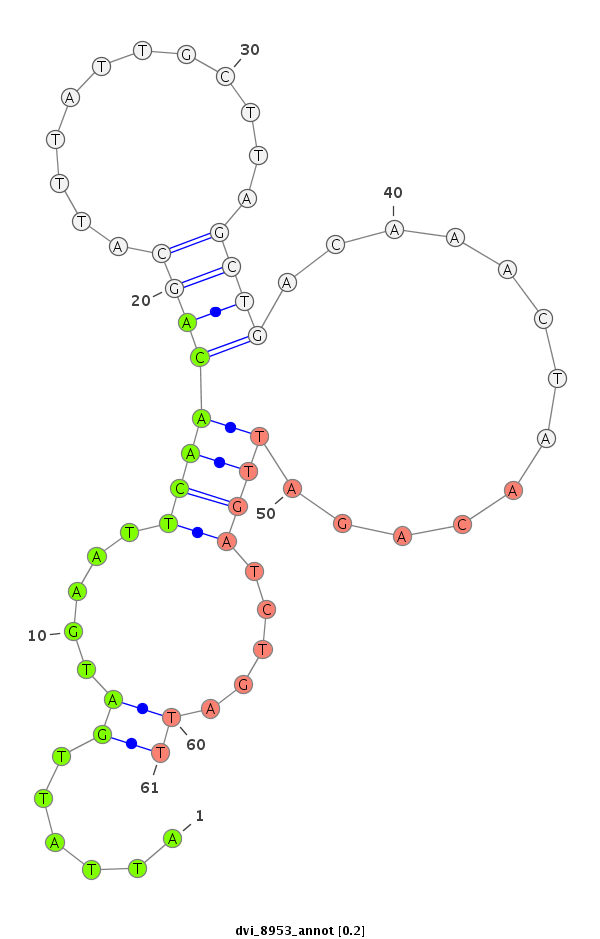
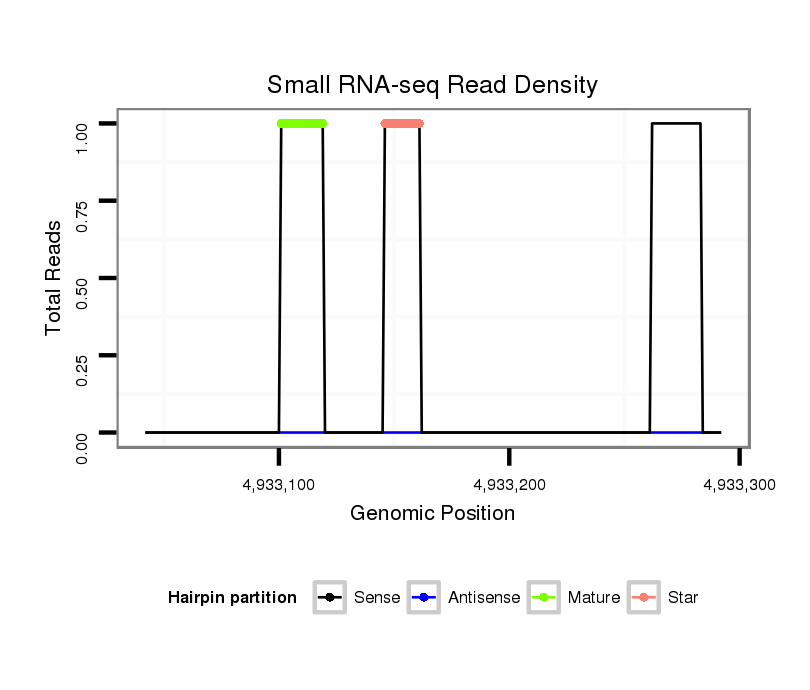
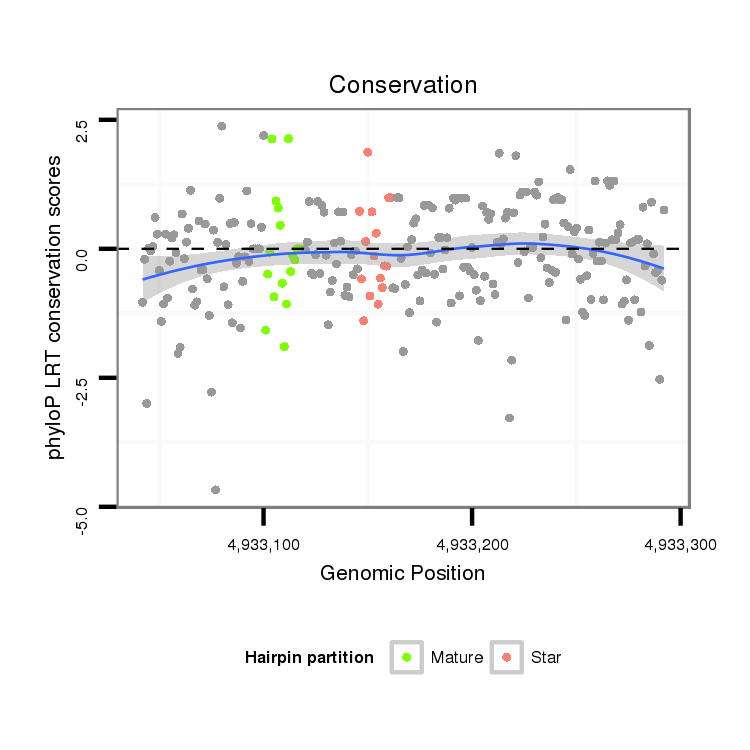
ID:dvi_8953 |
Coordinate:scaffold_12928:4933092-4933242 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -7.7 | -7.1 | -6.9 | -6.7 |
 |
 |
 |
 |
CDS [Dvir\GJ16943-cds]; exon [dvir_GLEANR_17448:3]; intron [Dvir\GJ16943-in]
| Name | Class | Family | Strand |
| (ACTG)n | Simple_repeat | Simple_repeat | + |
| (ATTG)n | Simple_repeat | Simple_repeat | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CTGATTGATCGACTGATCAACTGATTGATAACAGTTTGAATAACTGATTGATCGATAGAATTATTGATGAATTCAACAGCATTTATTGCTTAGCTGACAAACTAACAGATTGATCTGATTGATAGCACTTGATTGATTTCTGGATTGACTGAGTAAATATGTAAGTGATTGATTGATTGACCTAATTTACTGGCTTTGCAGGTGGCCACCAAGATCGAGCAGCTGCTGGAGCGCACACCATACGAGTATAC ***********************************************************......((.....((((((((............)))).............)))).....))*********************************************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060663 160_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060674 9x140_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060670 9_testes_total |
SRR060657 140_testes_total |
M047 female body |
SRR060681 Argx9_testes_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................TCTGATTGATAACATTTGAGTGA................................................................................................................... | 23 | 3 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................CTGATTGATAACATTTGAGTGA................................................................................................................... | 22 | 3 | 3 | 1.67 | 5 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................ATTATTGATGAATTCAACA............................................................................................................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................AGCTGCTGGAGCGCACACCATA......... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................ACAGATTGATCTGATT................................................................................................................................... | 16 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GATTGAGAGCTCTTTATTGATTT................................................................................................................ | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TCTGATTGATAACATTTGAGTG.................................................................................................................... | 22 | 3 | 2 | 1.00 | 2 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GATTGAGAGCTCTTTATTGAT.................................................................................................................. | 21 | 3 | 5 | 0.80 | 4 | 0 | 1 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................CTGATTGATAACATTTGAGT..................................................................................................................... | 20 | 3 | 20 | 0.50 | 10 | 0 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................CTGAGTAAATATGTAAGTA.................................................................................... | 19 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................CTGATTGATAACATTTGAGTG.................................................................................................................... | 21 | 3 | 6 | 0.33 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................GACTGAATGACCTCATTTAC............................................................. | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TCTGATTGATAACATTTGA....................................................................................................................... | 19 | 2 | 9 | 0.22 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GATTGAGAGCACTTTCTTGATT................................................................................................................. | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................AGCAGCTGCTGGAGGGCA................ | 18 | 1 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GATTGATAGCTCTTTCTTGATTT................................................................................................................ | 23 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................ATCAGATTGATAGCCCTT......................................................................................................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................CGATTGATAACAGGTTGA.................................................................................................................................................................................................................... | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................AGCTGGTGGAGGGGACACCA........... | 20 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 |
| .....................CGATTGATAACAGGTTGAG................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................................TCTGATTGATAACATTTGAGT..................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................CAGCTGGTGGAGGGGACACCA........... | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GGTGGTCTACAAGATCGAGC............................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................GATGGATAACAGTTTGAT................................................................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................TGATGGATAACAGGTTGAG................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GACTAACTAGCTGACTAGTTGACTAACTATTGTCAAACTTATTGACTAACTAGCTATCTTAATAACTACTTAAGTTGTCGTAAATAACGAATCGACTGTTTGATTGTCTAACTAGACTAACTATCGTGAACTAACTAAAGACCTAACTGACTCATTTATACATTCACTAACTAACTAACTGGATTAAATGACCGAAACGTCCACCGGTGGTTCTAGCTCGTCGACGACCTCGCGTGTGGTATGCTCATATG
***********************************************************************************************************************************......((.....((((((((............)))).............)))).....))*********************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
V053 head |
SRR060689 160x9_testes_total |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................CTACCGACCTAACTGACACAT................................................................................................ | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GTGGTGCTAGTTCGTCGA........................... | 18 | 2 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................GGTGGTTCTAGCTACACGAC.......................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 |
| .............ACTAGTAGACTGACTATTAT.......................................................................................................................................................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 |
| ............................................................................................................................................................................................................CGGTTGTTGTAGCTCGTAGAC.......................... | 21 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 |
| ............GACTAATTCACTAGCTATTG........................................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:4933042-4933292 + | dvi_8953 | CTGATTGATCGACTGATCAACTGAT--------TGATA-ACAGTT----TGAATAAC-----------------------------------------------------------------TGATTGATCGATAGAATTATTGATGAATTCAACA-GC----------------------------------ATTTATT------------------GCTTAGCTGACAAACTAACAGATTGATC----TGATTGATAG--CACTTGATTGATTTCTGGATTGAC--------TGAGTAAATAT--GTAAGTGATTGATTGATT-G----AC-----------------------CTAATTTACTGGCTTTGCAG-GTGGCCACCAAGATCGAGCAGCTGCTGGAGCGCACACCAT---A-----------CGAGTATAC |
| droMoj3 | scaffold_6540:30383779-30384001 - | CTGATTGATTAAAGGACCAAAGAACTAAAAAATCCATTGAT--------TGATTGAT---------------------------------------------------------TGGTTCGCTGGATGACT----G--------ATTGATGA--------AAAGGCTTAAAAACTTAGAAACTG-ACCGA--------CT------------------GAAT--CTGATTGCCTGATTGATTGACTAACT---------A----AGT--AAGAATGATTGACTGATTTTTCAACTGACTGAGAAACCACCA------------AT-GATTGATTG----ACTGA------------------------TC----------------------------------ACGTAACTAACAT---T-----------CTAGTATGC | |
| droGri2 | scaffold_15081:2841297-2841568 - | TTGATTGATTGATTGACTGACTGAT--------TGACAGAAA-AC----TGATTTTAAATTTACATGCTTGAGCATGATTTAAAGAAAATCCCAGATATGCATATATGTGAGATTGATTGACTCTCTAAAT----GATTGACTGACTG-------------------------------------ACTGA--------CTGAAAGATTGTCGATCAATGCACAG----CGAGAAA--------AGT----GAGTTTGTA-----TCCGATTGATTGAATGGCAACT----------------------GAAATGATTGATTGATAAGATTG------------A---------------------TGGCTAAGTAGGGTAACCAC--------------------ACAGGAACCTCTGCTT-----------GCAGCGCAC | |
| droWil2 | scf2_1100000004590:2210896-2210964 - | ATCTATTTGTTTTTATCAGG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCGCCACACTTATTGAACGTATGCTAGAAACGAATCCCT---T-----------CAATTATTC | |
| dp5 | 3:8133826-8133882 - | TTTTTAGGTCGCTCTGTTAATTGAG--------CAATTGCTCGAA----CGA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATCCCT---T-----------CGAGTACAC | |
| droPer2 | scaffold_1:5858587-5858758 + | TGGATTGATAGATTGATGGATTGAT--------AGATTGATGCAT----TGATAGAT-----------------------------------------------------------------TGATGGATT----GAGGGATTGATGAATTG-----CT--------------------------ATGTA--------TT------------------GATGGAGTGATAGATTGATGGATTGATAGATT---------GATGAATTGATAGATTGATGGGCTGAT----------------------GGATTGATGGATTGATA-GGTTGATGG---------------------------------------------------------------------------------------------------AATGTAC | |
| droAna3 | scaffold_13335:1748834-1748919 - | CTAACGGACTGATTGACTAACTGAC--------TGATTGACTAAC----TGACTGAT-----------------------------------------------------------------AGACTGACT----GACTGACTGACCGAC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GACAGTC---G-----------CCGCCAAGC | |
| droBip1 | scf7180000396425:711437-711493 - | TTTCCAGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGCAAACCTTCTGAAACGCATGCTGGACAAGAATCCAT---A-----------CCAGTATAC | |
| droKik1 | scf7180000302577:1524041-1524118 - | CGAACTGACGAACTGACGAACTGAC--------TGACTCGCTGGA----TGACTAAC-----------------------------------------------------------------TCACCAACT----GAGTGAGTACTTAAAGC---------------------------------AATGG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CA----------------------------------------------------------------------- | |
| droFic1 | scf7180000454038:419894-419958 + | TTTGCTAAATTTTAGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCGCTGCCCGCTTTGAGAAAATGTTGGAGGACAACCCAT---A-----------CGAGTACGC | |
| droEle1 | scf7180000491006:1189461-1189655 - | CTAACTGACTAACTGACTAACTGAC--------TATCTGACTAAC----TGACTAGTTAGTCACA----------------------------------------ATGTGCGACTTAGTGACTATTTGGCT----AA------------CTG----ACTTACTGAAT-----------------ATCTCA--------CT------------------ATTAGACTATCTAGCCAACTGATTTTCTGACT---------AACTGACTATCTTGCTAACTGATTGAC----------------------TGCCTTCTTGACCAAAT-GACTGACTG----ACTGG------------------------CT----------------------------------------------------------------------- | |
| droRho1 | scf7180000779914:687998-688191 + | CTGACTGACTGACTGACTGACTGAC--------TGACTGACTGAC----TGACTGAC-----------------------------------------------------------------TGACTGACT----GACTGATTGACTAACTG---------------------------------ACTGACTGACTGATT------------------GACTAGCTGATTAACTGACTGATTGACTGACT---------GA--------CTGACTGACTGACTGAC----------------------TGACTGACTGACTGATT-GACTAACTGACTG----ATTGACTAACTGACTGATTGACTAACT----------------------------------------------------------------------- | |
| droBia1 | scf7180000301754:2030365-2030545 - | TAGATAGATAGATAGATAGATATGA---T----AGATAGATAGAT----AGATAGAT-----------------------------------------------------------------AGATAGATA----GATAGATAGATAGATAG---------------------------------ATAGATAGATAGATA------------------GATAGATAGATAGATAGATAGATAGATAGATA---------GA--------TAGATAGATAGATAGAT----------------------AGATAGATAGATAGATA-GATAGATAGATAG----ATAGAT-------------------------------------------------------------------------------------AGATAGA | |
| droTak1 | scf7180000415734:40264-40432 - | CAGACTGACAGACTGACAGACTGAC--------AGACTGACAGAC----TGACAGAC-----------------------------------------------------------------TGACAGACT----GACAGACTGACAGACTG---------------------------------ACAGA--------CT------------------GACAGACTGACAGACTGACAGACTGACAGACT---------GACAGACTGACAGACTGACAGACTGAC----------------------AGACTGACAGACTGACA-GACTGACAG----ACTGA------------------------------------------------------------------------------------------------C | |
| droEug1 | scf7180000408988:48205-48441 - | CAGACTGACAGACTGACAGACTGAC--------AGACTGACAGAC----TGACAGAC-----------------------------------------------------------------TGACAGACT----GACAGACTGACAGACTG---------------------------------ACAGA--------CT------------------GACAGACTGACAGACTGACAGACTGACAGACT---------GACAGACTGACAGACTGACAGACTGAC----------------------AGACTGACAGACTGACA-GACTGACAGACTG----ACAGACTGACAGACTAACAGACTGACAGAG-AG-GCGGCCGCTGCGCCCA--------------------CCCT---ATCGTTGCTGCTGCAGCG--- | |
| dm3 | chrUextra:3676622-3676817 - | CTAAATGACTAAATGACTAAAGGAC--------TAAAGTACTAAAGGACTAAATGAC---------------------------------------------------------TAAATGACTAGATGACT----A--------A----ATG----ACTAGATGACT-----------------AAATGA--------CTAAAGTACTAAA--T----GACTAAATGACTAAATGACTAAATTACT----AAATTACTA-----AAGGACTAAATGACTAAAGGAC----TAAATGACTAAATGA--CTAA-----------------------------------------ATGACTAAATGACTAA------------------------------------------------------------------------- | |
| droSim2 | x:14689804-14689911 + | CTAACTGACAGACACACTAGCTGAC--------TAGCCGACTTAG----CGACTAAC-----------------------------------------------------------------TGACCAACT----GACAAACTGGCTAACTG---------------------------------ACTA----------------------------------------------------------------------------------GACTAACTGACTTAG----------------------TGACT---TAACTGATA-----------------AA------------------------CT----------------------------------------------------------------------- | |
| droSec2 | scaffold_15:918267-918436 - | CTGACTGACTGACTGACTGACTGAC--------TGACTGACTGAC----TGACTGAC-----------------------------------------------------------------TGACTGACT----GACTGACTGACTGACTG---------------------------------ACTGA--------CT------------------GACTGACTGACTGACTGACTGACTGACTGACT---------GACTGACTGACTGACTGACTGACTGAC----------------------TGACTGACTGACTGACT-GACTGACTG----ACTGA------------------------CT----------------------------------------------------------------------- | |
| droYak3 | X:11490440-11490594 + | CTGACTGACTGACTGACTGACTGAC--------TGACTGACTGAC----TGACTGAC-----------------------------------------------------------------TGACTGACT----GACTGACTGACTAACTG---------------------------------ACTGA--------CT------------------GACTGACTAACTGACTATCTGACTATCTGACT---------AACTCATTGACTCACTGACTAACTGAC----------------------TCACTGACAGACTGTCT-G------------------------------------------------------------------------------------------------------------------ | |
| droEre2 | scaffold_4690:7756832-7756991 + | GAAACTGAGTAACTGAGCAACTGAG--------GAACTGAGCAAG----TGAATAAA-----------------------------------------------------------------TGAGTAACT----GAGTAACTGAGCAACTG---------------------------------AGGAA--------CT------------------GAGTAACTGAATAACTGAGAAATTGAGTAACT---------GAGCAACTGAGCTACTGAACAACTGAG----------------------AAACTGAGTAACTGAGC-AACTGA------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 05:04 AM