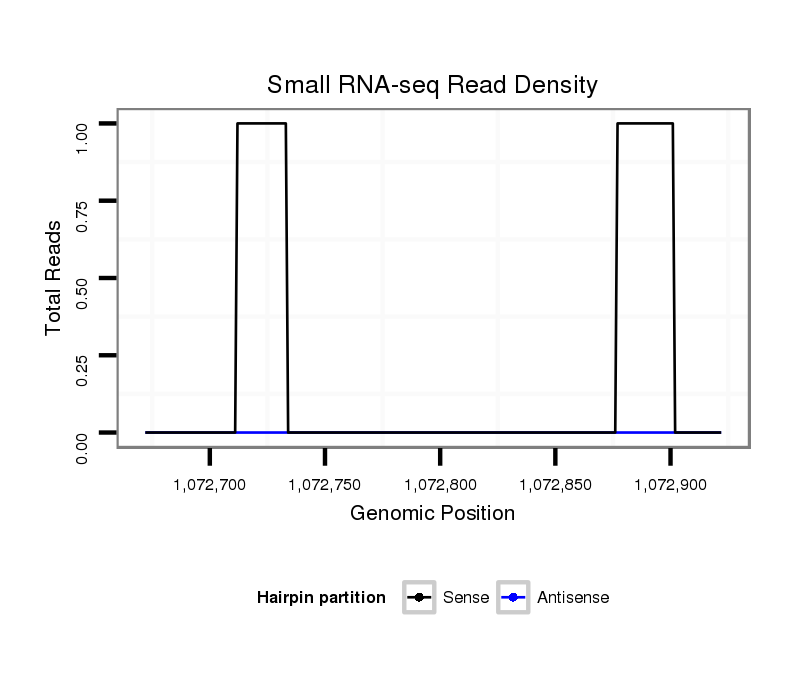
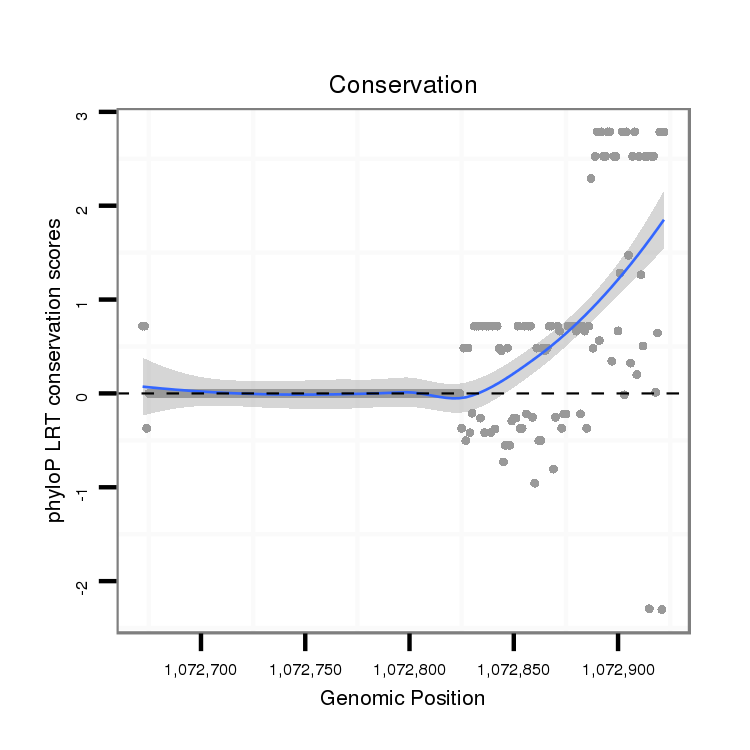
ID:dvi_895 |
Coordinate:scaffold_10324:1072722-1072872 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ15134-cds]; exon [dvir_GLEANR_15431:3]; intron [Dvir\GJ15134-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## ATGACAACATTATTTTTGGTCCAAATTCATGTGAAAATAGTACTCTCAAATATCCGATCTAGACATATGTCAAAATTGTCTCATTCGATTTCAACGAGGGTTTCCTCATTGCTTACTTATTTTATACAACGGACAAGGCCGGGCTATACCATACAGTTTATTTATATATAATCGAAACTTACCTGTTTTTCGTCTTTCTAGGGCATATGTAACCTCGGTGAGCATTGGCGAGAATGTGGCTGGCGGCCTAA **************************************************........(((((...(((..(((((.((....(((((((.(((.((((....)))).)))...((((((..((((...(((......)))...))))..))).)))............)))))))....))..))))).)))...)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
SRR1106729 mixed whole adult body |
M027 male body |
M047 female body |
SRR060672 9x160_females_carcasses_total |
V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060676 9xArg_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................................................................TATGTAACCTCGGTGAGCATTGGCG..................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................TTAGCTTATACAACGCACAAGGC................................................................................................................ | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................TACTCTCAAATATCCGATCTAG............................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................TTATACAACGCACAAGGC................................................................................................................ | 18 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................CCATTTCAACGAGGGTTTCA.................................................................................................................................................. | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TAGTACAAATTCATGGGAAAATA.................................................................................................................................................................................................................... | 23 | 3 | 18 | 0.11 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 |
| ........................................................................................CTTCAACGAGGGATTCCTA................................................................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................CAAATGCATGTACAAATAGTACTC.............................................................................................................................................................................................................. | 24 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................TTCAATTTCAATGAGGGT...................................................................................................................................................... | 18 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................TTCAACGAGGAATTCCT................................................................................................................................................. | 17 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......CATTATTTTTGGTCCAAATTCATG............................................................................................................................................................................................................................ | 24 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TACTGTTGTAATAAAAACCAGGTTTAAGTACACTTTTATCATGAGAGTTTATAGGCTAGATCTGTATACAGTTTTAACAGAGTAAGCTAAAGTTGCTCCCAAAGGAGTAACGAATGAATAAAATATGTTGCCTGTTCCGGCCCGATATGGTATGTCAAATAAATATATATTAGCTTTGAATGGACAAAAAGCAGAAAGATCCCGTATACATTGGAGCCACTCGTAACCGCTCTTACACCGACCGCCGGATT
**************************************************........(((((...(((..(((((.((....(((((((.(((.((((....)))).)))...((((((..((((...(((......)))...))))..))).)))............)))))))....))..))))).)))...)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
SRR060676 9xArg_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
M047 female body |
SRR060657 140_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................................GACAAAAAGCAGAAAGATCCCGTATGC.......................................... | 27 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................CTGTAGAAAGTTTTACCAGAGT........................................................................................................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................GCGTCTACAGTTTTAACAGAGT........................................................................................................................................................................ | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................TTAAGTACACTTTTATCATGAGAGTTT......................................................................................................................................................................................................... | 27 | 0 | 5 | 0.40 | 2 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................CAGTCTAAAGTTTTAACAGAGT........................................................................................................................................................................ | 22 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................TTGTCTATAGTTTTAACAGAGT........................................................................................................................................................................ | 22 | 3 | 7 | 0.29 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................CCGTATAAAGTTTTAATAGAGT........................................................................................................................................................................ | 22 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................TTAAGTACACTTTTATCATGAGAGTT.......................................................................................................................................................................................................... | 26 | 0 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................TTTAAGTACACTTTTATCATGAGAGTTT......................................................................................................................................................................................................... | 28 | 0 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................AAGTACACTTTTATCATGAGAGTTTAT....................................................................................................................................................................................................... | 27 | 0 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................AAGTACACTTTTATCATGAGAGTTT......................................................................................................................................................................................................... | 25 | 0 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................AGTCTACAGTTTTACCAGAGT........................................................................................................................................................................ | 21 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................TGTCTACAGTGTTAACAGGGT........................................................................................................................................................................ | 21 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................TTTACGTACACTTTTATCATGAGAGTTT......................................................................................................................................................................................................... | 28 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................AAAAGTACACTTTTGTCATGAGAGTTT......................................................................................................................................................................................................... | 27 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................TGTCTATAGTTTTACCAGAGT........................................................................................................................................................................ | 21 | 3 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................CCCCCTTTATCATGAGAGTTT......................................................................................................................................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................AGAGGCTAGAGCTGTGTACA..................................................................................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......TGTAATAAAAACCAGGTTTAAGTACAC.......................................................................................................................................................................................................................... | 27 | 0 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................AGCACACTTTTAGCATGAGAGTTT......................................................................................................................................................................................................... | 24 | 2 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................TGTCTAAAGTTTTAAGAGAGT........................................................................................................................................................................ | 21 | 3 | 16 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................TTAAGTACAGTTTTATCATGAGAGTTT......................................................................................................................................................................................................... | 27 | 1 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................AGTCTAAAGTTTTAACAGAGT........................................................................................................................................................................ | 21 | 3 | 18 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......TGCAATAAAAACCAGGTTTAAGTACAC.......................................................................................................................................................................................................................... | 27 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_10324:1072672-1072922 + | dvi_895 | ATGACAACATTATTTTTGGTCCAAATTCATGTGAAAATAGTACTCTCAAATATCCGATCTAGACATATGTCAAAATTGTCTCATTCGATTTCAACGAGGGTTTCCTCATTGCTTACTTATTTTATACAACGGACAAGGCCGGGCTATACCATACAGTTTATTTATATATAATCGAAACTTACCTGTT-TTTCGTCTTTCTAGGGCATATGTAACCTCGGTGAGCATTGGCGAGAATGTGGCTGGCGGCCTAA |
| droMoj3 | scaffold_6496:4969870-4969971 - | ATA------------------------------------------------------------------------------------------------------------------------------------------------------TAATATATTAAAATTTAATCTTATCAAATTTATTGCCTTATCTTTGCAGAATATATGTAACTTCGGTGAGCATTGGCGAGAATGTGGCTGGTGGCTTGA | |
| droGri2 | scaffold_15245:9152915-9153000 + | ATA------------------------------------------------------------------------------------------------------------------------------------------------------T---ACATTTAAATTTCA------ATTATTTGTT-TA------TTGTAGAGCATATGTTACTTCAGTGAGCATTGGCGAAAATGTAGTTGGCGGCTTAA | |
| droWil2 | scf2_1100000004513:2486916-2486951 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGGTAAGCATCGGTGAGAATGTAGCTGGGGGTCTGA | |
| dp5 | 3:1202333-1202368 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAGAACGTGGCCGGGGGCCTGA | |
| droPer2 | scaffold_2:1370550-1370585 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAGAACGTGGCCGGGGGCCTGA | |
| droAna3 | scaffold_13266:3164702-3164737 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAAAATGTGGCTGGAGGTCTAA | |
| droBip1 | scf7180000396427:1631290-1631325 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAAAATGTGGCCGGAGGTCTAA | |
| droKik1 | scf7180000302630:247983-248016 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTCAGCATCGGTGAGAGCGTCGCCGGCGGCCTCA | |
| droFic1 | scf7180000453955:347846-347881 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATCGGTGAGAACGTGGCCGGCGGTCTCA | |
| droEle1 | scf7180000491265:19662-19697 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAAAATGTGGCCGGTGGCCTGA | |
| droRho1 | scf7180000762630:31038-31073 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAGAATGTGGCCGGTGGCCTCA | |
| droBia1 | scf7180000302292:3221421-3221456 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAAAATGTGGCCGGTGGCCTGA | |
| droTak1 | scf7180000415382:149803-149838 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGTGAAAATGTGGCCGGTGGTCTCA | |
| droEug1 | scf7180000409672:4939272-4939305 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTCAGCATTGGTGAAAATGTGGCCGGCGGCCTCA | |
| dm3 | chr2R:2923036-2923071 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGGAAAAATGTGGCCGGGGGCCTAA | |
| droSim2 | 2r:3772377-3772412 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGGGAAAATGTGGCCGGGGGCCTAA | |
| droSec2 | scaffold_1:590926-590961 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGGGAAAATGTGGCCGGGGGCCTAA | |
| droYak3 | 2L:15636548-15636583 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGGGAAAATGTGGCCGGGGGCCTGA | |
| droEre2 | scaffold_4929:19642892-19642927 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCAGCATTGGGGAAAATGTGGCCGGCGGCCTCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:45 PM