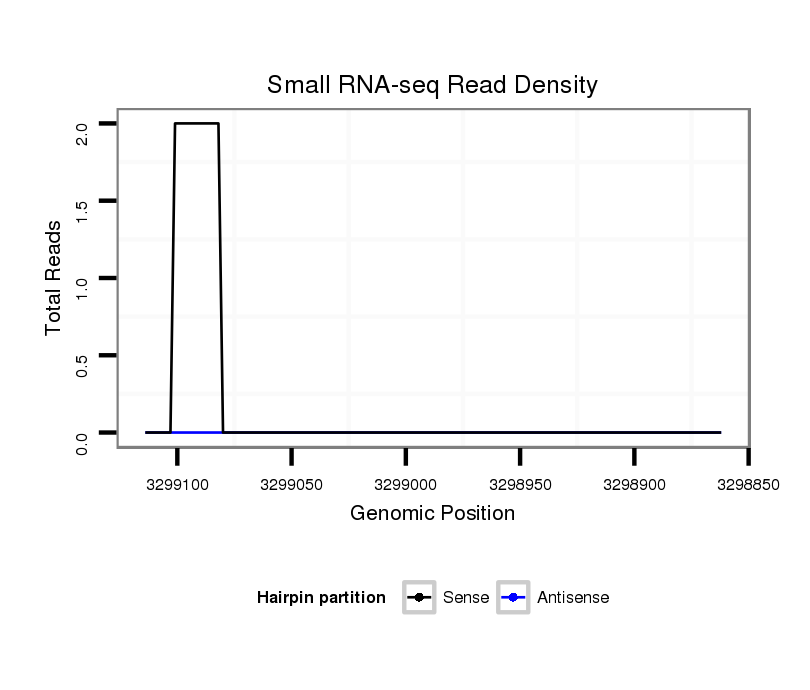
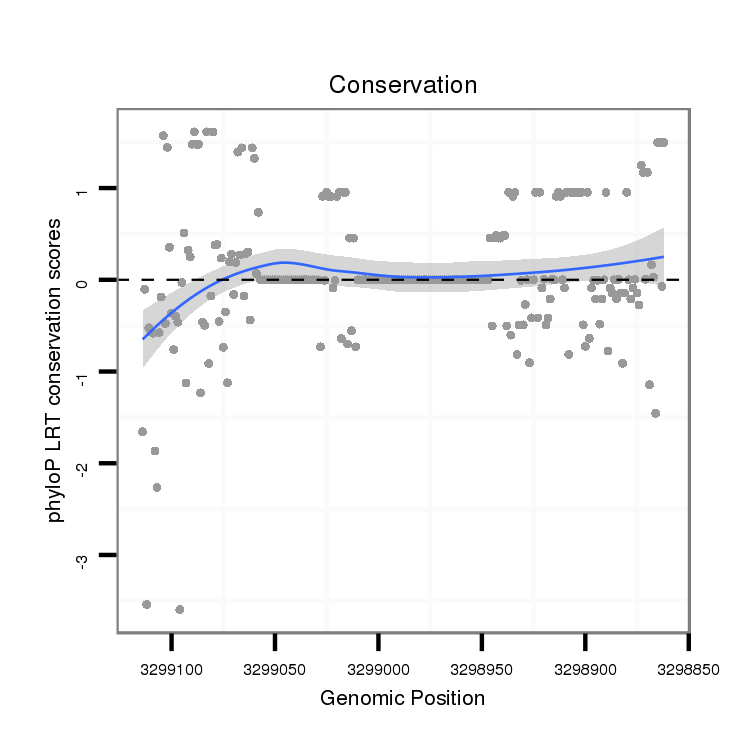
ID:dvi_8720 |
Coordinate:scaffold_12928:3298912-3299064 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ16574-cds]; CDS [Dvir\GJ16574-cds]; exon [dvir_GLEANR_17082:5]; exon [dvir_GLEANR_17082:4]; intron [Dvir\GJ16574-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GGCGGCGACTATGACATGCATATGCTGGAACAGCTAAATCACCAGCTAACGTAAGCAACTTAAACCGGGCAGAGCACTCAGTATGCACTTGGCTCAAATCCTCGATTCAATCCATGTTGTTTGCTGCAAGTGCCTGAATCGTGTCGTATATTATTGGAATGTGAAATACCCTGCTATTTCTAATATCGCCTCTCTTCGTGCAGCAATTTTTATTCTACACTGCGTGGCGCTGACTGGCAGCTGCCGAGCTACT **************************************************....(((......(((((.(((.((((((((((.((((.(((....((((...))))....))).)).)).)))))..))))))))..))).))((.((((((..((((((((........))).)))))))))))))..........)))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060673 9_ovaries_total |
SRR060683 160_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060659 Argentina_testes_total |
SRR060668 160x9_males_carcasses_total |
M047 female body |
SRR1106729 mixed whole adult body |
M061 embryo |
V053 head |
SRR060662 9x160_0-2h_embryos_total |
SRR1106725 embryo_14-16h |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............GACATGCATATGCTGGAACAGC........................................................................................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................GCATATGCTGGAACAGCTGAA....................................................................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............ACATGCATATGCTGGAACAG............................................................................................................................................................................................................................ | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................GCGTGGCGCAGCCTGGTAGC............ | 20 | 3 | 13 | 0.38 | 5 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 1 | 0 | 1 | 0 | 0 |
| ..............CAAGCATGTGCTGGAACAGC........................................................................................................................................................................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................TACTGGAACAGCGAAATCA.................................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................................CGTGGCGCAGCCTGGTAGC............ | 19 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 1 |
| ..........................................................CTTAAACCGGGCAGC.................................................................................................................................................................................... | 15 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................TCGATTCAATCGATGTG....................................................................................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............AAGCATGTGCTGGAACAGCTG......................................................................................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
|
CCGCCGCTGATACTGTACGTATACGACCTTGTCGATTTAGTGGTCGATTGCATTCGTTGAATTTGGCCCGTCTCGTGAGTCATACGTGAACCGAGTTTAGGAGCTAAGTTAGGTACAACAAACGACGTTCACGGACTTAGCACAGCATATAATAACCTTACACTTTATGGGACGATAAAGATTATAGCGGAGAGAAGCACGTCGTTAAAAATAAGATGTGACGCACCGCGACTGACCGTCGACGGCTCGATGA
**************************************************....(((......(((((.(((.((((((((((.((((.(((....((((...))))....))).)).)).)))))..))))))))..))).))((.((((((..((((((((........))).)))))))))))))..........)))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060667 160_females_carcasses_total |
SRR060672 9x160_females_carcasses_total |
V053 head |
SRR060675 140x9_ovaries_total |
SRR060657 140_testes_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................ACCGTGTCGAACTAGTGGTCGAT............................................................................................................................................................................................................. | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................TAAGTGAACCGAGTTTAGAAGG..................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................AGGTTAGGAGGAAAGTTAGGT........................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................TAGCGGGGAGATGCACGT................................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................TCGATTGAAGTCGTTGAATTAG............................................................................................................................................................................................ | 22 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................TTTGTCGATTTATTGGTCGCT............................................................................................................................................................................................................. | 21 | 3 | 14 | 0.07 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................AGAGAAGCACGTCCATGA............................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................GAAGCGAGTTGACGAGCTA................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................AATTTGGCCTGTCTTGTTA............................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:3298862-3299114 - | dvi_8720 | GGCGGC---GACTATGACATGCATATGCTGGAACAGCTAAATCACCAGCTAACG--------TAAGCAACTTAAACCGGGCAGAGCACTCAGTATGCACTTGGCTCAAATCCTCGATTCAATCCATGTTGTTTGCTGCAAGTGCCTGAATCGTGTCGTATATTATTGGAATGTGAAATACCCTGCTATTTCTAAT--------------ATCGCCTCT----CTTCGTGCAGCAATTTTTATTCTAC------ACTGCGTGGCGCTGACTGGCAGCTGCCGAGCTACT |
| droMoj3 | scaffold_6308:169669-169849 - | GAGGGC---CCCTACGATATTAATTTGCTGGGTCACCTAAGTTTTCAGCTAACGTAATCAATCGAGT------------------------------CCTTGGCTCACATTCACT----------------------------------------------------------------CTCTGCTACTGCTCTTAACTAATTTGCATTTCCCTCTTT----CTATGTGCAGCAAGTTTTATAATCC------ACTTCGTCGTGCTACTTGGCAACTGCCCAAATATT | |
| droWil2 | scf2_1100000004963:4706786-4706801 - | CAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGCCCAACTATT | |
| dp5 | XL_group1a:3918895-3918946 + | GAGGAA---AATTATGCTCGCCGT---CTGGAAAATCTGCATTTGCAGATAAGG--------TAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droPer2 | scaffold_11:481859-481910 + | GAGGAA---AATTATGCTCGCGAT---CTGGAAAATCTGCATTTGCAGATAAGG--------TAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droAna3 | scaffold_13117:2653265-2653268 - | TATT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454045:326055-326224 - | GGCACA---ATGTACGATATTTGTTTTCTGGAAGACATGAACCTGCGTATAAAG--------TAAGCA------------------------------CATGGAACAAATA-----------------------------------------------------------------------------TGCTTTAGATCAATCAACATGTCTCTTTTTTGTAACATCCACAGTCAATTTTATAATAATAACACTGAGGATGTCAGCGGCTACAGGATGGCGGAGTACT | |
| droEle1 | scf7180000491272:221690-221727 + | AACGGC---TTCCAAGACTCCAATATGCTGGCCGAGTTGAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000779588:250646-250683 - | AATAGC---TCGCACGACACCCAATTGCTGGCCGAGTTGAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000301345:295839-295847 + | CGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTACT | |
| droTak1 | scf7180000415769:426949-426957 - | CGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTACT | |
| droYak3 | X:3255425-3255478 + | CAGCATCCCACGGACGACTTCGATCCGCTGGTCCAGCTGACCAACGA----AAT--------TAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEre2 | scaffold_4690:2565814-2565860 - | GATGGC---GA---------CGATCCGCTGGCCGAGCTGAGCAACGAAATGAAG--------TGAGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:00 PM