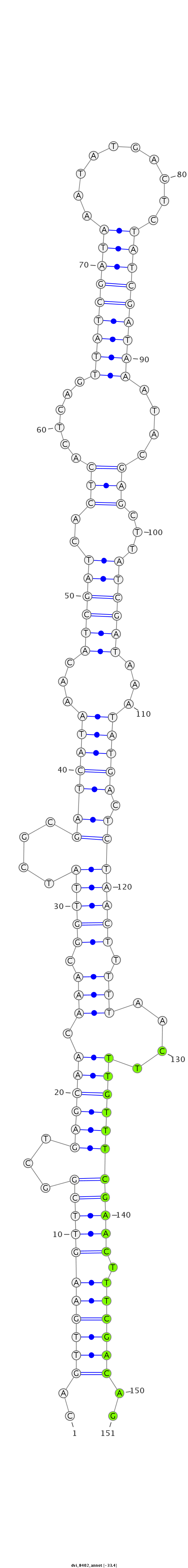
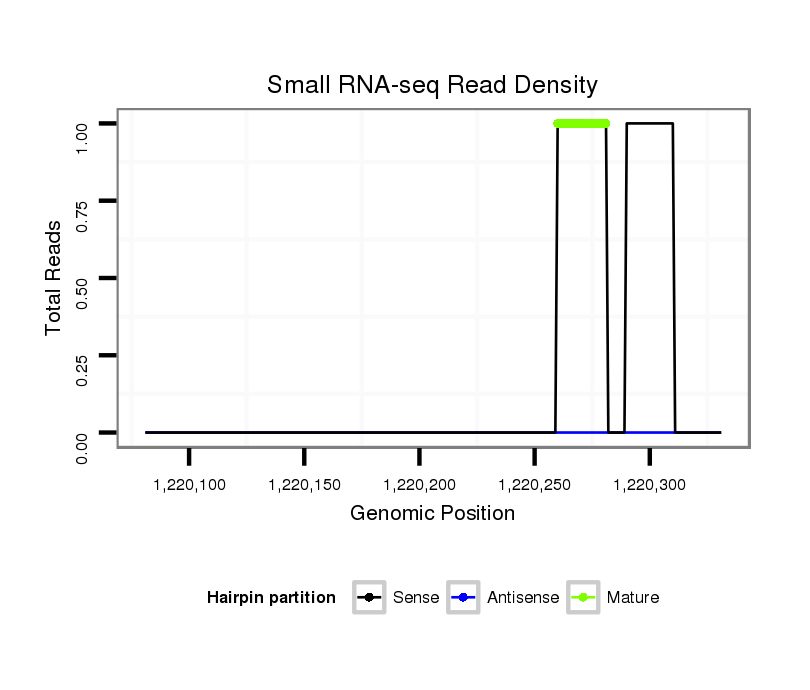
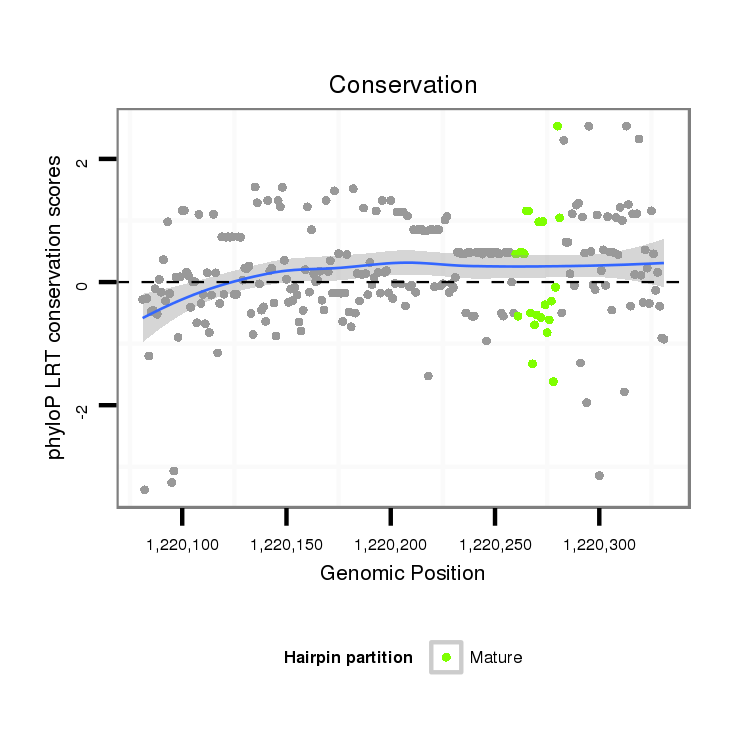
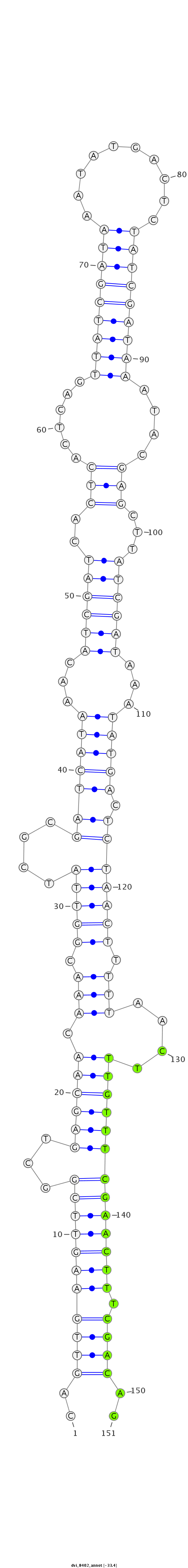
ID:dvi_8402 |
Coordinate:scaffold_12928:1220131-1220281 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -33.4 | -33.4 | -33.1 |
 |
 |
 |
CDS [Dvir\GJ16771-cds]; exon [dvir_GLEANR_17279:3]; intron [Dvir\GJ16771-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGTATCTGTATCTGCGTAATTTACGGCCAGATATCACGTACAGACTTGGACAGTTGAAGTTCGGCTGAGCAACAAACGGTTATCGCGATCATAAACATCGATCACTCACTCAGTTATCGATAAATATGACTCTATCGATAAATACGAGCTTATCGATAAATATGACTCTAACTTTTTAACTTTGTTTCGAACTTTCGACAGGGATCGCTTCCGTAGCCGCAGACGCGCCTCGATAGCCCCGCTGCCCGCCC **************************************************..(((((((((((...((((((.(((.(((((....(((((((...((((((..(((......(((((((((..........)))))))))....)))...))))))...))))).))))))).)))....))))))))))).))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060671 9x160_males_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060658 140_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060689 160x9_testes_total |
SRR060679 140x9_testes_total |
SRR060659 Argentina_testes_total |
SRR060660 Argentina_ovaries_total |
M028 head |
SRR060657 140_testes_total |
SRR060666 160_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060667 160_females_carcasses_total |
M061 embryo |
SRR060656 9x160_ovaries_total |
SRR060675 140x9_ovaries_total |
SRR060678 9x140_testes_total |
M047 female body |
SRR060676 9xArg_ovaries_total |
SRR060677 Argx9_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060680 9xArg_testes_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................ATCGATAAAAACGAGCTTCCCG................................................................................................ | 22 | 3 | 1 | 13.00 | 13 | 2 | 2 | 2 | 2 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................TCGATAAAAACGAGCTTCCCG................................................................................................ | 21 | 3 | 1 | 3.00 | 3 | 1 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................CGATAAAAACGAGCTTCCCG................................................................................................ | 20 | 3 | 10 | 1.60 | 16 | 2 | 1 | 1 | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 2 | 1 | 0 | 1 | 1 | 1 | 0 | 0 |
| ......................................................................................................................................TCGATAAAAACGAGCTTACCG................................................................................................ | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................TCCGTAGCCGCAGACGCGCCT..................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................CTTTGTTTCGAACTTTCGACAG.................................................. | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................CATCGATAAAAACGAGCTTACCG................................................................................................ | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................ATCGATAAAGACGAGCTTCCCG................................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................ACAGGGATTGCCTCAGTAGC.................................. | 20 | 3 | 6 | 0.33 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................GATAAAAACGAGCTTCCCG................................................................................................ | 19 | 3 | 20 | 0.25 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................................................................ATCGATAAGTACGAGCTTCCCG................................................................................................ | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................ATCACGATCTTAAACTTCGA...................................................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................CCATCGATAAAAACGAGC...................................................................................................... | 18 | 2 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................ATATCACGCACAGTCGTGG.......................................................................................................................................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................GATAAATACCAGCTTCCCG................................................................................................ | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................CAGACGCGCCGGGATAGG.............. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................GGCGATCCTAAACTTCGA...................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................CCATCGATAAAAGCGAGCT..................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................TGAAGATCGGCTGAGTAAAA................................................................................................................................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
ACATAGACATAGACGCATTAAATGCCGGTCTATAGTGCATGTCTGAACCTGTCAACTTCAAGCCGACTCGTTGTTTGCCAATAGCGCTAGTATTTGTAGCTAGTGAGTGAGTCAATAGCTATTTATACTGAGATAGCTATTTATGCTCGAATAGCTATTTATACTGAGATTGAAAAATTGAAACAAAGCTTGAAAGCTGTCCCTAGCGAAGGCATCGGCGTCTGCGCGGAGCTATCGGGGCGACGGGCGGG
**************************************************..(((((((((((...((((((.(((.(((((....(((((((...((((((..(((......(((((((((..........)))))))))....)))...))))))...))))).))))))).)))....))))))))))).))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060668 160x9_males_carcasses_total |
V053 head |
SRR060666 160_males_carcasses_total |
M047 female body |
M028 head |
|---|---|---|---|---|---|---|---|---|---|---|
| ...............CATGAGATGCCGGTCTATAATG...................................................................................................................................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................CGTGTTTGTAGCTAGTGGGT............................................................................................................................................... | 20 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................GAGTTTGAAAAGTTGAAACA.................................................................. | 20 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................TGAGTGAGTCAATATCTC.................................................................................................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................CGTATTAGTAGCTAGTGGGT............................................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................................................................AAAGCTTGAAAGATGGCCA................................................ | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 |
| ........................................................................................TGTGTTTGTAGCTAGTGGGT............................................................................................................................................... | 20 | 3 | 15 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................CTATCGGGGGAAAGGGCG.. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:1220081-1220331 + | dvi_8402 | TGTATC--TGTATCTGCGT---AA-----------------------------TTTACGGCCAGATATCACG-----------------------------------------------------------------------------------TACAGACTTGGACAGTTGAAGTTCGGCTG--------------------------------AGCAACAAACGGTTATCGCGATCATAAA-------CATCGATCACTCACTCAG---------------------------------------------------------TTATCGATAAATATGACTCTATCGA-TAAATACGAGCTTATCGATAAATATGACTCTAACTTTTTAACTTTGTTTCGAACTTTCGACAGGGATCGCTTCCGTAGCCGCAGACGCGCCTCGATAGCCCCGCTGCCCGCCC |
| droMoj3 | scaffold_6473:13771936-13772134 - | TGTTGC--TGTATCTCGAT--------------------------------AATTTACAATCGGCTATCAGAAAT-----GCCCGCATT----------------------------------------------------------------------------GACAGCTGAAGATCAGTTG--------------------------------AGCG-----------G-------ACAGA-------CATCG------------G---------------------------------------------------------TTATCGAC------------------------TGAATCTATCGGTATTTATGAGTCTAACCATTT-GCATTGTTTACGACTCTCGGCAGGGATCGCTTTCGTAGCCGCAGACGTGCTTCAATCGCCCCGCTGCCCGCCC | |
| droGri2 | scaffold_15203:8491375-8491612 - | GTTAAC--GA-------AT--------------------------------AATTTATAATCGAA-GCCATAAATACAATGT-----------------------------------------------------------------------------------GACAAGTGAAATTAACTTGGTCA-CATTCACTATC----------------AGCGATCAG---------CGGTCGTAAATATTGAGTCACATTCAATCACATAGCTTTGTGTACATTCGTTAATATGTAGGACACTCGAACTTAAACTTGATTTGTTG---------------------------------------------------------------------------TTGTTG-ATCGTGACAGGGATCGCTTTCGAAGTCGGAGACGCGCCTCAATTGCCCCGCTGCCCGCCC | |
| droWil2 | scf2_1100000004909:11963830-11963906 + | ACCTTT--TGTCTCTTTCT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTGTTCTAGTGATCGCTTTCGCAGCCGGCGACGTGCCTCGATTGCCCCCCTGCCCGCTC | |
| dp5 | XL_group3a:1931853-1931938 + | CTCTCT--CTGA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------C-------TGACTGTTTTCCGTT-------------------------------------------------------------------------------------------------------------------------------------------TTCCTTCAAGCGATCGCTTTCGCAACCGGAGGCGTGCCTCCATCGCCCCTCTACCGGCCC | |
| droPer2 | scaffold_11:2747751-2747802 - | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGATCGCTTTCGCAACCGGAGACGTGCCTCGATCGCCCCTCTACCGGCCC | |
| droAna3 | scaffold_13340:4310049-4310182 + | TTTGTGGCCGTATCTGAAT---CTG------CC--------------------------------TT---------------------------------------------------------------------TCATG--CCAGCCTCCATGTGCAGATCTGGGTATCTGGG-----------------------------GATCCTCAATCGAGCG-----------TAGCATTTACAGA-------CATCATTG--------------------------------------------------------AGTTGTCTCTGCCACTTACGAAAAAG--------------------------------------------------------------------------------------------------------------------GCTCTGCC | |
| droBip1 | scf7180000395553:49945-50019 + | TAATTT--TTAAAATTTC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTTTTAGTGATCGTTTCCGGAGCAGAAGGCGTGCCTCTATCGCCCC-CTCCCCGCCC | |
| droKik1 | scf7180000302577:1583421-1583564 + | ATTAAT--TTAATTTGTTA---TAA------------------------------------------------AT-----TTTCCTATT----------------------------------------------------------------------------T---------ATTCAAATGAAATCTTA---ATTTTTGTAAATTTTAATTTAAATATTTATGTAT------------------------------------T-------------------------------------------------------------------------------------------------------------------------------------------TTCCCCATAGCGATCGCTTTCGAAGTCGAAGACGCGCCTCAATTGCCCCTCTGCCTGCTC | |
| droFic1 | scf7180000454072:3390999-3391088 + | TATATC--TGTATCTGCAT---CTG------CA----------------------------------------GT-----CCGCACATT----------------------------------------------------------------------------G-------GCATTCAACTGGCCT-TCG---ACTTT---CGACCTTCGACCGAGTATTTGTCTATT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AT------------------------------------------------------TGCG | |
| droEle1 | scf7180000491356:166460-166512 + | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGATCGTTTCCGCAGTCGGAGACGTGCCTCGATTGCCCCCCTGCCCGCGT | |
| droRho1 | scf7180000779467:463238-463290 - | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGATCGTTTCCGCAGTCGGAGACGTGCCTCGATTGCCCCCCTGCCCGCTT | |
| droBia1 | scf7180000302421:2021557-2021675 - | TGTGTC--TGTGTCTGTGT---ATC------T------------------------G--------------------------------------------------------------TGGCCAGGTACGCCAAGCCAAACACTAACAAT--------------G----TTGATTC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATG-ACTCTGCCACAGATAGTTTGCCCAGTTTCAGAGC------CACTGGCCACTCACCCTGCT | |
| droTak1 | scf7180000415769:918174-918231 + | TTT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TACAGCGATCGTTTTCGAAGTAGAAGACGTGCCTCGATTGCCCCTCTGCCCGCCC | |
| droEug1 | scf7180000409243:58977-59094 - | TGTATC--TGTGTTTGCGA---CTC------CCCCGGCAGCAGCTACCGGTTTTTCAAAAGCAACCT-CTTGAAT-----T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCACAGCGGCAGCGCAAAAAACAGACATCGTATAAACATCGACCGTCGAC---GCA | |
| dm3 | chr3L:9839626-9839718 + | TGTATC--CGTTGTGGCCAAATTTGGGTTTGCC--------------------------------CG---------------------------------------------------------------------CTATTCACAAACTTC--------------CATGATT----A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAACTTG-------GTTATCATACCT----TATTCATTCATTCGGTT | |
| droSim2 | x:5251437-5251495 - | TTT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGCAGAGATCGTTTTCGGAGCCGGAGACGCGCCTCGATTGCTCCTCTGCCCGCCC | |
| droSec2 | scaffold_9:1445143-1445270 - | TGAATC--TGTATCTGAAT---CTG----------------------------------------------------------------------------------------------AGGCCAAGTCCGC--------------------------------------------------------------------------------------CGACTTATGATAATAATCATTATAAA-------TTATTATAATCCAATCGGCTTTGAA--------------------------------------------------TAATTAATAAATACGTTTATATCGGCTATATGCGAACTC-----------------------------------------------------------------------------------------------AGCGA | |
| droYak3 | 2R:17883702-17883871 - | TGAATT--TGTATCTGAAT---CTG------TA-----------------------------------------T-----CTGAAAATGTATCTGAATCTAATTCTGAAACTGAATCTGAGGCCAAGTCCGG--------------------------------------------------------------------------------------CGACTTATGATAATAATCATTATAAA-------TTATTATAATCCAATCGGCTTTGAA--------------------------------------------------TAATTAATAAATACGTTTATATCGCCTATATGCGAACTC-----------------------------------------------------------------------------------------------GGCGA | |
| droEre2 | scaffold_4690:2958189-2958263 - | TTTTAT--TT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTATTTATTTTCTGCAGCGATCGTTTTCGGAGCCGCAGACGCGCCTCGATTGCTCCTCTGCCCGCCT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 03:46 AM