
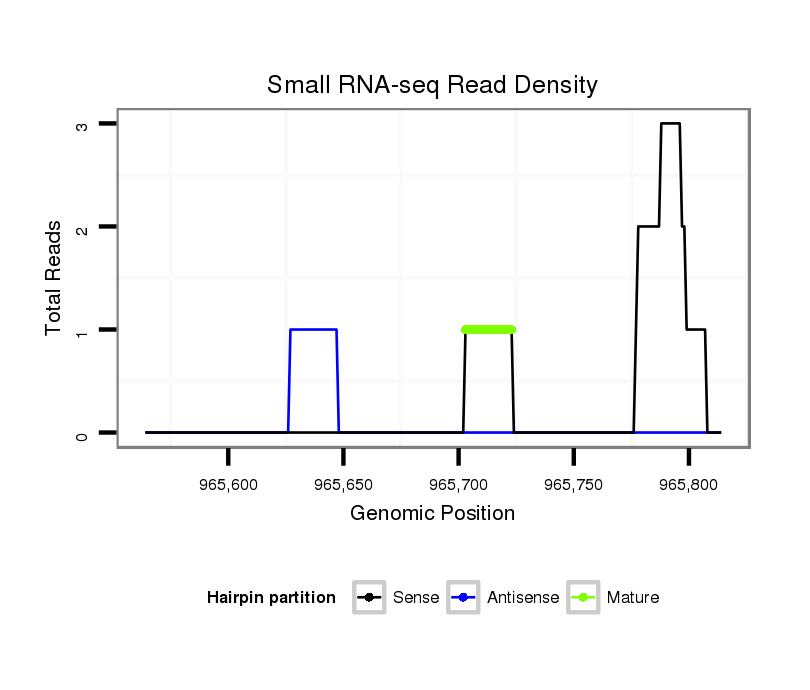
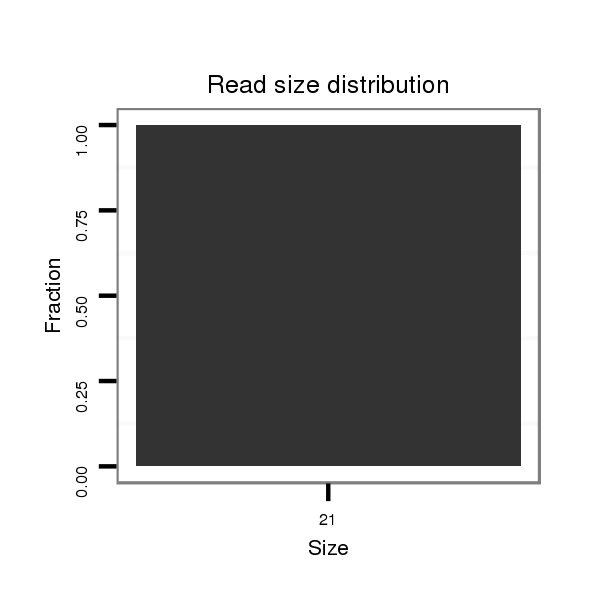
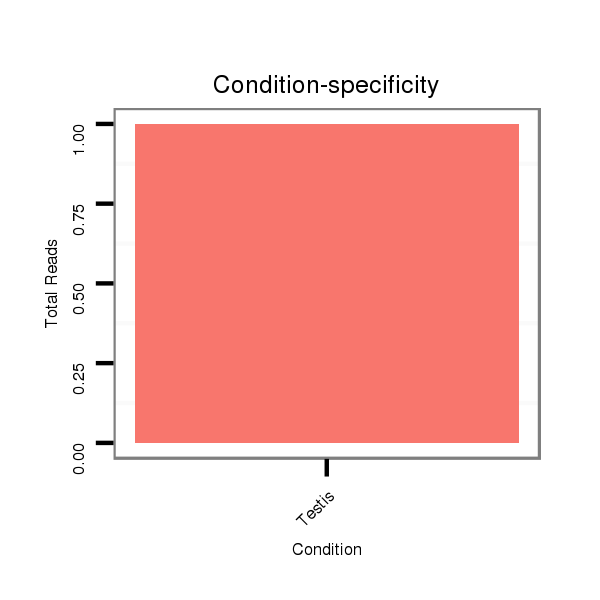

ID:dvi_837 |
Coordinate:scaffold_10324:965614-965764 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -23.2 | -23.2 | -23.0 |
 |
 |
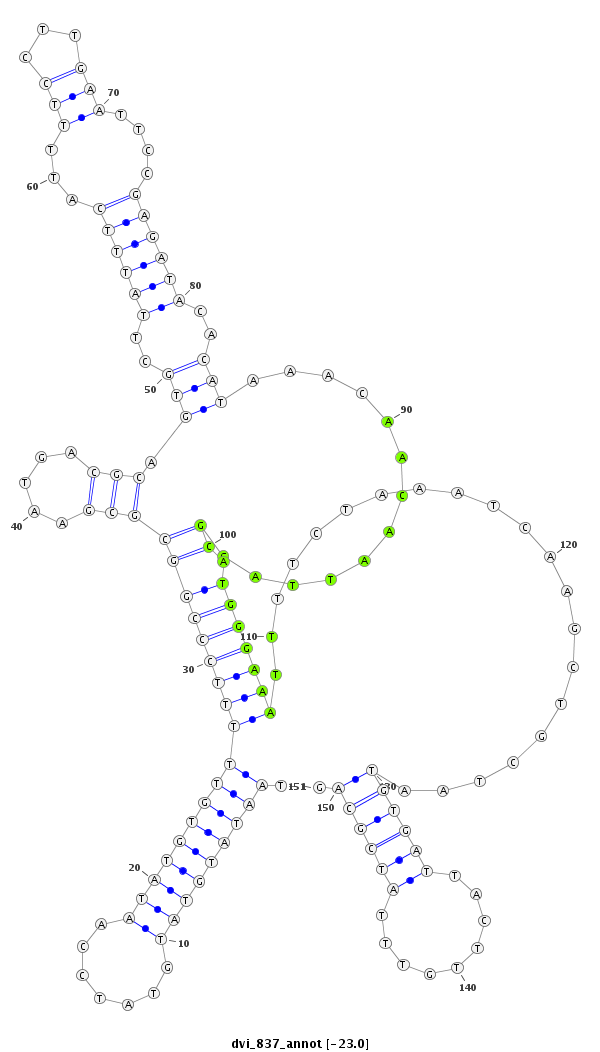 |
exon [dvir_GLEANR_15413:2]; CDS [Dvir\GJ15114-cds]; intron [Dvir\GJ15114-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GCATTTAACATTTGAAATCAGAGTTCTGCCAGTGTACATGCAAATGTAAATAATATGTATGTATCCAATATGTGTTTTTCCCGGCGCGAATGACGCAGTGCTTATTTCATTTTCCTTGAATTCCGAGATACACATAAACAACAATTAGGCATGGGAAATTTTCTAAATCAAGCTGCTAATGTGATTACTTGTTTATCGCAGAGCCCCTCAACAATGATGAGCGTTATCCACCGATACCATTGCCAGCGCCA **************************************************.(((((((((.......)))))))))((((((((((((.....))).(((..((((((...(((...)))....)))))))))...............)).))))))).....................((((((.........)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060687 9_0-2h_embryos_total |
M047 female body |
SRR060657 140_testes_total |
SRR060669 160x9_females_carcasses_total |
SRR060680 9xArg_testes_total |
SRR060658 140_ovaries_total |
SRR060676 9xArg_ovaries_total |
V053 head |
SRR060668 160x9_males_carcasses_total |
GSM1528803 follicle cells |
SRR060663 160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................................................................CGATGAGCGTTATCCACCGATT............... | 22 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................TATCCACCGATACCATTGCC....... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................TGATGAGCGTTATCCACCGAT................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................ATGATGAGCGTTATCCACCG.................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................AACAATTAGGCATGGGAAATT........................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................TTTCCCGGCCGGAATGAC............................................................................................................................................................. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......ACCATTTGAAATCAGTGTT.................................................................................................................................................................................................................................. | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................GCTCGTATGACGCAGTGGT..................................................................................................................................................... | 19 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................TTGGGCGTGGGAAATGTTCT....................................................................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................AATTCCGAGGTACACCAAAA................................................................................................................. | 20 | 3 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................TTGGGCATGGGACATCTTCT....................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................TGTGTTTTTCCAGGC...................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......ACCTATGGAATCAGAGTT.................................................................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CGTAAATTGTAAACTTTAGTCTCAAGACGGTCACATGTACGTTTACATTTATTATACATACATAGGTTATACACAAAAAGGGCCGCGCTTACTGCGTCACGAATAAAGTAAAAGGAACTTAAGGCTCTATGTGTATTTGTTGTTAATCCGTACCCTTTAAAAGATTTAGTTCGACGATTACACTAATGAACAAATAGCGTCTCGGGGAGTTGTTACTACTCGCAATAGGTGGCTATGGTAACGGTCGCGGT
**************************************************.(((((((((.......)))))))))((((((((((((.....))).(((..((((((...(((...)))....)))))))))...............)).))))))).....................((((((.........)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR1106725 embryo_14-16h |
V053 head |
SRR060677 Argx9_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060658 140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................AGGTTATACACAAAAAGGGCC....................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................TAATCCGTACCCTTTGAAAGA....................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................GACTCAAGACGGTCACCGGTA.................................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................AAAAAATTTAGTTCTACGAT......................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ............................................................................................................................CTCTATGACTAATTGTTGTTA.......................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................AGTACGACGATTACTCTA.................................................................. | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................GAATAAAGTAATAGGTAC..................................................................................................................................... | 18 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................................................................................................TAAGGTCGACGATTACTCTA.................................................................. | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_10324:965564-965814 + | dvi_837 | GCATTTAACATTTGAAATC------AGAGTTCTGCCAGTG--TACATGCAAAT-GTAAATAATATGTATG------TATCCAATATGTGTTT---------TTCCCGGCGCGAATGACGCAGTGCTTATTTCATTTTCCTTGAATTCCGAGATACACATAAACAACAATTAGGCAT-GGGAAATTTTCTAAATCAAGCTGCTAATGTGATTAC-----------------TTGTTTATCGCAGAGCCCCTCAACAATGATGAGCGTTATCCACCGATACCATTGCCAGCGCC---A |
| droMoj3 | scaffold_6496:807773-808019 + | GTATTTA---TTTGCAACTCGACTCTTAGC-ATGTATGTG--TGTATGTCGGT-ATGGAT-----GTGTGTATCTGTATACAATATGCGTTT-------T-TTCCCGGTACAAATGACGCA-------------------TCAAATCTGTGATATGGACAACT---AGTTTCGCATTCGTAGATTC----AACAAACCTGCTCATGCAAATAATTAAAATAACACAACAATTGTTTTTTGAAGGGCCCATCAATAATGGAGAGCGCTATCCACCAATACCCTTACCACCGCC---G | |
| droGri2 | scaffold_15245:6101216-6101457 + | TAAATAAGTGAGCCGAATG------ATAGAAATGCATGCAATAATATGTATATAGTAGATATCATGTATG------TATCCCATTTGTGAAACGTTCCTTGTTCCGCTTGCAAACAACTTA-------------------TTTATGCGTCGTCATAAATAATCACTATCAAGAAAC-GGGGAATTCTCTAAAACGATAATCTAATGGAAATGTTTG----------------AAATTTTGTAGAGCGAGTCTACCAGGGAGAGCGTTATCCACCTATACCG---CTACCGCC---A | |
| droWil2 | scf2_1100000004513:5565869-5565925 + | TT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTCGCAGAACCACTTAATAATGGCGAACGATATCCACCTATACCATTGCCT---CC---A | |
| dp5 | 3:13380298-13380362 - | TAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCGCTAATCGCAGAGCCCCTCAATAATCATGAACGCTATCCTCCTATTCCGTTGCCACCGCC---- | |
| droPer2 | scaffold_2:8254189-8254253 + | TAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCGCTAATCGCAGAGCCCCTCAATAATCATGAACGCTACCCTCATATTCCGCTGCCACCGCC---- | |
| droAna3 | scaffold_13266:16148954-16149017 + | ATTC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTTTGCAGTACCCGTAAATAATGGTGAACGTTTTCCGCCAATTCCACTTCCTCCGCC---- | |
| droKik1 | scf7180000302471:449033-449087 + | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTACCTGTCAACAACACTGATCGCTATCCGCCGATCCCACTGCCGGCACCTCCA | |
| droFic1 | scf7180000453215:384440-384498 - | TG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTGCTCCAAAG---CCCAAAATAAATGAGCGTTACCCACCGATTCGATTGCCAGAACC---T | |
| droEle1 | scf7180000489105:192-236 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCAGAAACAATGACCGTTACCCGCCGATTCCATTGCCAGCACC---T | |
| droRho1 | scf7180000780226:298374-298412 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATCCTGATCGTTACCCACCGATTCCATTGCCAGCACC---T | |
| droBia1 | scf7180000302291:1656353-1656412 + | TTG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTGTTTTAGTA---CCCAACAACAATGACCGCTTCCCCCCGATTGCATTGCCAGCACC---G | |
| droTak1 | scf7180000415088:312920-312978 - | TG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTGTTGCAGTT---CCCACCAACAATGACCGCTACCCACCGATTCCATTGCCAGCCCC---T | |
| droEug1 | scf7180000409672:5848696-5848745 + | AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCACCCAAAAACAATGACCGTTACCCACCTATTCCATTGCCAGCGCC---T | |
| droSim2 | 2r:4651714-4651758 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCGAAAACTCTGACCGTTACCCACCGATTTCATTGTCAGCCCC---T | |
| droSec2 | scaffold_1:1465641-1465685 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCGAAAACTCTGACCGTTACCCACCGATTTCATTGTCAGCCCC---T | |
| droEre2 | scaffold_4929:18799204-18799248 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCGAAAACTCTGACCATTACCCAACAATTTCATTGTCAGCGCC---T |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:22 PM