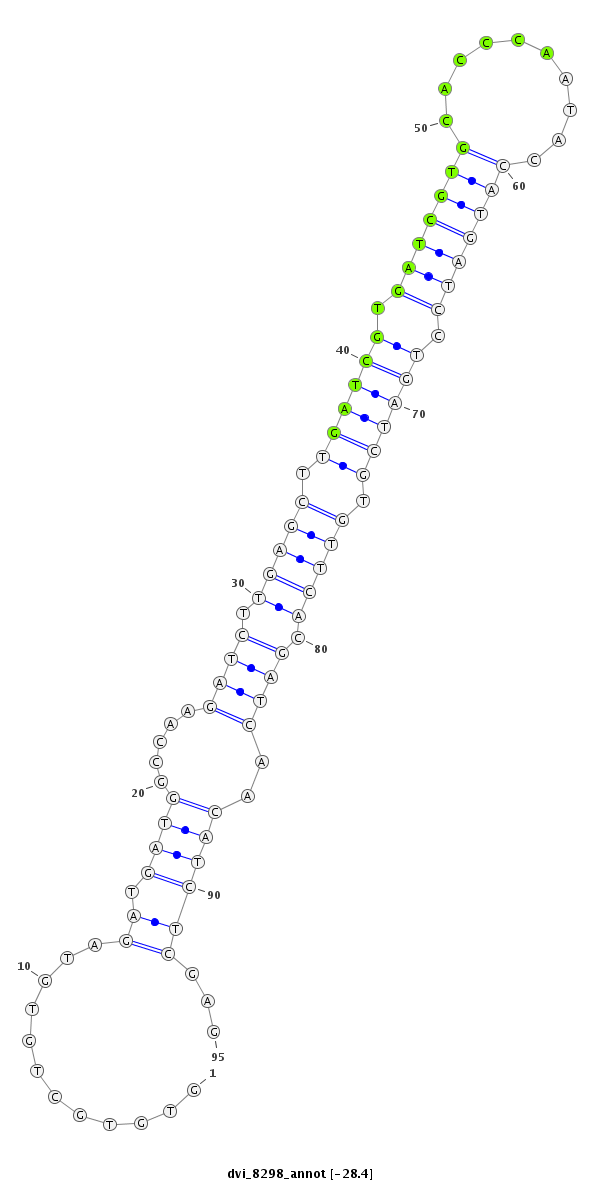
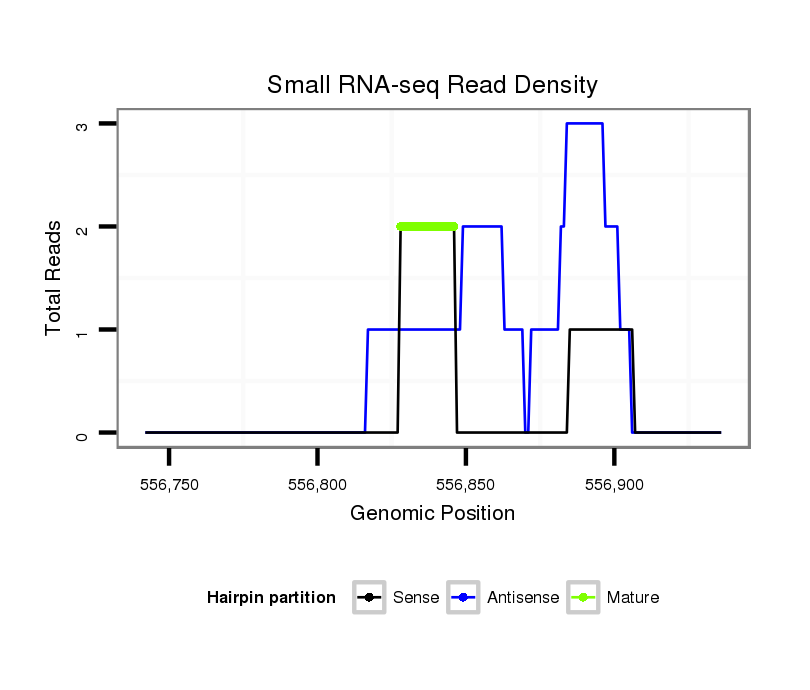
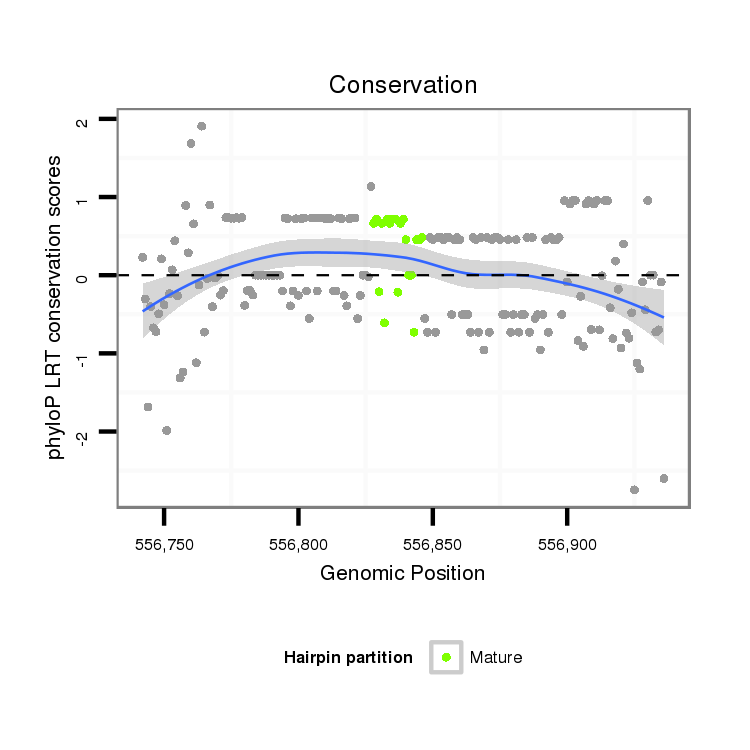
ID:dvi_8298 |
Coordinate:scaffold_12928:556792-556886 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -29.1 |
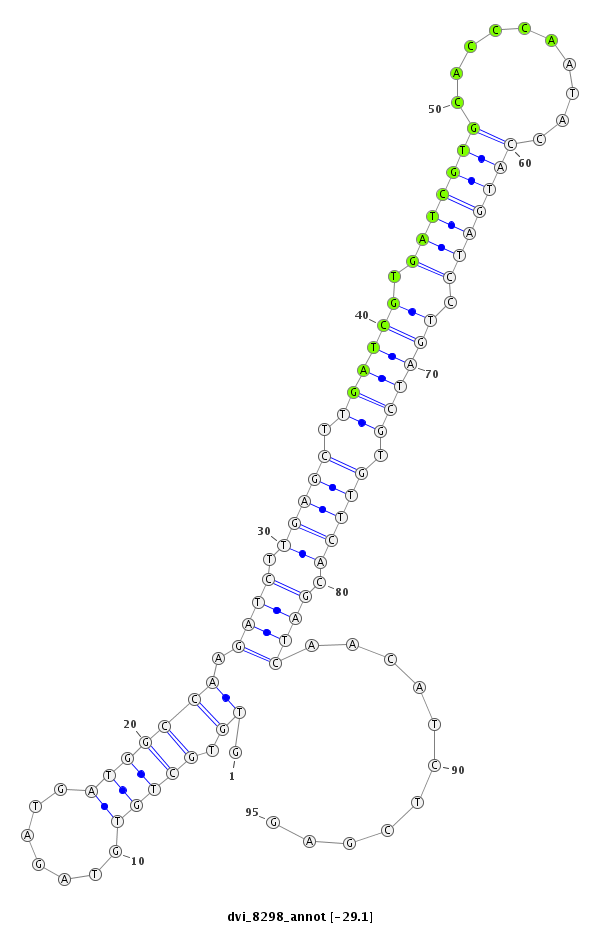 |
CDS [Dvir\GJ16738-cds]; exon [dvir_GLEANR_17244:1]; CDS [Dvir\GJ16738-cds]; exon [dvir_GLEANR_17244:2]; intron [Dvir\GJ16738-in]
No Repeatable elements found
| ----------------##################################-----------------------------------------------------------------------------------------------################################################## TAGACTTATGCAAAGCATGATCATAACCGTGATCGTGAGCATGATCAAGCGTGTGCTGTGTAGATGATGGCCAAGATCTTGAGCTTGATCGTGATCGTGCACCCAATACCATGATCCTGATCGTGTTCACGATCAACATCTCGAGAACCCGATCGTGATCATCATCAAGACATTGAGCGTGATCATGAACCCGAT **************************************************............((.((((.....((((.(((((.((((((.(((((((..........))))))).)))))).))))).))))..))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060677 Argx9_ovaries_total |
V053 head |
SRR060682 9x140_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060672 9x160_females_carcasses_total |
M027 male body |
M028 head |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................GATCGTGATCGTGCACCCA.......................................................................................... | 19 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................AATCGTGATCGTGCACCCA.......................................................................................... | 19 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................AGAACCCGATCGTGATCATCAT.............................. | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................CGTGAGCGTGATCAAGCTTGTG............................................................................................................................................ | 22 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............AAGCATGAACATCACCGT..................................................................................................................................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TTACCAGGAGCCTGATCGTG...................................................................... | 20 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................CATGATCAAGGGTGTGACGT........................................................................................................................................ | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................GAGCAGGATCCAGCGCGTGCT.......................................................................................................................................... | 21 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................ACCGTGTTCACGATAAAG.......................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................TCTTGAGGTAGATCGTCA..................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................................................................CCATGATACTGAACGTGAT.................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .......................................................................................................................................................ACCGTGATCATCATCAAC.......................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
ATCTGAATACGTTTCGTACTAGTATTGGCACTAGCACTCGTACTAGTTCGCACACGACACATCTACTACCGGTTCTAGAACTCGAACTAGCACTAGCACGTGGGTTATGGTACTAGGACTAGCACAAGTGCTAGTTGTAGAGCTCTTGGGCTAGCACTAGTAGTAGTTCTGTAACTCGCACTAGTACTTGGGCTA
**************************************************............((.((((.....((((.(((((.((((((.(((((((..........))))))).)))))).))))).))))..))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
M027 male body |
SRR060681 Argx9_testes_total |
M047 female body |
M028 head |
M061 embryo |
SRR060675 140x9_ovaries_total |
V053 head |
SRR060682 9x140_0-2h_embryos_total |
SRR060655 9x160_testes_total |
SRR060661 160x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................................................................CTAGTTGTAGAGCTCTTGGGCTAGC........................................ | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................TAGCACAAGTGCTATTTGTAG....................................................... | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................CACGTGGGTTATGGTACTAGGACTA.......................................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................TAGAACTCGAACTAGCACTAG................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........CGTGTCGTACTGGTATTGGC...................................................................................................................................................................... | 20 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................CAGTAGTTCTGTAACTCGCAC.............. | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................AGCTCTTGGGCTAGCACTAG................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................TGGTACTAGGACTAGCACAAG................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................CTCTTGGGCTAGCACTAGTAGT............................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................AGTTGGACTAGCTCAAGTGC................................................................ | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........TGTCGTACTGGTATTGGCCC.................................................................................................................................................................... | 20 | 3 | 8 | 0.25 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........TGTCGTACTGGTATTGGC...................................................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................GTAATGCTACCAGGACTAGC........................................................................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................AATGGTCCTCGGACTAGCAC...................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................CTCTGGGGATAGCACTAT................................... | 18 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................GGCTAGCAGTAGGAGTAG............................. | 18 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................CAAGCACTCGCACAAGTGC................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:556742-556936 + | dvi_8298 | TAGACTTATGCAAAGCATGATC-----ATAACCG----------------------------------------------------------------------------TGATCGTGAGCATGATCAAGCGTGTGCTGTGTAGATGATGGCCAAGATCTTGAGCTTGATCGTGATCGTGCACCCAATACCATGATCCTGATCGTGTTCACGATCAACATCTCGAGAACCCGATCGT------------------GATCATCATCAAG--ACATTGAG--C-----GTGATCATGAACCCG------------AT |
| droMoj3 | scaffold_6328:1516087-1516315 + | TAAGCTTATACAAAACATGAGC-----ATAATCGGGTG----------CACTATCATGAGCATGTTCCTGACTTTGACCTTGAGCGCAGTTGATGATCGTGAATGGGACC-------------------------------------------------------TTGATCCTGATTGTG--ACCATGACAATGATTCTGGCTTTGGTGAAGATCGGCCCCGTAATATTGTGCTCGTATCTTTTCAAAAATAATGAATCATTGGCATG--ATATTACGATCGAATGATGTTTAAGAACCAA------------AA | |
| droGri2 | scaffold_14853:7911690-7911806 - | TAAGCTTATACAAAGCAT----TT-----------ACG----------CAT-GCTGTGAGCGTGACAGTGACTGTGATCGTGACAGTGACTG-TGATCGTGACTGTGATC-------------------------------------------------------GTGACCGTGATCGT--------------------------------------------------------------------------------------------------GACCGTGATCATGACTACC------------------A | |
| droWil2 | scf2_1100000004512:423174-423220 + | TGTACATACA--ATATACGAAC-----AATATTT-ATG----------TAGA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATTATGCGTGTG------------------C | |
| dp5 | XL_group1a:6254489-6254500 - | TATACATATATA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13250:878435-878533 - | TAGATTGAATTAAATAATGA-----TAAC----T-TTGCGATAATATTCG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCATGGCCAGGCTAATTTATTGTTCTATTTAAATTAATAATTATAAACATCTCGAGCT | |
| droBip1 | scf7180000395198:51423-51472 - | TTGGTTTGTGTTTGT-------------------------------------------------------------------------ATCG-AATTCGTAACTG-----AAATCGTGATTGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATTA------------------A | |
| droFic1 | scf7180000452523:908092-908130 - | TAAAGCCACATTAAAGAG----TTGCAAA----G-TATAA--------------------------------------------------------------ATA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAC------------------C | |
| droEle1 | scf7180000490141:264113-264179 - | TCACCTTCTTAAAATTATGATT-----AT----G-CCCCGC--------------------------------------------------------------------------------------------ATGACGAGTGCATAATGGCTGAGTGCTTCT--------------------------------------------------------------------------------------------------------------------------------ATTTG------------------A | |
| droRho1 | scf7180000777217:294572-294594 + | TTAAGCCACG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCATGATCATG------------------A | |
| droBia1 | scf7180000302142:78356-78368 + | TATACTTATGTAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415103:408672-408710 - | GTGACTAGCGCAATA-------------------------------------------------------------------------ATTA-TAATTATAAGTGTGAAC-------------------------------------------------------TT---------------------------------------------------------------------------------------------------------------------------------------------------G | |
| droEug1 | scf7180000409533:368749-368778 - | TATATATATATAAATTGTGAAA-----AT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAATG------------------T | |
| droSim2 | 3r:4835023-4835090 - | CAGATTCATGAAGGGAATGG-----TAAA----G-TCCAAAGCCTGTTCA-----------------------------------CGCTCCG-A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGTGATGGTCAAATCCGTG------------------A | |
| droSec2 | scaffold_27:264476-264543 - | CAGATTCATGAAGGGAATGG-----TAAA----G-TCTAAAGCCTGTTCA-----------------------------------CGCTCCG-A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGTGATGGTCAAATCCGTG------------------A | |
| droEre2 | scaffold_4845:5228432-5228452 - | GAGCACCATCATGATCATGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:04 PM