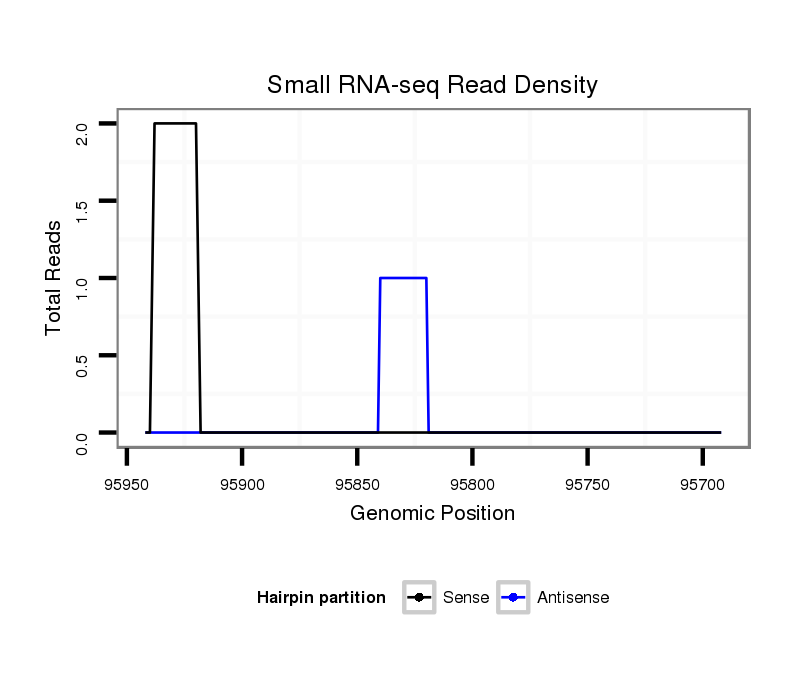
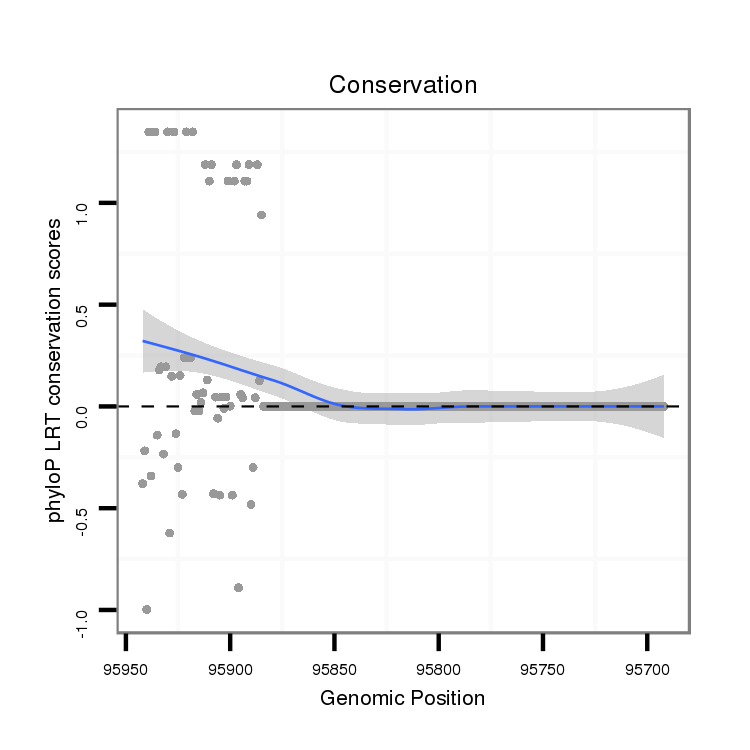
ID:dvi_8203 |
Coordinate:scaffold_12928:95742-95892 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_17197:1]; CDS [Dvir\GJ16694-cds]; intron [Dvir\GJ16694-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ACGTCAACGAAATAATTGCGCTGCTCACCAACGAAGCCGCCGCGGAAAAGGTATGTACAAGAGGAAATGCTCATCTATAGGTTCTTGTTGAAAATGTCCCTCTGGCATTTTGAGTCCACACTTTTGGGGAAATAGCTGTTAAATAAATGTCCCATTCAAGGCTTTTTTACATTCACAACAAACACGACTGCAACACGCAGTCGCAGCCGCCGTTAGCCGAACAGCTGTTTTTTTTTTTCACGCTGAGCGCA **************************************************...((((...(((......)))...)))).((..((((((..(((((......(((..(((((((...........((((...................)))))))))))))).....)))))..))))))..)).(((((.....)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060664 9_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060677 Argx9_ovaries_total |
SRR060660 Argentina_ovaries_total |
V053 head |
GSM1528803 follicle cells |
SRR1106729 mixed whole adult body |
M028 head |
M027 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....CAACGAAATAATTGCGCTG.................................................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................ACGAAGCCGCCGCGGAAAAGC........................................................................................................................................................................................................ | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...TCAACGAAATAATTGCGCTGC................................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TTTTTTTTTTTCACGATGAGGGC. | 23 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................TACAGGTTCGTGTTGAAA.............................................................................................................................................................. | 18 | 2 | 7 | 0.29 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ....................CGGCTCATCAACGAAGCTGC................................................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................GTTTTTTTTTTTCACGAT....... | 18 | 1 | 15 | 0.13 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| ................................................................................................................................................................................................ACACGCAGTCGGAGC............................................ | 15 | 1 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................GTACTGATCACCAACGAAG....................................................................................................................................................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................GGTACGGACAAGGGGAAAT....................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TGCAGTTGCTTTATTAACGCGACGAGTGGTTGCTTCGGCGGCGCCTTTTCCATACATGTTCTCCTTTACGAGTAGATATCCAAGAACAACTTTTACAGGGAGACCGTAAAACTCAGGTGTGAAAACCCCTTTATCGACAATTTATTTACAGGGTAAGTTCCGAAAAAATGTAAGTGTTGTTTGTGCTGACGTTGTGCGTCAGCGTCGGCGGCAATCGGCTTGTCGACAAAAAAAAAAAGTGCGACTCGCGT
**************************************************...((((...(((......)))...)))).((..((((((..(((((......(((..(((((((...........((((...................)))))))))))))).....)))))..))))))..)).(((((.....)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR1106729 mixed whole adult body |
SRR060687 9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................................................................AAGTTCCGAACAACTGTAAGTT........................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................ACCGTAAAACTCAGGTGTGAA................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................TGGTTTCTTCGGCGGGGCCTA............................................................................................................................................................................................................ | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................CGAGTGGTTGCTTCGGTTG.................................................................................................................................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................................................AAGTTCGGAATAAATGTAAGGG........................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............AAAGCGACGAGTGGTTAC.......................................................................................................................................................................................................................... | 18 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................................................AAAAATGTATGGGTTGTTT..................................................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12928:95692-95942 - | dvi_8203 | ACGTCAACGAAATAATTGCGCTGCTCACCAACGAAGCCGCCGCGGAAAAGGTATGTACAAGAGGAAATGCTCATCTATAGGTTCTTGTTGAAAATGTCCCTCTGGCATTTTGAGTCCACACTTTTGGGGAAATAGCTGTTAAATAAATGTCCCATTCAAGGCTTTTTTACATTCACAACAAACACGACTGCAACACGCAGTCGCAGCCGCCGTTAGCCGAACAGCTGTTTTTTTTTTTCACGCTGAGCGCA |
| droMoj3 | scaffold_6359:1415426-1415450 - | ATTTAAATGACATGATTTCATTATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_14853:6722767-6722823 - | CCATTAACGAAATGATTGCTCTGCTCACAGACGAGAGCAATGCGGACATGGTGAGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454072:2438294-2438348 - | ---TCAATTCGCTGGTGGTACTGCTGCGCGATGACACGAATGACGAACAGGTAGATGC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:436377-436431 + | ---TCAATTCGCTCATAACACTGCTGCGCGATGACACAAATGCTGAGCAGGTGGGTGC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_10:370593-370647 + | ---TCAATTCGCTCATAACACTGCTGCGCGATGACACAAATGCTGAGCAGGTGGGTGC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/16/2015 at 08:37 PM