
ID:dvi_7872 |
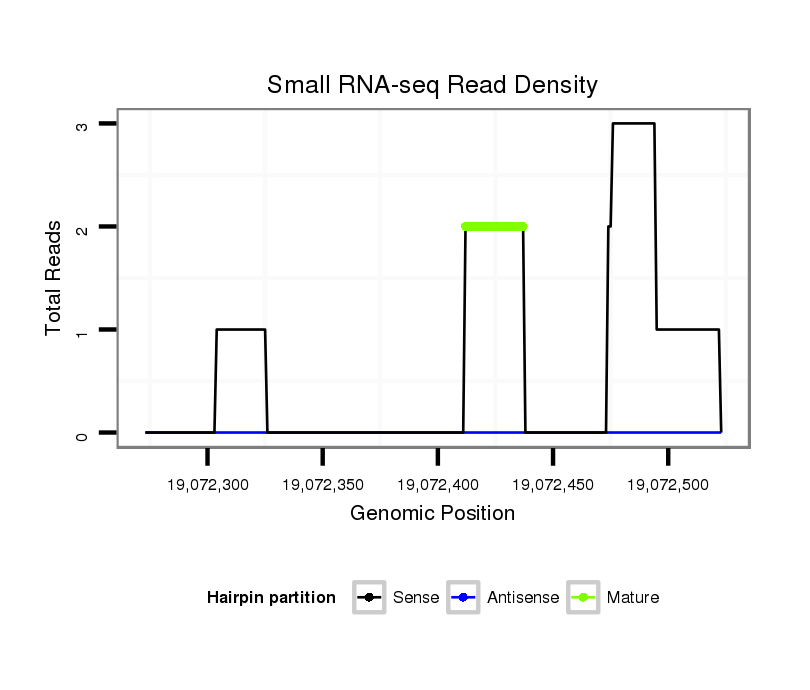
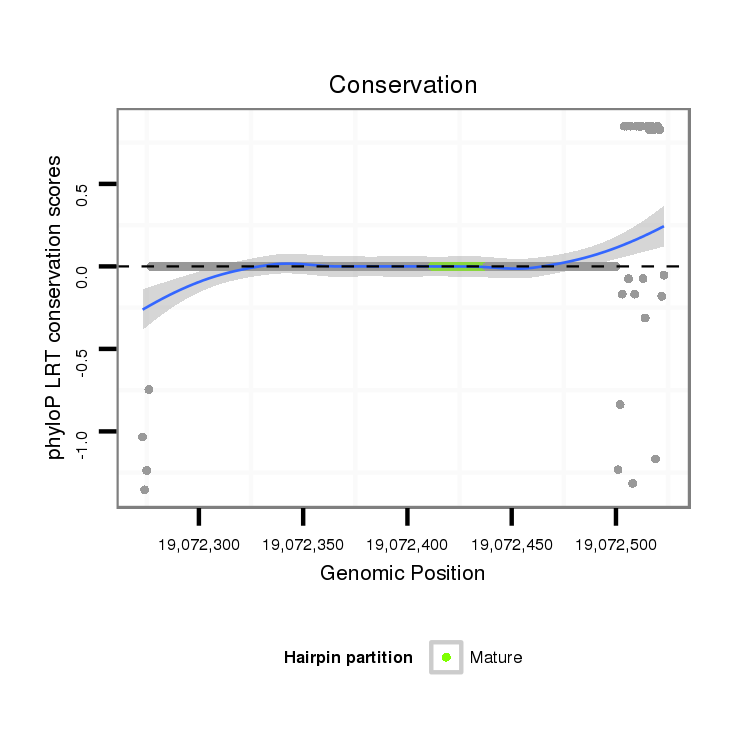
Coordinate:scaffold_12875:19072323-19072473 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -26.7 | -26.6 | -26.6 |
 |
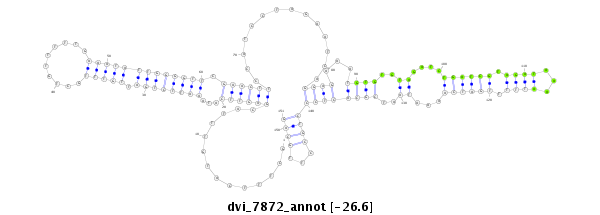 |
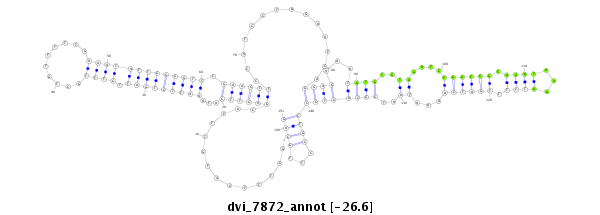 |
exon [dvir_GLEANR_7676:3]; CDS [Dvir\GJ22387-cds]; intron [Dvir\GJ22387-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GCAGGTTCATTGCGTAAATGATCCAATTAACAAGAAGAGTTCTATGCTGGGCTTTCATGTTTACGAGTTCATAAATTCTGATTGTTTGCTGTTTTTGAAATATTGGGGTTCGAACTTCCATCCTAGAATCAGGAGAAATGTGTGTAGATCTGATCGTAGATAAAATTTTTGATCAAAATAATCGCACTCCCTGCCTTGCAGCTGGTCCAGATGAGCTCCGGTGTTGAAAACAATACCAAATCAGTGGACAG **************************************************..............((((((...(((((((((.((((..........))))))))))))).))))))............((((.((...(((((......((((((.((((...)))).)))))).......)))))))))))........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060683 160_testes_total |
SRR060681 Argx9_testes_total |
SRR060679 140x9_testes_total |
V116 male body |
SRR060666 160_males_carcasses_total |
SRR060660 Argentina_ovaries_total |
SRR060668 160x9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
GSM1528803 follicle cells |
M027 male body |
SRR060672 9x160_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................GTGTGTAGATCTGATCGTAGATAAAA...................................................................................... | 26 | 0 | 1 | 2.00 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................CTGGTCCAGATGAGCTCCGGT............................. | 21 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................GTTCGATTCCGGGCTTTC................................................................................................................................................................................................... | 18 | 3 | 20 | 1.50 | 30 | 1 | 0 | 0 | 0 | 16 | 0 | 6 | 4 | 0 | 0 | 0 | 1 | 1 | 1 |
| ...........................................................................................................................................TTGTGTAGATCTGATCGTAGATAAAA...................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................GGTCCAGATGAGCTCCGGT............................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AAGAAGAGTTCTATGCTGGGCT...................................................................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................................GTTGAAAACAATACCAAATCAGTGGACA. | 28 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................GTGTGTAGATCTGCTCGTAGATAAAA...................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................GTAATGTTGGGGTTCTAACT....................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................GATCCAATTATCAAGAAGGGC................................................................................................................................................................................................................... | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................GCAGATCTGATCGTA............................................................................................. | 15 | 1 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................AATTGCACTCCCGGCCTGG..................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGCTGGTGCAGAAGAGCT.................................. | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................GATGTGTGAAGATCTGGTC................................................................................................ | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
CGTCCAAGTAACGCATTTACTAGGTTAATTGTTCTTCTCAAGATACGACCCGAAAGTACAAATGCTCAAGTATTTAAGACTAACAAACGACAAAAACTTTATAACCCCAAGCTTGAAGGTAGGATCTTAGTCCTCTTTACACACATCTAGACTAGCATCTATTTTAAAAACTAGTTTTATTAGCGTGAGGGACGGAACGTCGACCAGGTCTACTCGAGGCCACAACTTTTGTTATGGTTTAGTCACCTGTC
**************************************************..............((((((...(((((((((.((((..........))))))))))))).))))))............((((.((...(((((......((((((.((((...)))).)))))).......)))))))))))........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
M047 female body |
SRR060672 9x160_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060655 9x160_testes_total |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................TTTCTTGGCGTGAGGGGCGG........................................................ | 20 | 3 | 7 | 0.29 | 2 | 0 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................TCTCTAGGCCACAACTTT...................... | 18 | 2 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................CTTCTCTAGGCAACAACTTT...................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................................................................................................TCCTCTAGGCAACAACTTT...................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................AGAATCGTAGTCCTCTTCA................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:19072273-19072523 + | dvi_7872 | GC-AGGTTCATTGCGTAAATGATCCAATTAACAAGAAGAGTTCTATGCTGGGCTTTCATGTTTACGAGTTCATAAATTCTGATTGTTTGCTGTTTTTGAAATATTGGGGTTCGAACTTCCATCCTAGAATCAGGAGAAATGTGTGTAGATCTGATCGTAGATAAAATTTTTGATCAAAATAATCGCACTCCCTGCCTTGCAGCTGGTCCAGATGAGCTCCGGTGTTGAAAACAATACCAAATCAGTGGACAG |
| dm3 | chr2R:5997933-5997959 + | GC-GG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGAAAAAGAAAAAGAGTGCACCA | |
| droEre2 | scaffold_4929:8373528-8373555 - | TGTTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAAAAAACAAAAAGAGTGTACCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:37 PM