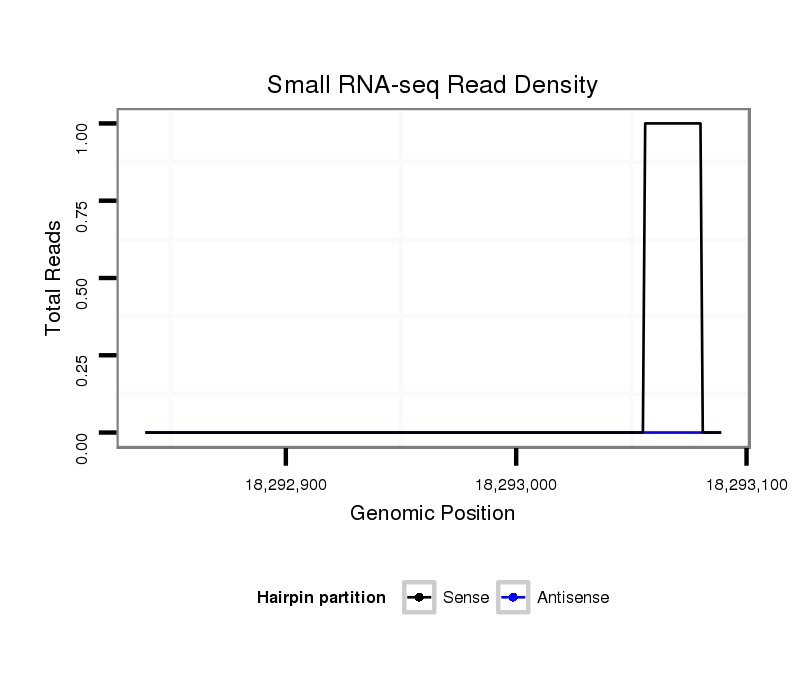
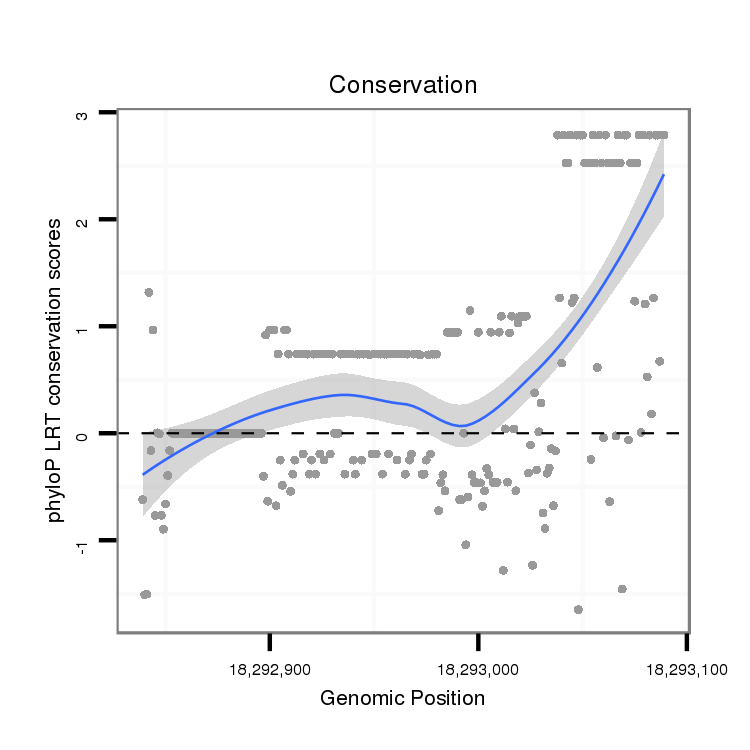
ID:dvi_7713 |
Coordinate:scaffold_12875:18292889-18293039 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ22340-cds]; exon [dvir_GLEANR_7633:3]; intron [Dvir\GJ22340-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AACTGTGCAGGTTCTCAATACCAACAGACAGCTACGATTAACAGAAAACTGCTCTCTAACTTTTATTGATAGGATTAGAAGTTGAGTTAATTTGAATTTAATTAAATTACGAGTAGTTATATTAAACTTGAATCCCTGTTTTGATATTTATTTCTCCTATTATTTGTTTTTAAGTATACTGTAATTAAATTTTTTGCATAGCTGGTTGTGATGGGCACTTCATCGGCCTCCATTCCGGATACGACCATCAA **************************************************...(((.((((((((..........))))))))))).((((((((......))))))))....((((((((.((.(((((((.........(((((...........)))))......))))))).)).))))))))..............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
M061 embryo |
SRR1106729 mixed whole adult body |
SRR060663 160_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR1106720 embryo_8-10h |
SRR060671 9x160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........TTCTCAATACCAACCGACAGC........................................................................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................CTTCATCGGCCTCCATTCCGGATAC......... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................GTTTGCATCGCTGATTGTGAT....................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGCAGGTTGGGCTGGGCACT................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................AGGTTGGGATGGGCACT................................ | 17 | 2 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................TGTTTGCATCGCTGCTTGTGA........................................ | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................................GTTGTATAGCTGGTTGTG......................................... | 18 | 2 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................ATCAGGTTGTGCTGGGCACT................................ | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................AGAATTAGAAGTTGAGCTATT................................................................................................................................................................ | 21 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................................................................................................................................................................GTTATCATGGGCACTTGAT............................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTGACACGTCCAAGAGTTATGGTTGTCTGTCGATGCTAATTGTCTTTTGACGAGAGATTGAAAATAACTATCCTAATCTTCAACTCAATTAAACTTAAATTAATTTAATGCTCATCAATATAATTTGAACTTAGGGACAAAACTATAAATAAAGAGGATAATAAACAAAAATTCATATGACATTAATTTAAAAAACGTATCGACCAACACTACCCGTGAAGTAGCCGGAGGTAAGGCCTATGCTGGTAGTT
**************************************************...(((.((((((((..........))))))))))).((((((((......))))))))....((((((((.((.(((((((.........(((((...........)))))......))))))).)).))))))))..............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
V116 male body |
V047 embryo |
SRR060667 160_females_carcasses_total |
SRR060681 Argx9_testes_total |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
V053 head |
SRR060659 Argentina_testes_total |
SRR060668 160x9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....ATGTCCATGAGTTATAGTT................................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.55 | 11 | 8 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................AGGGACATAGCAATAAATAA................................................................................................... | 20 | 3 | 13 | 0.38 | 5 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............AGTTGTGGTTATCTGTCGA.......................................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................AGGTTAGGCCTATGCTTG..... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................TATAAATAAAGTGAATAATAAA...................................................................................... | 22 | 2 | 13 | 0.15 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| .................................................ACCCGAGATTGAAAATAGCTA..................................................................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................TCTTCAACTCGACTAAATTTAA......................................................................................................................................................... | 22 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............GTTATGGTTTTCTTTCGACGC....................................................................................................................................................................................................................... | 21 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................CTTGAAAATAACCATCCCAA............................................................................................................................................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................................CCTATGATGGTAGTT | 15 | 1 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ................TTATGGTTGTCTGGGGATC........................................................................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................AGGATGGGCCTATGCTGG..... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................ATGTTGCCAGAGGTAAGG............... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:18292839-18293089 + | dvi_7713 | AACTGTGCAGGTTCTCAATACCAACAGACAGCTACGATTAACAGAAAACTGCTCTCTAACTTTTATTG-------------------ATAGGATTAGAAGTTGAGTTAATTTGAATTTAATTAAATTACGAGTAGTTA----------TATTAAACTTGAATCCCTGTTTTG--ATATTTATTTCT-----CCTATTATTTGTTTTTAAGTA--------------------------------------------------TA----CTGTAATTAA---ATTTTTTGCATAGCTGGTTGTGATGGGCACTTCATCGGCCTCCATTCCGGATACGACCATCAA |
| droMoj3 | scaffold_6496:11941346-11941408 + | A-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCTCTTGCACAGCTGGTTGTCATGGGCACTTCGTCGGCTTCCATACCGGACACGACCATCAA | |
| droGri2 | scaffold_15245:4410281-4410400 + | TGATATCCATTCAT--------------------------------------------TCGTTTT--TATCCTAATTTAAATTAGTCAT----------------------------------------------------------------------------------------------CTAATGCCATT---------------------------------------------------------------------------------GAT---ATTTTATATTTAGCTGGTTATAATGGGTACTTCGTCTGCATCGATACCGGATACGACCATCAA | |
| droWil2 | scf2_1100000004510:419531-419582 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATTGGTGGTTATGGGCACTTCGTCGGCCTCAATACCGGACAACAATATCAA | |
| dp5 | 3:7373890-7373941 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTGGTGGTGATGGGCACATCGTCGGCCTCTATTCCGGACACCAACATCAA | |
| droPer2 | scaffold_2:7576272-7576323 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTGGTGGTGATGGGCACATCGTCGGCCTCTATTCCGGACACCAACATCAA | |
| droAna3 | scaffold_13266:2062972-2063024 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAGCTGGTGGTCATGGGCACCTCCTCGGCCTCCATTCCGGACACAACCATCAA | |
| droBip1 | scf7180000395838:55005-55057 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAGCTGGTGGTCATGGGCACCTCCTCGGCCTCCATTCCGGACACAACCATCAA | |
| droKik1 | scf7180000302471:1451581-1451632 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTGGTGGTGATGGGCACCTCGTCCGCCTCTATACCGGACACCAACATCAA | |
| droFic1 | scf7180000454066:1936728-1936812 + | TTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACTA--------------------------------------------------TA----AAGTAATTTATTTACTTCCTCTGCAGCTGGTGGTCATGGGAACCTCGTCGGCTTCCATTCCGGACACCACCATCAA | |
| droEle1 | scf7180000491240:693030-693099 - | TAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAT---GTCTTTTTTGCAGCTGGTGGTCATGGGAACCTCGTCGGCTTCTATTCCGGACACCACCATCAA | |
| droRho1 | scf7180000767854:9552-9603 + | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTGGTGGTGATGGGAACCTCGTCGGCCTCCATTCCGGACACCACCATCAA | |
| droBia1 | scf7180000302143:2667804-2667855 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTGGTGGTCATGGGGACCTCATCGGCCTCGATTCCGGACACCACCATCAA | |
| droTak1 | scf7180000415379:43065-43361 - | AACTGTCTTACAAC--------------------------------------------ACTTTTGTAGACCTTAGTTTTTGTTGGGCATACTTTTAAAATATTAATAAACT---ATGTAAAGAATTTACAAATATTTGTAAATAATATTATAAAAATAAAATCAAAGCTTTATAGTTTTTATTTTTCTGCCACTATTACTAATTTTTAAATTGGCAGAATGAAATAAATAATGGTTTCTAATTGAAAACTAGAAATTGACAGTACCTTATGTAATTTCTCTACTCCAATCTTAGCTGGTGGTCATGGGAACCTCATCAGCCTCGATTCCCGATACCACCATCAA | |
| droEug1 | scf7180000409672:5943540-5943593 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGCTGGTGGTCATGGGTACCTCGTCCGCCTCCATACCGGATACCACCATCAA | |
| dm3 | chr2R:15215774-15215896 - | TTTT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------T--ACATTTATTCCT-----TTTGAAATGTGCTAAAAAGC----------------------------------------------AACAG-ACCAGAAGTAATTTCTGTAATCCCCTTGCAGCTGGTGGTGATGGGCACCTCATCAGCCTCGATTCCGGACACCACCATTAA | |
| droSim2 | 2r:15867876-15867927 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTGGTGGTGATGGGCACCTCATCAGCCTCGATTCCGGACACCACCATTAA | |
| droSec2 | scaffold_1:12729424-12729475 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTTGGTGGTGATGGGCACCTCATCAGCCTCGATTCCGGACACCACCATTAA | |
| droYak3 | 2R:13961422-13961488 + | CT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTAATCCCATTGCAGCTGGTGGTGATGGGCACCTCATCAGCCTCGATTCCGGACACCACCATTAA | |
| droEre2 | scaffold_4845:9413668-9413731 - | TAAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCCTTGCAGCTGGTGGTGATGGGCACCTCATCGGCCTCGATTCCGGACACCACCATCAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:39 PM