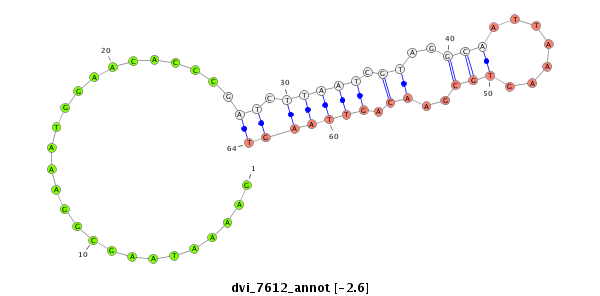
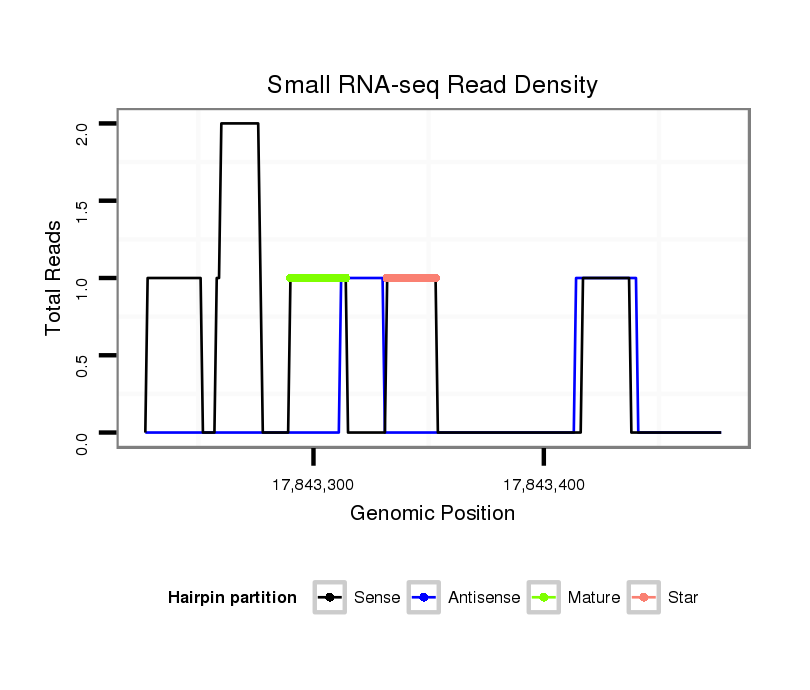
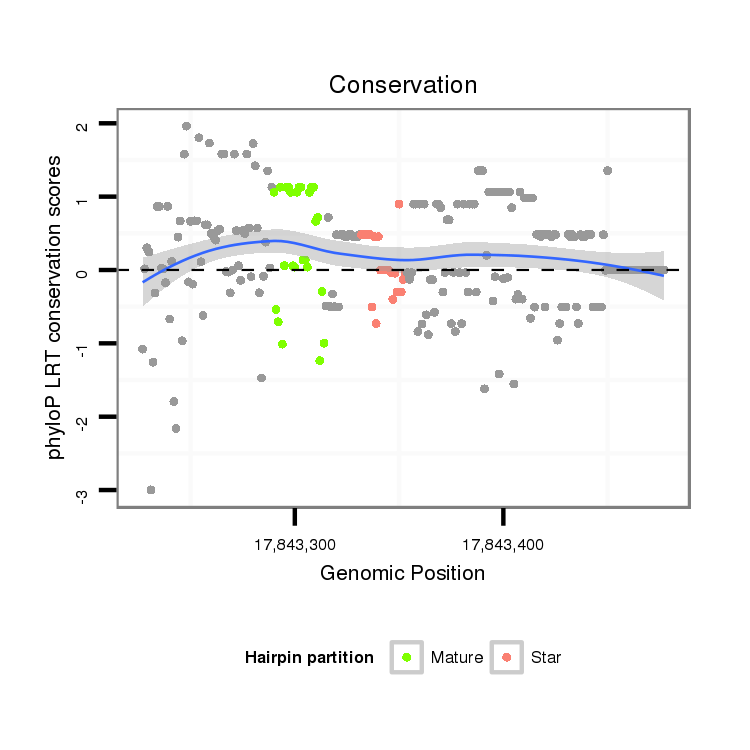
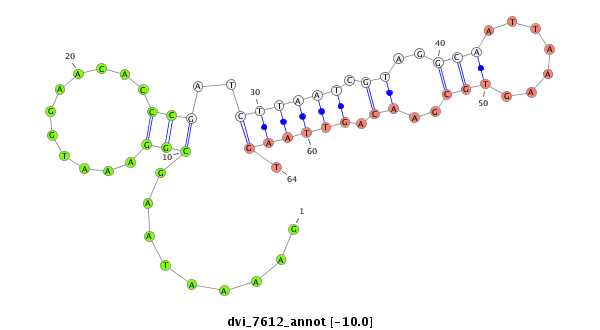
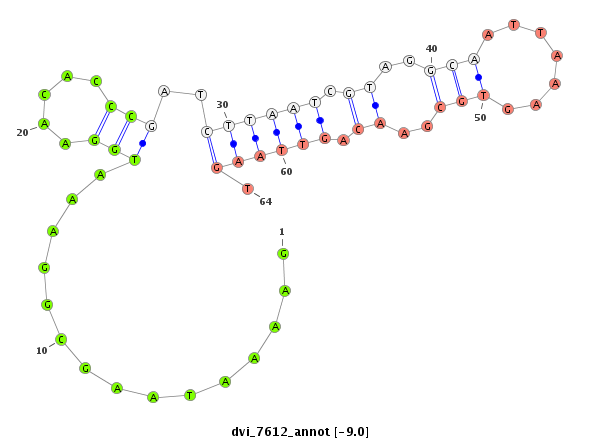
ID:dvi_7612 |
Coordinate:scaffold_12875:17843277-17843427 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -10.0 | -9.0 |
 |
 |
CDS [Dvir\GJ22317-cds]; exon [dvir_GLEANR_7612:1]; intron [Dvir\GJ22317-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CAGCGCTGCTGTTAGTTCTGCTGGTGGCCGCCACCCAAGCAGCTGAAGAGGTAAGTGCCAATTGAAAATAAGCGGAAATGGAACACCCGATCTTAATCGTAGGCAATTAAAGTGCGAACAGTTAAGTGGGAAGACAATTTTAACAGAATTGAGTTGAAATAAAACAAATAATACATTTCTAAAAATAACATGACAAGAACTAAGAGAGGCTGGAATGCAACAGATCGCTAGCATAACAGAGCTGTATACGC ***************************************************************..........................((.(((((.((..(((.......)))..)).)))))))**************************************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060682 9x140_0-2h_embryos_total |
M061 embryo |
SRR060663 160_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
V116 male body |
SRR060656 9x160_ovaries_total |
SRR060668 160x9_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060655 9x160_testes_total |
SRR060667 160_females_carcasses_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................GAACACTCGGTCTTATTCGT....................................................................................................................................................... | 20 | 3 | 4 | 1.50 | 6 | 0 | 1 | 0 | 0 | 0 | 2 | 1 | 0 | 0 | 1 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................TGACAAGAACTAAGAGAGGCT........................................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .AGCGCTGCTGTTAGTTCTGCTGGT.................................................................................................................................................................................................................................. | 24 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................CACCCAAGCAGCTGAAGAG......................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................GAAAATAAGCGGAAATGGAACACCC................................................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................ATTAAAGTGCGAACAGTTAAGT............................................................................................................................ | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................CCCAAGCAGCTGAAGAGG........................................................................................................................................................................................................ | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................ATGGGAAGACAACTTTAAC........................................................................................................... | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................TTAATCGTAGGAAATTAAATG.......................................................................................................................................... | 21 | 3 | 12 | 0.25 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................ATGCAAGAGATCGATAGCCT................. | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................ACTCAAGTGGCTGAAGAGGT....................................................................................................................................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................CTCAAGTGGCTGAAGAGGT....................................................................................................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................AACACTCGGTCTTATTCGT....................................................................................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................GTTAGCTGAATTGAGTTGAA............................................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GTCGCGACGACAATCAAGACGACCACCGGCGGTGGGTTCGTCGACTTCTCCATTCACGGTTAACTTTTATTCGCCTTTACCTTGTGGGCTAGAATTAGCATCCGTTAATTTCACGCTTGTCAATTCACCCTTCTGTTAAAATTGTCTTAACTCAACTTTATTTTGTTTATTATGTAAAGATTTTTATTGTACTGTTCTTGATTCTCTCCGACCTTACGTTGTCTAGCGATCGTATTGTCTCGACATATGCG
****************************************************************************************************************************..........................((.(((((.((..(((.......)))..)).)))))))*************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060675 140x9_ovaries_total |
SRR060679 140x9_testes_total |
SRR060657 140_testes_total |
V053 head |
M027 male body |
SRR060667 160_females_carcasses_total |
SRR060671 9x160_males_carcasses_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................................................TGTACTGTTCTTGATTCTCTCCGACCT..................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................GGGCTAGAATTAGCATCCG................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................ACCCTGTGGGCTACAAGTA.......................................................................................................................................................... | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................GGCCACCGGGGGGGGGTTC.................................................................................................................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................CGTTGATTTCATGGTTGTCA................................................................................................................................. | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................AGAATTCGCATCTGTTGATT............................................................................................................................................. | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................................................................................................................................TCTAAAGATTTTTATTGGA............................................................ | 19 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................TTCCTCGACTTCTCCAATTA................................................................................................................................................................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:17843227-17843477 + | dvi_7612 | CAGCGCTGCTGTTAGTTCTGCTGGTGGCCGCCACCCAAGCAGCTGAAGAGGTAAGTGCCAATTGAAA------ATAAGCGGAAATGGAACACCCGATCTT----AATCGT-AGGCAATTAAAGTGCGAACAGTTAAGTGGGAAGA-----------------CAATTTTAACAGAATTGAGTTGAAATAAAACAAATAATACATTTCTAAAAATAA---C---------------ATGACAA----GAACTAAGAGAGGCTGGAATGCAACAGATCGCTAGCATAACAGAGCTGTATACGC |
| droMoj3 | scaffold_6496:11467233-11467492 + | CAGCAATGCTGTTAGTTCTGCTGGCGGCCGCCACCCAAGCAGCTGATGAGGTAAGTGGCAATTGACA------CTAAGCGGAAATGGAACAGCAGAGAGTGTAGGATCGTCAGGCAATTAAGGGG-----AATCAGGTGGGAATAGATCACAATAGGCGAAAGACCTTGAACGG--CTTAGCTGGAATGAAAGGAATGAGAAAAT-GTAAAAATGATTAAAGAAAGGCATCAACAATGATAATAAAGAAGGGGAAGAGAATGGAATATGGTAGA--------------------------- | |
| droGri2 | scaffold_15245:13847828-13847926 - | CAGCGTTGCTGTTAATTCTGCTGGTGGCCGCCACCCAAGCAGCTGATGAGGTAAGTGATAAATGGAATGGGATTTAAGTGGAAATGGAACATATCATCT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004954:4661824-4661896 - | ATGGTAGGCAATT--------TAATGGCAAAA--------------------------------------------------------------------------------------------------GAATAATAATAAAAA-----------------CAAGGGGAACAAAAACAAAATAAAATAAAATA------------------------------------------------------------------------------AA--------------------------- | |
| dp5 | XR_group6:9435604-9435631 + | A-GCGCTGCTGCAGGCGCCA----CGGCCACCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_4:3402543-3402603 + | CAGCACTGCTGATGAGCCTTCTAGTGGCCGCCACCCATGCCGTAGATCAGGTAAGTCTCAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBip1 | scf7180000395751:985992-986051 - | TCGCCATGCTGATGGGTCTTCTGGTGGCCTCCATCCAAGCCATCGATCAGGTGAGTCCCA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302411:632882-632937 - | GCGCCCTGCTAATCGGAATTCTGGTGGCCACCACCCAAGCCCAAGATCAGGTGAGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453851:85848-85928 - | CAGCA-GGCTGTTTGTCATACTTGCAACAGCCACAGCAGCAGCAGCAGCAGCTA-CATACATTGCCA------AGAAGCTGAAGCTGAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000490644:260057-260068 - | AACCGCCGCCGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chrUextra:3243083-3243115 + | AATA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAACAAATAACACATTTCAAATAATTA---C-----------------------------------------------------A--------------------------- | |
| droEre2 | scaffold_4770:3214617-3214660 + | AATAAAAATGATTCGTTATG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATAAAAAATACATTTTTGAA-------------------------------------------------------------AA--------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 02:32 PM