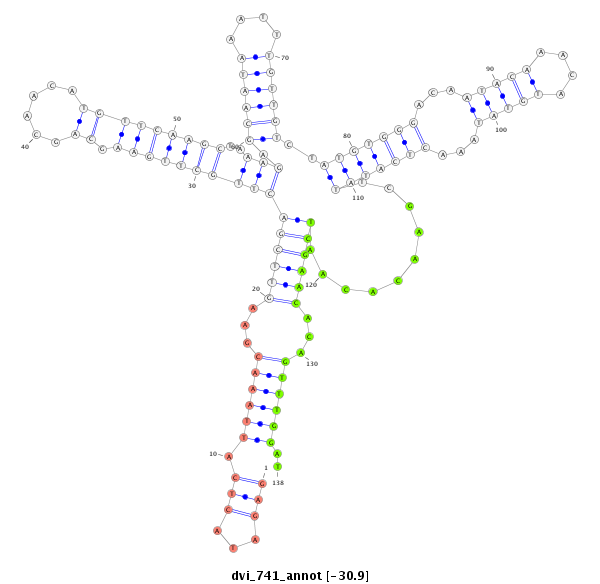
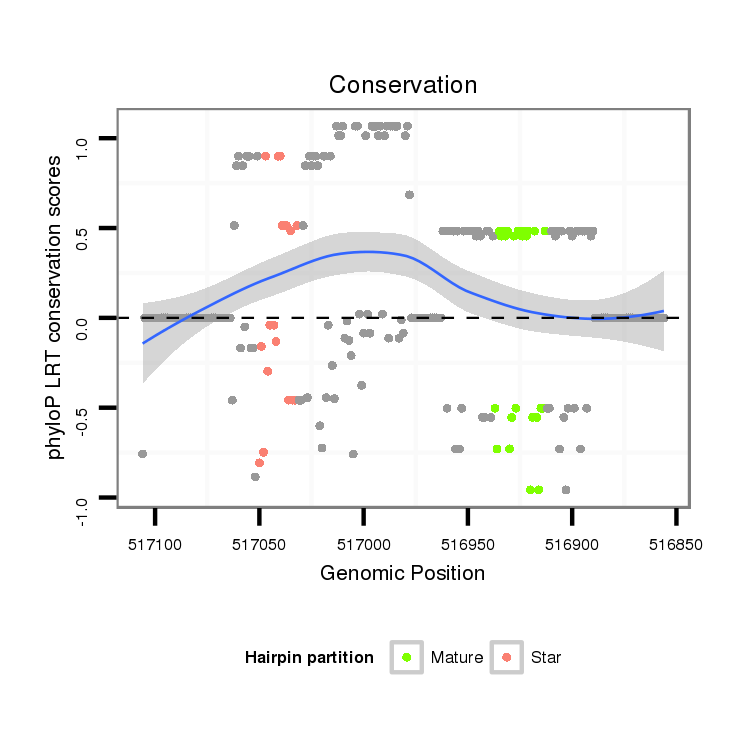
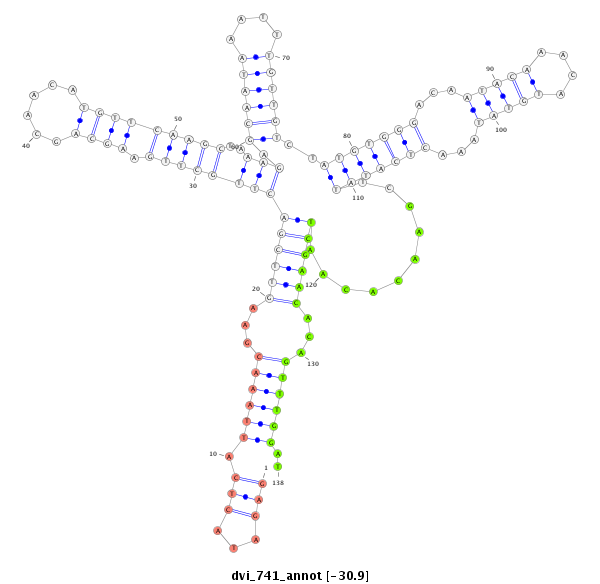
ID:dvi_741 |
Coordinate:scaffold_10324:516906-517056 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -30.9 | -30.9 | -30.7 |
 |
 |
 |
exon [dvir_GLEANR_15304:5]; CDS [Dvir\GJ14996-cds]; intron [Dvir\GJ14996-in]; intron [Dvir\GJ14996-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------#####--------------------------------------------- TCGAACGTTCTAGTAGCAAACTAAGTTGTGTTCTAACATGGAAGTCAGCAAACCCAGAGATACTCATTAAACGAAGTTCGACTTGCTTGAAGCAGCAACATGTTCAAGCTAAAGAGCAATAAATTTGTTGTCTATGTGGGACAATACAAACATGTATAAACTCATATTCGAACACAATCGAACACAGTTTGGATCGAACAGAAGTAGTAAGAAGTGTATTGCCTGTCTGCAATTCGTACGGCTCGATCCAA ********************************************************(((...))).((((((...((((((((((((((((.((......)))))))))..))).((((((....))))))..(((((((...(((((....)))))...)))))))..........))))))...))))))..********************************************************* |
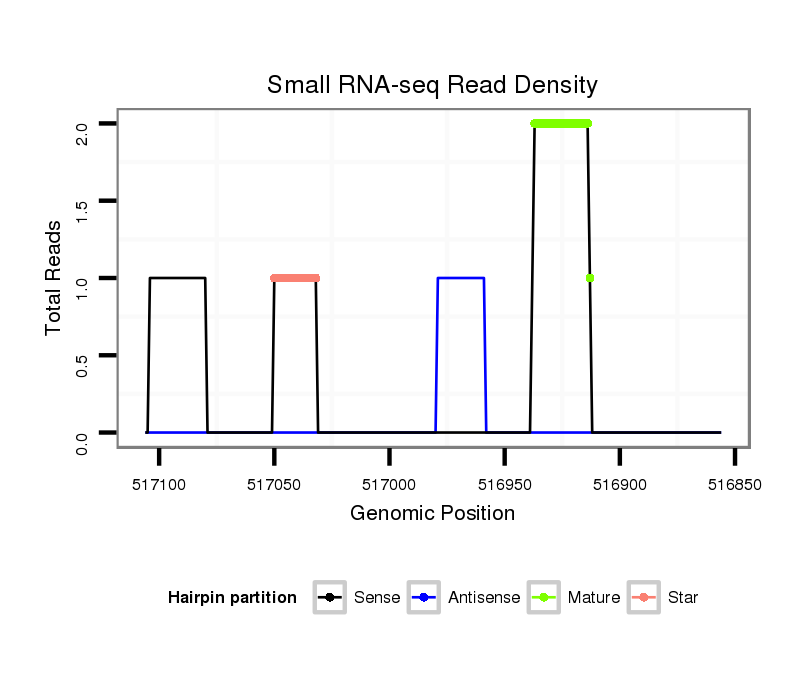
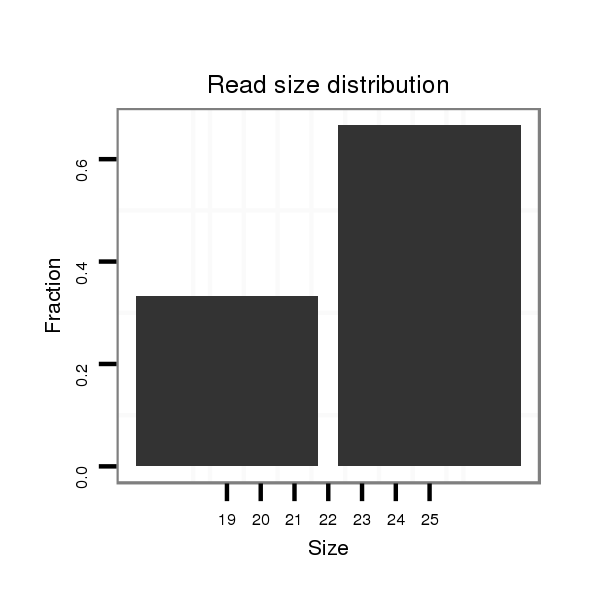
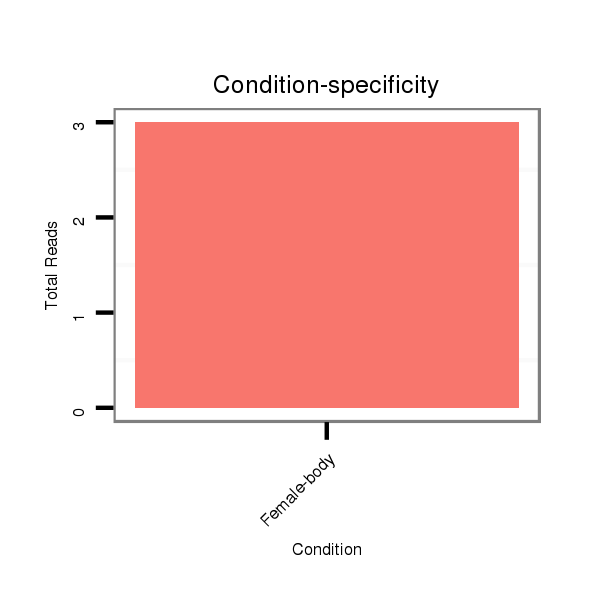
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
V116 male body |
SRR060669 160x9_females_carcasses_total |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060664 9_males_carcasses_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
V047 embryo |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................GTTCTACCATGGAAGTCTGC.......................................................................................................................................................................................................... | 20 | 2 | 1 | 3.00 | 3 | 0 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................GTGTTCTACCATGGAAGTCT............................................................................................................................................................................................................ | 20 | 2 | 2 | 1.50 | 3 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................GAACAGAAGTAGTAAGCAGT.................................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................TGTTCTACCATGGAAGTCTGC.......................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..GAACGTTCTAGTAGCAAACTAAGTT................................................................................................................................................................................................................................ | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................GAACACAATCGAACACAGTTTGGAT......................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................GTGTTCTACCATGGAAGTCTGC.......................................................................................................................................................................................................... | 22 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................GAGATACTCATTAAACGAA................................................................................................................................................................................ | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................CGAACACAATCGAACACAGTTTGGA.......................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................GTTCTACCATGGAAGTCTGCT......................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......GTTCTAGTAGTAAACTATG.................................................................................................................................................................................................................................. | 19 | 2 | 5 | 0.40 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................TTGTGTGCTACCATGGAAG............................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AAAAGTCGTAACAAGTGTATT............................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................CTTGAAGCAGCACCCTGTGC.................................................................................................................................................. | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................CAACGTGTTCAAGCTAAG.......................................................................................................................................... | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................AACACAGAGATACCCATT....................................................................................................................................................................................... | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............................GTTCTACCATGGAAGTCTG........................................................................................................................................................................................................... | 19 | 2 | 17 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................GTACTACCATGGAAGTCAGA.......................................................................................................................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............................GTACTACCATGGAAGTCAGCC......................................................................................................................................................................................................... | 21 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
AGCTTGCAAGATCATCGTTTGATTCAACACAAGATTGTACCTTCAGTCGTTTGGGTCTCTATGAGTAATTTGCTTCAAGCTGAACGAACTTCGTCGTTGTACAAGTTCGATTTCTCGTTATTTAAACAACAGATACACCCTGTTATGTTTGTACATATTTGAGTATAAGCTTGTGTTAGCTTGTGTCAAACCTAGCTTGTCTTCATCATTCTTCACATAACGGACAGACGTTAAGCATGCCGAGCTAGGTT
*********************************************************(((...))).((((((...((((((((((((((((.((......)))))))))..))).((((((....))))))..(((((((...(((((....)))))...)))))))..........))))))...))))))..******************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
SRR060678 9x140_testes_total |
SRR060658 140_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
V047 embryo |
SRR060659 Argentina_testes_total |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................................................................................................................CGTAACGGACAGACGATAAGGAT............. | 23 | 3 | 1 | 2.00 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GTAACGGACAGACGATAAGGAT............. | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................AACAGATACACCCTGTTATGT....................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................TAACGGACAGACGATAAGGATT............ | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................TAACGGACAGACGATAAGGAT............. | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................CCCGTAACGGACAGACGATAAG................ | 22 | 3 | 2 | 1.00 | 2 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GTAACGGACAGACGATAAG................ | 19 | 2 | 3 | 0.67 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................AACTTGTGTTAGCTGGTG.................................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................CATAACTGACGGACGTTAA................. | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................TCTCGTTATTGAAACAAAA........................................................................................................................ | 19 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................TCGTCATTGAAAGAACAGAT..................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................................................................................................................................................................................................................................ATGTTAAGCTTGCCGCGCT..... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_10324:516856-517106 - | dvi_741 | TCGAACGTTCTAGTAGCAAACTAAGTTGTGTTCTAACATGGAAGTCAGCAAACCCAGAGATACTCATTAAACGAAGTTCGACTTGCTTGAAGCAGCAACATGTTCAAGCTAAAGAGCAATAAATTTGTTGTCTATGTGGGACAATACAAACATGTATAAACTCATATTCGAACACAATCGAACACAGTTTGGATCGAACAGAAGTAGTAAGAAGTGTATTGCCTGTCTGCAATTCGTACGGCTCGATCCAA |
| droMoj3 | scaffold_6496:7142627-7142700 - | A-----------------------------------------------------------------------------------------------------------------------------------------------TATAAAAAGATATAAACTCTAATACACACACACACAAACACACATACAGTTAAACATATCCAGCAATAAATGT---------------------------------- | |
| droGri2 | scaffold_15126:105836-105885 + | A------------------------------------------ATCAACAAATTTATGCACACTCATTAAGCAGAACTCTACTTGACTTAAG--------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004540:816155-816228 + | T--------------------------------------------CAGCTAACCGAAGGATGTCAAT-----------CGACTTGAATGGACAAGCATTTTCTTCCGGCCAAAGAGCATTAAAATCGTT-------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491046:1704374-1704409 + | C--------------------------------------------------------------------------------------------AGCAACTGATTTAAGTTAAAGAACAATAAATCTGT--------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
Generated: 05/16/2015 at 08:51 PM