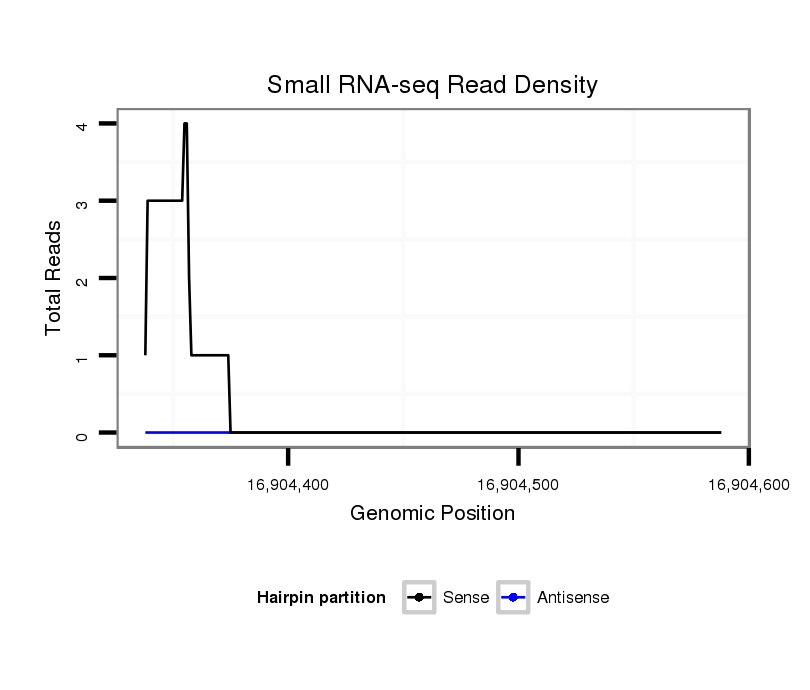
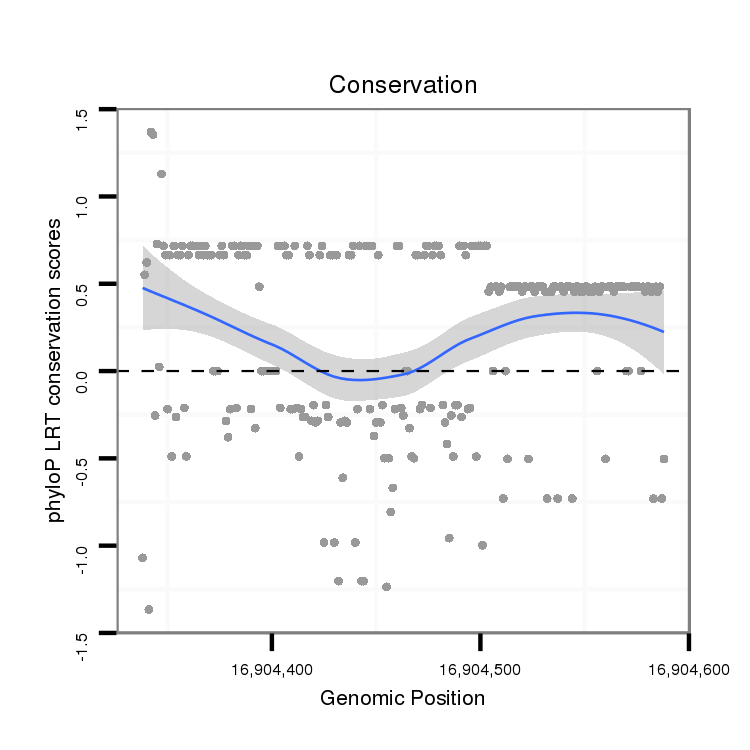
ID:dvi_7368 |
Coordinate:scaffold_12875:16904388-16904538 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_7544:1]; CDS [Dvir\GJ22242-cds]; intron [Dvir\GJ22242-in]
No Repeatable elements found
| --------------------------------------------######--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GAAGAATCGTTGAGCTAGCAAAGTTATGAGAGCCAATGACAACAATGAAGGTGAGTTTGCGTTCGTATTGCATTTGGTTTGTGGACAGCTGGCGGGGAGGGAGATGGAAATCCCCCTCTGGGTTTTCCCATGCCGGTTAGAAAGCGAATGGCATTCAATTTAAGATCTCTTCATTTTAAATTGAATAACAAAAGCCGATATTCAATTGATCGATGCGAACAAATTTCAATTTCCAAATATTCAAGCTCTGG **************************************************.........(((((((.((....(((((.((.(((.((((..(((((.(((.(((....))))))))))))))).))))).)))))....)).)))))))..((((((((((((((......))))))))))))))...............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M061 embryo |
SRR060665 9_females_carcasses_total |
SRR060679 140x9_testes_total |
V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
M047 female body |
V116 male body |
M027 male body |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .AAGAATCGTTGAGCTAGC........................................................................................................................................................................................................................................ | 18 | 0 | 1 | 2.00 | 2 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................GCAAAGTTATGAGAGCCAAT...................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| GAAGAATCGTTGAGCTAGCA....................................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................TAGAAAGGGAAGGGCATT................................................................................................ | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................GATATTCAATGGATTGAGGC................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................TGTGGACTGCTGGTGGGG.......................................................................................................................................................... | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................TTGAAAGCGAAGGGCATT................................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................AGTGCCAATGATAACACTGA........................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................GAAAGCGAAGGGCATTTGAT............................................................................................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CTTCTTAGCAACTCGATCGTTTCAATACTCTCGGTTACTGTTGTTACTTCCACTCAAACGCAAGCATAACGTAAACCAAACACCTGTCGACCGCCCCTCCCTCTACCTTTAGGGGGAGACCCAAAAGGGTACGGCCAATCTTTCGCTTACCGTAAGTTAAATTCTAGAGAAGTAAAATTTAACTTATTGTTTTCGGCTATAAGTTAACTAGCTACGCTTGTTTAAAGTTAAAGGTTTATAAGTTCGAGACC
**************************************************.........(((((((.((....(((((.((.(((.((((..(((((.(((.(((....))))))))))))))).))))).)))))....)).)))))))..((((((((((((((......))))))))))))))...............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR060658 140_ovaries_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060659 Argentina_testes_total |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060688 160_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................AGGGTAGTGTCAATCTTTCG.......................................................................................................... | 20 | 3 | 5 | 4.00 | 20 | 6 | 0 | 4 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TTAACTGGCTACGCTTGTTG............................ | 20 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................AGGGTAGTGTCAATCTTTC........................................................................................................... | 19 | 3 | 20 | 0.40 | 8 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 |
| ..........................................................................................................................................................................................................................TGGTGAAAGTTAAAGGTTTA............. | 20 | 2 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................GAACTAGCTACGCTTGGTTC........................... | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TTAACTGGCTACGGTTGTT............................. | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................TGCACTCAAAAGCAAGGATA....................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................AAAGTTAAAGGTTTAGGAG......... | 19 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................AAATTTAAAGGTTGATAAG......... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:16904338-16904588 + | dvi_7368 | GAAGAATCGTTGAGCTAGCAA---AGTTATGAGAGCCAATGACAACAATGAAGGTGAGTTTGCGTTCGTATTGCATTTGGTTTGTGGACAGCTGGCGGGGAGGGAGA--TGGAAATCCCCCT---------------------CTGGGTTTTCCCATGCCGGTTAGAAAGCGAATGGCATTCAATTTAAGATCTCTTCATTTTAAATTGAATAACAAAAGCC-GATAT-TCAATTGATCGATGCGAACA-----------AATTTCAATTTCCAAATATTCAAGCTCTGG |
| droMoj3 | scaffold_6496:11097894-11098160 - | AATC----GTTGAGCTTGCAACGACGTTATGAGAGCC---GACGATAATGAAGGTAATTT--------TATTGCATTTGAAATGCGGGCAACAGGTGAGCGGGGAAAATTAAAAATTCCCCGCTTCATCTTATTGTTGTTTATGGCTATTTC--ACGGCTGGTTAGAAAGCGTGCGGCAATCAATTGAATATCT-TTCAG-CTAAATTGAACAACAAAAGACAGATCTCTCAATGGATCGATGCGA-CAATAATGTGTGAAGTTTCAATTT--AAATA-TCAAGATCTTA | |
| droGri2 | scaffold_15245:16445363-16445510 + | CAAC----TTTGTGGTAGCAG---CGTTATGAGAGCC---GACGCCAACGAAGGTGAGT---------TGTTGCTATCCGTTTGCATGCATTTGGAGCTGGTGGATT--TCCAATTTACATT---------------------TTCTGTTCT--CCTGCCAGTTGGAAAGTTTTTCATATTCTGTTGAAAAT-------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004902:2136692-2136697 - | ACAGAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12929:2412026-2412035 + | AAATAAACGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396434:401963-401965 + | AAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chrX:2486347-2486349 + | AAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:2298701-2298706 + | AAAAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_10:2259576-2259580 + | GAAAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/16/2015 at 11:54 PM