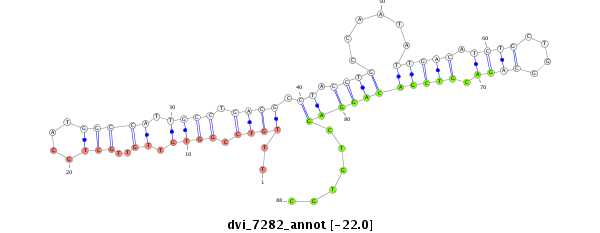
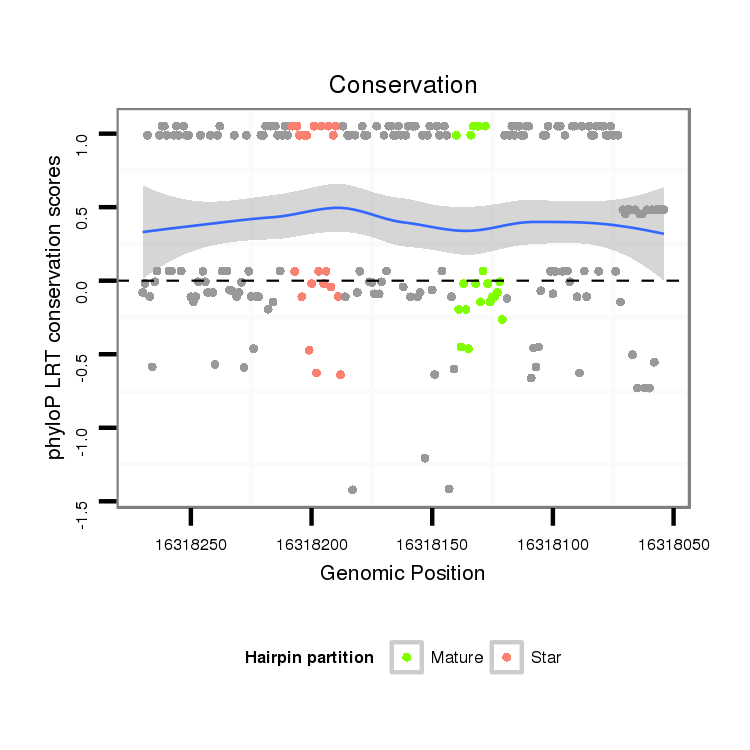
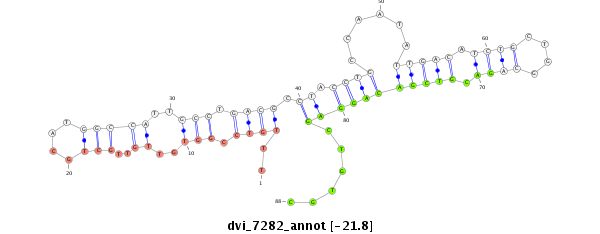
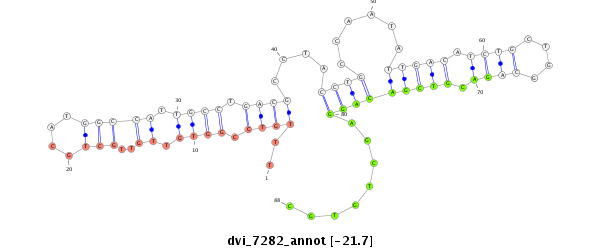
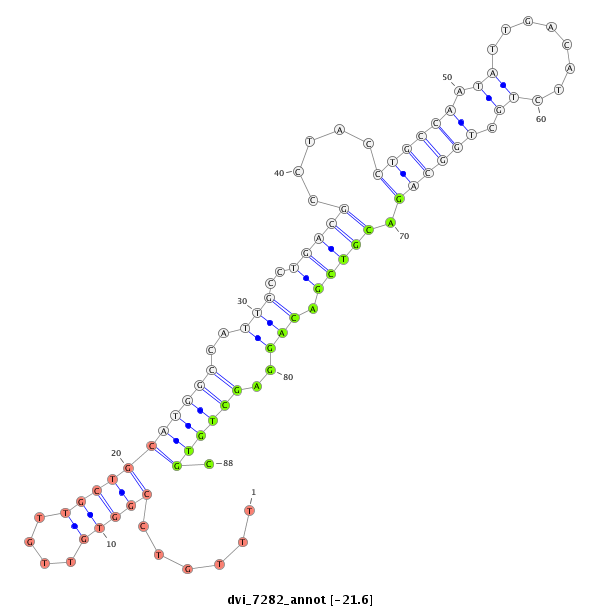
ID:dvi_7282 |
Coordinate:scaffold_12875:16318104-16318220 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -21.8 | -21.7 | -21.6 |
 |
 |
 |
intron [Dvir\GJ20142-in]; CDS [Dvir\GJ20142-cds]; exon [dvir_GLEANR_5597:3]; exon [dvir_GLEANR_5597:2]; CDS [Dvir\GJ20142-cds]; intron [Dvir\GJ20142-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------------------------------------------------------------------##########################------------------------ TGCGGCGCATGCGCAGCTGCAGGCGACACATGAGGCATCCTTGGTGGCGCGTAAGTGCGTCATTTGTCCGGTGTTGTTGCTGCATGGCCATTGCCTGACGCCTACCTGCCAATATTGACATCTGCTGGCAGACGTCGACAGGAGCTGTGCGACTGTACCAACTGCAGGATGTATACGCTTTCAGCTGCTTAAGGTGAGCTGAAGATGGGTTATATAT **************************************************************..((((.((((.((..(((....))))).)))).)))).((.((((......(((((.((((....)))).)))))))))))......******************************************************************* |
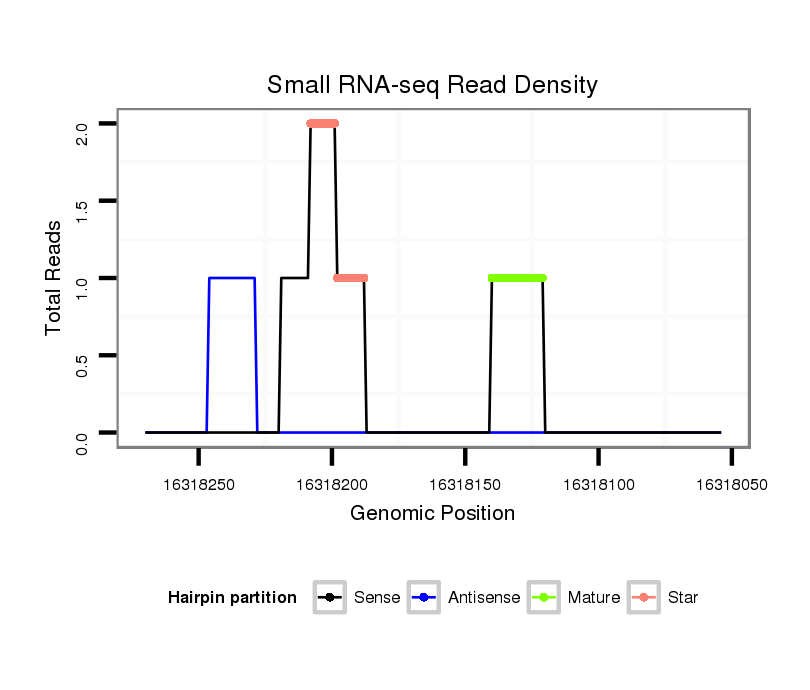
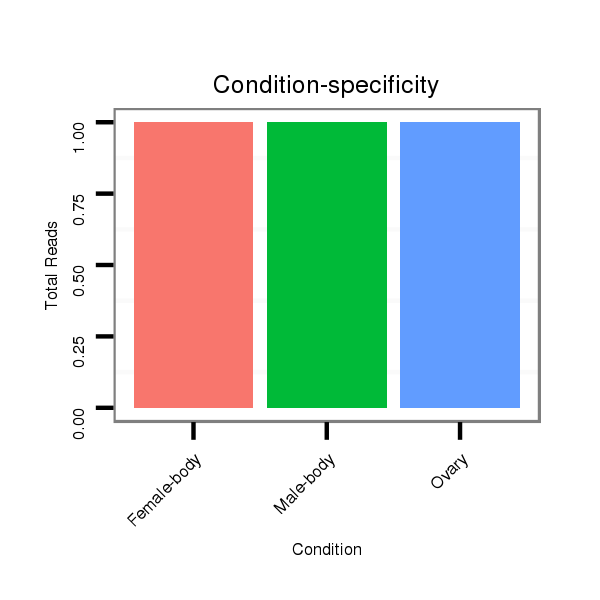
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
V116 male body |
SRR060654 160x9_ovaries_total |
V053 head |
SRR060667 160_females_carcasses_total |
V047 embryo |
SRR060661 160x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
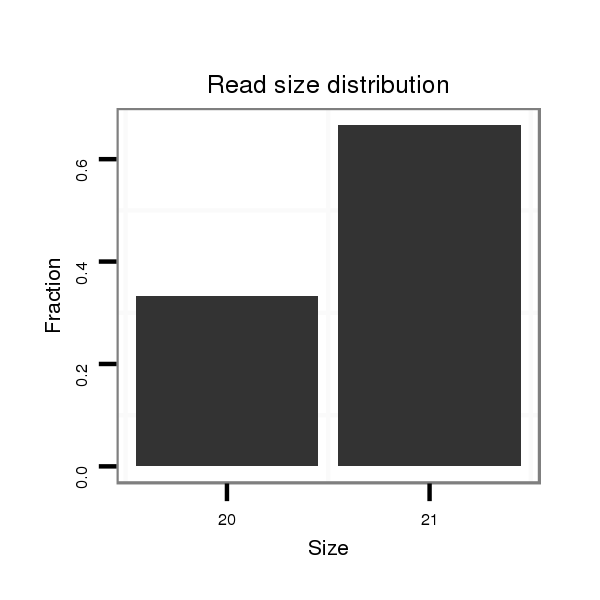
| ..................................................................................................................................GACGTCGACAGGAGCTGTGC................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................TTTGTCCGGTGTTGTTGCTGC...................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................TAAGTGCGTCATTTGTCCGGT................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................AATATTGACATCTGCTGGCG....................................................................................... | 20 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................GCTTAGGCTGAGCTGATGATGG......... | 22 | 3 | 4 | 0.50 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..............................TCAGGCATCGTTGGTGGTGCG...................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................GAGCCATCGTTGGTGACGCG...................................................................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................TGAAGTTGCTGCATGGCCAGT............................................................................................................................. | 21 | 3 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................CTGCTTTGCCATTGCCTGA....................................................................................................................... | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................ACCCATGATGCATGCTTGGT............................................................................................................................................................................ | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................................................................CTGAAGATGGGTTAGGTGT | 19 | 3 | 17 | 0.12 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 1 |
|
ACGCCGCGTACGCGTCGACGTCCGCTGTGTACTCCGTAGGAACCACCGCGCATTCACGCAGTAAACAGGCCACAACAACGACGTACCGGTAACGGACTGCGGATGGACGGTTATAACTGTAGACGACCGTCTGCAGCTGTCCTCGACACGCTGACATGGTTGACGTCCTACATATGCGAAAGTCGACGAATTCCACTCGACTTCTACCCAATATATA
*******************************************************************..((((.((((.((..(((....))))).)))).)))).((.((((......(((((.((((....)))).)))))))))))......************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060656 9x160_ovaries_total |
SRR060663 160_0-2h_embryos_total |
M047 female body |
SRR060681 Argx9_testes_total |
SRR060679 140x9_testes_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................CTGTGTACTCCGTAGGAA............................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................TTCGACACGCTGACATGGTTGA...................................................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................GTCCGCTGTGTACTCAGT.................................................................................................................................................................................... | 18 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................TGTCCTCGACACGCAGAC.............................................................. | 18 | 1 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................................CACTCAACATCTACCCCAT..... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................ACGGACTGCTAATGGATG............................................................................................................ | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:16318054-16318270 - | dvi_7282 | TGCGGCGCATGCGCAGCTGCAGGCGACACATGAGGCATCCTTGGTGGCGCGTAAGTGCGTCATTTGTCCGGTGTTGTTGCTGCATGGCCATTGCCTGACGCCTACCTGCCAATATTGACATCTGCTGGCAGACGTCGACAGGAGC---TGTGCGACTGTACCAACTGCAGGATGTATACGCTTTCAGCTGCTTAAGGTGAGCTGA----------AGATGGGTTATATAT |
| droMoj3 | scaffold_6496:7440062-7440288 + | CACGGCGCATGCGCAGCTGCAAGCGACGCGCGAGGCGACCCTAGTGTCGCGTCAATGCGTCATTTGTCCGATGTTATTGCTGGATGGACGTTGCCTTAAGCCTACCTGCCAATATCGTCACGTGCTGTCAGCTAGAGATAAGAAT---TGCGGGACTGTACCAAGAGTTGGATTTATACGCTTTCAGCTGCTTAAGGTGAGTTGATTTAGGATGCAAAGGGTTGAAATAT | |
| droAna3 | scaffold_13266:17824518-17824718 - | CGCCCAACATGTACAGTTGCAACAGGAGCGGGAAATATCGCGCGCCAGCCGTCAATGTGTCATATGTCCTGTCCTGCTGCTCTATGGACGCTGCAGGACTCTTACCTGCCAATATCGCCATTTGTTGTGTGAAGTTGACAAAAGTGTATCCTCGGCTGTACCAAAGCAAGGACGAATTCAATTTGAAGTGCTCAAGGTGGG----------------------------- | |
| droBip1 | scf7180000396735:1089247-1089448 - | CGCCGAACATGTACAGTTGCGACAGGAGCGGGAAATATCGCGCGCCAGCCGTCAATGTGTCATATGCCCAGTTCTGCTACTCTACGGCCGCTGCAGGACTCTTACCTGTCAGTACCGTCATTTGTTGGGCGAAGTTGACAAAAGTGTACCCTCGACTGTACCAAAGGAAGGACGAATTCAATTCAAAGTGCTCAAGGTGGGC---------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
Generated: 05/16/2015 at 10:07 PM