

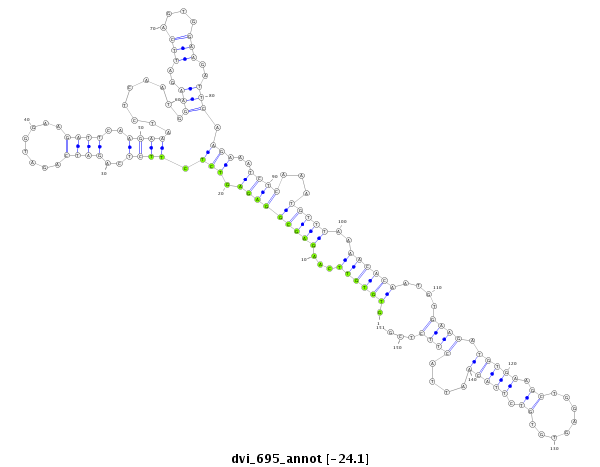
ID:dvi_695 |
Coordinate:scaffold_10324:242043-242193 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
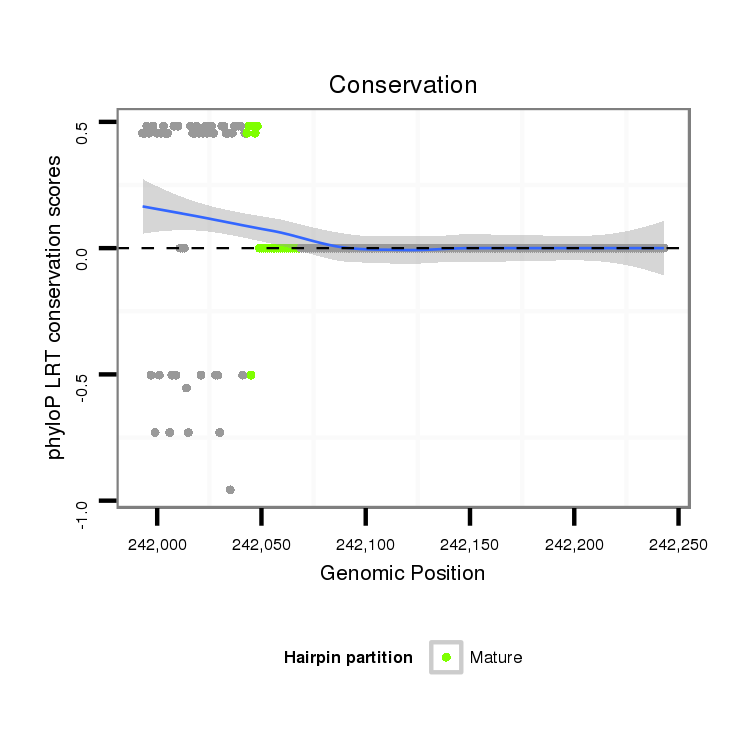
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.1 | -24.1 | -24.1 |
 |
 |
 |
exon [dvir_GLEANR_15360:1]; CDS [Dvir\GJ15058-cds]; intron [Dvir\GJ15058-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCTGCTTGCCAGCGCTCTACTATTGCTGGCTGTAGTTCAAGCCGAAAAAGGTGTGTTCAAGAGCGGAGAGTCTCTTCTCAGATCAGATGGAAGATTCAAGAAATCTCAATGGAAGATTCAGTGGAAGATTCAAGAAATCTCAAATGTTTAAAACACAATGTGAAGATGTGAAGCTGGAGTGTGTCTTACAATTACTTCTCGAGTTAAGTTTTTGTGTATGAGTTCTCAGGAAAGTGTTTACCGAGTTCCTA **************************************************.((((((...(((((((((.(((.((((..((((........))))..)))).........(((..(((....)))..))).)))..))))...)))))..)))))).((.((((.(((((.((........)).)))))....)))).))************************************************** |
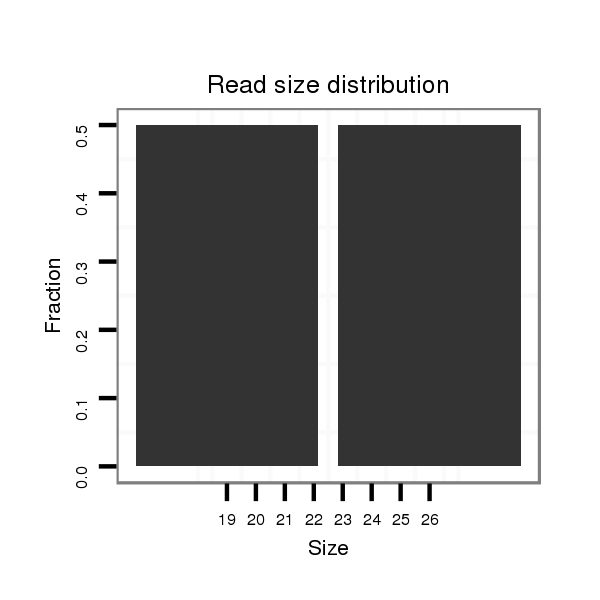
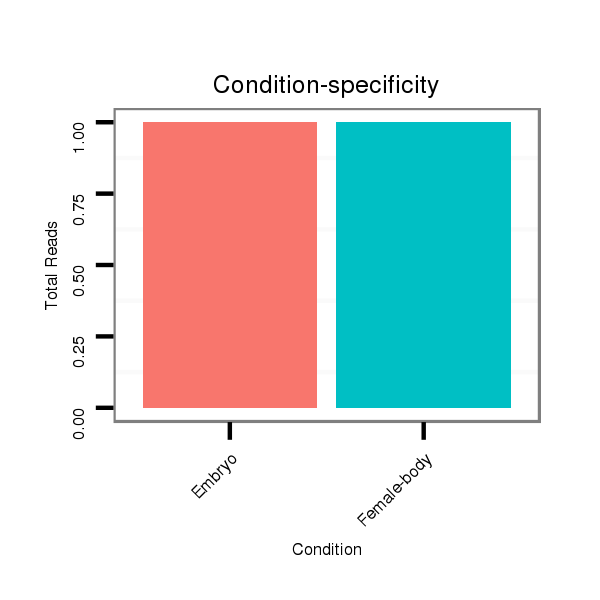
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
M061 embryo |
V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060678 9x140_testes_total |
SRR060683 160_testes_total |
SRR060689 160x9_testes_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
V053 head |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........AGCGCTCTACTATTGCTGGCTGTA......................................................................................................................................................................................................................... | 24 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................CGATTATTGCTGGCTGTAGTTCAAGCC................................................................................................................................................................................................................ | 27 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................GCTGGCTGTAGTTCAAGC................................................................................................................................................................................................................. | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTGTGTTCAAGAGCGGAGAGTCTCTT............................................................................................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................TATTGCTGGCTGTAGTTCAAGC................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................CTACTATTGCTGGCTGTAGTTCAAGCC................................................................................................................................................................................................................ | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............CTCTACTATTGCTGGCTGTA......................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................GTGTTCAAGAGCGGAGAGT.................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................TTCAAGAGAGGAGGGTCTCT................................................................................................................................................................................ | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................CTGTAGTTCAACCCGAAAAA.......................................................................................................................................................................................................... | 20 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................TTTCAAGAGGGGAGGGTCTCT................................................................................................................................................................................ | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................GGAAGGTTAAGTGGAAGA........................................................................................................................... | 18 | 2 | 9 | 0.33 | 3 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................CTGGAAGGTTAAGTGGAAGA........................................................................................................................... | 20 | 3 | 8 | 0.25 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................GATGGAAGATTGAGGCAAT................................................................................................................................................... | 19 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 1 |
| .......................................................TTCAAGAGAGGAGGGTCTCTC............................................................................................................................................................................... | 21 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 |
| .......................................................TTCAAGAGAGGAGAGTCTCTC............................................................................................................................................................................... | 21 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................CACTGATCAGTTGGAAGAT............................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
CGACGAACGGTCGCGAGATGATAACGACCGACATCAAGTTCGGCTTTTTCCACACAAGTTCTCGCCTCTCAGAGAAGAGTCTAGTCTACCTTCTAAGTTCTTTAGAGTTACCTTCTAAGTCACCTTCTAAGTTCTTTAGAGTTTACAAATTTTGTGTTACACTTCTACACTTCGACCTCACACAGAATGTTAATGAAGAGCTCAATTCAAAAACACATACTCAAGAGTCCTTTCACAAATGGCTCAAGGAT
**************************************************.((((((...(((((((((.(((.((((..((((........))))..)))).........(((..(((....)))..))).)))..))))...)))))..)))))).((.((((.(((((.((........)).)))))....)))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
M027 male body |
M028 head |
V116 male body |
SRR060657 140_testes_total |
SRR060679 140x9_testes_total |
SRR060663 160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................CTAGTCTTCCTTCCAAGG......................................................................................................................................................... | 18 | 3 | 20 | 3.60 | 72 | 72 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................ACACATACTCAAGAGTCCTTTCACA.............. | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................TCAAAAACACATACTCAAGAGTCCTT................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................AAAATCACATAGTCAAGGGT....................... | 20 | 3 | 8 | 0.25 | 2 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| .....TCCGGTCGCGAGATGATA.................................................................................................................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................TCTACCCGACCTTCTAAGTT........................................................................................................................................................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................CGTGCTAAGTGCTTTAGA............................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........................................................................................CGTGCTAAGTGCTTTAGA................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................................................................................................................................................................................................................................TCCTTTCAGAAATGGTGC...... | 18 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_10324:241993-242243 + | dvi_695 | GCTGCTTGCCAGCGCTCTACTATTGCTGGCTGTAGTTCAAGCCGAAAAAGGTGTGTTCAAGAGCGGAGAGTCTCTTCTCAGATCAGATGGAAGATTCAAGAAATCTCAATGGAAGATTCAGTGGAAGATTCAAGAAATCTCAAATGTTTAAAACACAATGTGAAGATGTGAAGCTGGAGTGTGTCTTACAATTACTTCTCGAGTTAAGTTTTTGTGTATGAGTTCTCAGGAAAGTGTTTACCGAGTTCCTA |
| droMoj3 | scaffold_6496:2060315-2060367 + | GCTGTTGGTCAGCTTTTT---TGTGCTGACTGTAGCCAAAGCGGAAAAGGGTATGT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
Generated: 05/16/2015 at 08:15 PM