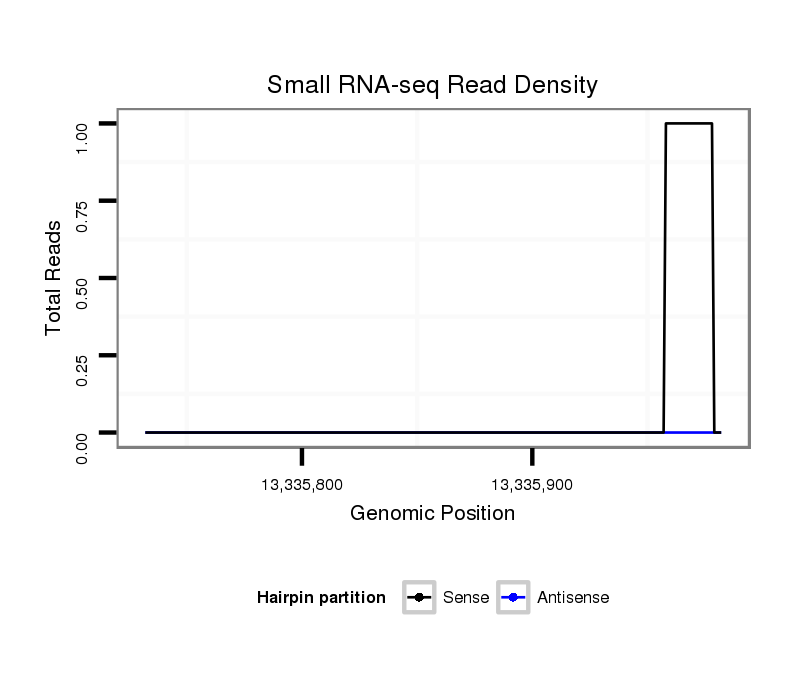
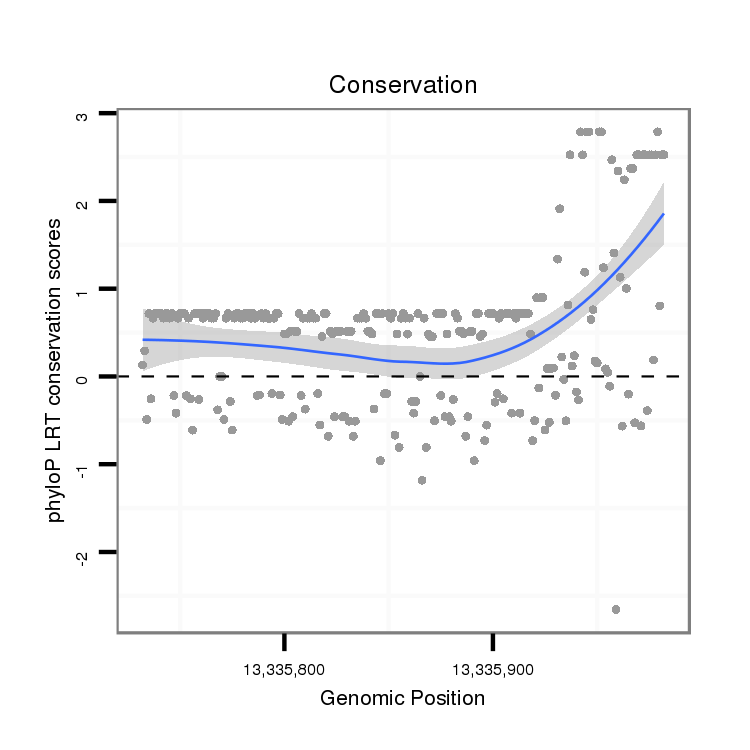
ID:dvi_6659 |
Coordinate:scaffold_12875:13335782-13335932 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_7306:2]; CDS [Dvir\GJ21989-cds]; intron [Dvir\GJ21989-in]
| Name | Class | Family | Strand |
| AT_rich | Low_complexity | Low_complexity | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TCGAACAATTGATGTTAGATTCCTGTAAAGTATCCTATGTCATCGAAGGTAAGTCTTTGACCGGTTTTCCATATATCCCGCTCCCTGATAAGCTAACCATTATCGCACTTCCAATTCCACACTGGACTCTTTTTTCGTGCTAAATCCGTTTGATCCAATTATCTCTTAACCCATTATTTGTTTTATTAAAAATAATAATAGATCAGAGGATCCAAGAACAAGGGAGAAGGTGGCGGGGGTGGTGGCGATGC **************************************************..(((...(((.(((((..(((.............((((((........))))))..........((((...))))..........)))..))))).))).)))..................((((((.(((((....)))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
M047 female body |
SRR060666 160_males_carcasses_total |
V116 male body |
V053 head |
GSM1528803 follicle cells |
M028 head |
SRR060686 Argx9_0-2h_embryos_total |
SRR060658 140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................CGAAGGTAAGTCTATCACCGGT.......................................................................................................................................................................................... | 22 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................GTAAGTCTATCACCGGTTTT....................................................................................................................................................................................... | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................GTTAAGACTTTGACCGGT.......................................................................................................................................................................................... | 18 | 2 | 2 | 1.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................AAGGTGGCGGGGGTGGTGGCG.... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................AGGTAGAAGGTCGCGGGGCT........... | 20 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................AAGGTAGAAGGTCGCGGG.............. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................GGTGGCTGGGGTGGTGGC..... | 18 | 1 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................GGTAGAAGGTCGCGGGGCT........... | 19 | 3 | 9 | 0.22 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ...............................................................................................................................................................................................................................GCGAAGGTGGCGGGATTGG......... | 19 | 3 | 20 | 0.20 | 4 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 1 |
| ....................................................................................................................................................................................................................CAAGTACAAGGTCGAAGGTG................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......AATTCATGTTAGATCCC.................................................................................................................................................................................................................................... | 17 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................TGGCGGGGGAGGTTGCG.... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
|
AGCTTGTTAACTACAATCTAAGGACATTTCATAGGATACAGTAGCTTCCATTCAGAAACTGGCCAAAAGGTATATAGGGCGAGGGACTATTCGATTGGTAATAGCGTGAAGGTTAAGGTGTGACCTGAGAAAAAAGCACGATTTAGGCAAACTAGGTTAATAGAGAATTGGGTAATAAACAAAATAATTTTTATTATTATCTAGTCTCCTAGGTTCTTGTTCCCTCTTCCACCGCCCCCACCACCGCTACG
**************************************************..(((...(((.(((((..(((.............((((((........))))))..........((((...))))..........)))..))))).))).)))..................((((((.(((((....)))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
M047 female body |
SRR060658 140_ovaries_total |
V053 head |
SRR060682 9x140_0-2h_embryos_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................................................................................TTCCATCTTCCAGCGCCC.............. | 18 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................TTTGGGCAAGCTAGGTGAATA......................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................CAAGCTGGGTTAATAAAGAA.................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................GATATATAGGACGAGGGAGT................................................................................................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................AGAAAAATGCAAGATTTA.......................................................................................................... | 18 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............CAATCGAAGGCCATTTGAT........................................................................................................................................................................................................................... | 19 | 3 | 19 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................CCATCCACGAACTGGCCA.......................................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:13335732-13335982 + | dvi_6659 | TCGAACAATTGATGTTAGATTC-CTGTAAAGTATCCTATGTCAT-------------------CGAAGGTAAGTCTTTGACCGGTTTTCCATATATCCCGCTCCCTGATAAGCTAACCAT----TAT--CGCACTTCC---A---------------ATTCCACA--CTGGACTCTTTTTTCGTGCT-AAA--TCCGTTTGATCCAATTATCTCTTAACCCAT-------------------TATTTGTTTTATTAAAAATAAT--AA--TAGATCAGAGGATCCAAGAACAAGGGAGAAGGT------GGCGGGGGTGGTGGCGATGC |
| droMoj3 | scaffold_6496:20632983-20633233 + | TCGAGCAATTGATGTTTGATTTGCCCTATAGTATCCTA--GCACCGTAATGGTCTATCCAGATGGAAGGTAAGTCTTTGACCGGTTTG-------TTCTGCTCCCAGAT-------------------GCGCAC---TATCAAAATTATTAGCACACAATCCACA--ATCGACTCCACT-GCCTGCCCAAA--T----ATG-------AATCTAATAACCCAC-----------------TATATATGTATTATTCGAAATCAA--AATACAGAATAGGCAATCCAAGGCCAAGGGGGAA------------GGTGGCGGTGGCGATGC | |
| droGri2 | scaffold_7485:4049-4325 + | TCAAACAATTGATGTATGATTC-CTGTAAAGTATCCTC--GCAC-------------------CGAAGGTAAGTCACTGACCAGTTCGCCTTGTATCCTGCTCCT--ATCAGTTAATTAAAGATTCACGCGCACTTCTATCAATGTTGCATAGACACACTTTACACCA-CGAC-CCACT-CCC-----AATTTTTCAATTGATCAGATCAT----TAAATCATAATATTAATTATATATATATATATGTATTAT--------AAATAA--CAGAATAGAGGGTCGAAGACAAAGGGAGAAGGT------GGCGGCGGTGGTGGCGATGC | |
| droWil2 | scf2_1100000004822:2211421-2211491 + | TAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACATATT--TG--TAGATACGCACGTCCAAGAGCAAGGGCGATGGCGGTGGTGGCGGCGGAGGGGGAGATGC | |
| dp5 | 3:17488248-17488296 + | AG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGAACAGAGCCTCCAAAGCGAAGGCCGATGGC------------GGCGGCGGTGATGC | |
| droPer2 | scaffold_31:303287-303335 - | AG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGAACAGAGCCTCCAAAGCGAAGGCCGATGGC------------GGCGGCGGTGATGC | |
| droAna3 | scaffold_13266:3712591-3712645 - | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCTGAGGAGCTCCAAGACCAAGGGAGGCGAAGGA---GGAGGCGGAGGCGGCGATGC | |
| droBip1 | scf7180000396427:2178680-2178728 - | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCTGAGGAGCTCCAAGACCAAAG------GAGGT---GAAGGCGGTGGCGGCGATGC | |
| droKik1 | scf7180000302476:1956334-1956388 + | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATGAGGAGCTCCAAGACAAAGACCGAGGGGGGC---GGAGGCGGTGGCGGCGATGC | |
| droFic1 | scf7180000454044:307885-307935 + | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAAAACAAGACCGAGGGAGGC---GGAGGAGGAGGTGGCGATGC | |
| droEle1 | scf7180000491240:176407-176457 - | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGGTCGAAGGCGGC---GGGGGCGGTGGTGGCGATGC | |
| droRho1 | scf7180000780045:26877-26927 - | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGGTCGAAGGCGGC---GGGGGCGGTGGTGGCGACGC | |
| droBia1 | scf7180000301754:647229-647279 - | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGGCCGAGGGCGGC---GGAGGTGGCGGTGGCGACGC | |
| droTak1 | scf7180000415391:192608-192658 - | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACGTCCAAGAACAAGACCGAAGGTGGT---GGAGGAGGTGGCGGGGATGC | |
| droEug1 | scf7180000409474:1083383-1083433 - | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGACCGATGGCGGT---GGAGGTGGTGGTGGCGATGC | |
| dm3 | chr2R:16868815-16868865 + | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGACAGATGGCCCC---GGCGGTGGTGGCGGCGATGC | |
| droSim2 | 2r:17426107-17426161 + | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCTGAGGACATCCAAGAACAAGACAGATGGCCCC---GGCGGTGGTGGCGGCGATGC | |
| droSec2 | scaffold_9:199525-199579 + | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCTGAGGACATCCAAGAACAAGACAGAAGGCCCC---GGCGGTGGTGGCGGCGATGC | |
| droYak3 | 2R:12328226-12328276 - | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGACAGAAGGCGCT---GGGGGTGGTGGCGGCGATGC | |
| droEre2 | scaffold_4845:11012336-11012386 + | GA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACCTCCAAGAACAAGACAGATGGCGCT---GGGGGTGGTGGCGGCGATGC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:30 PM