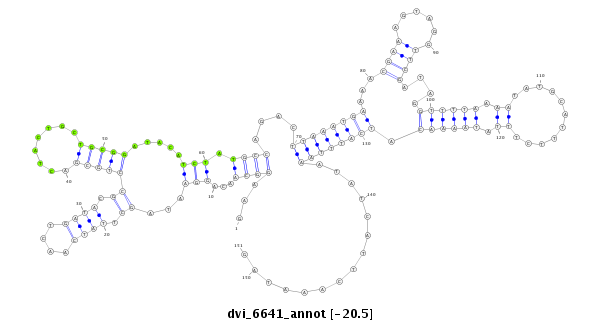
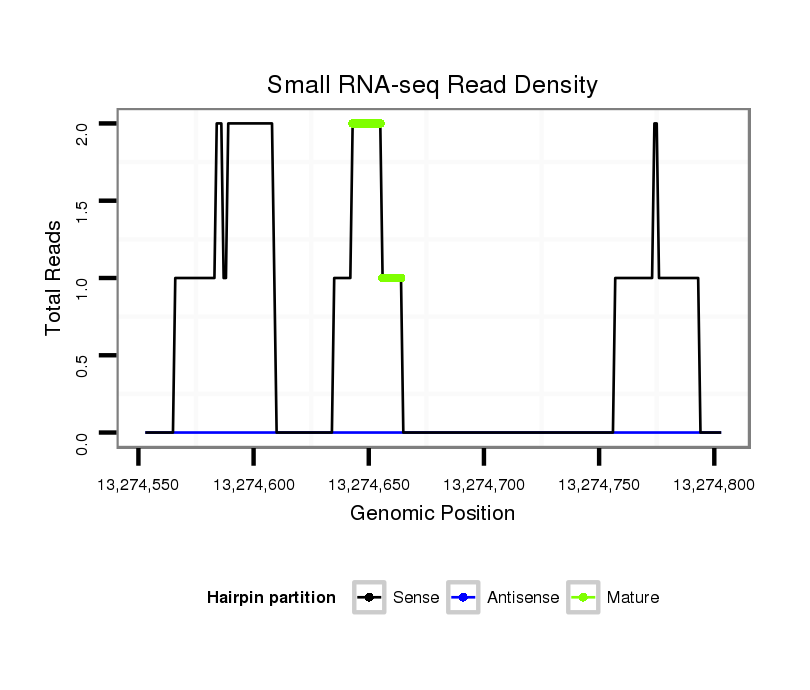
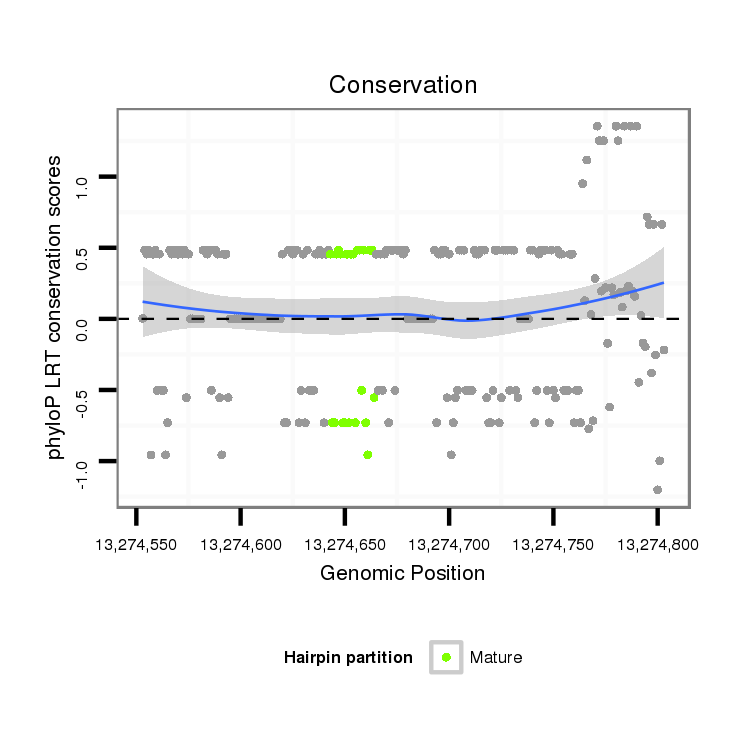
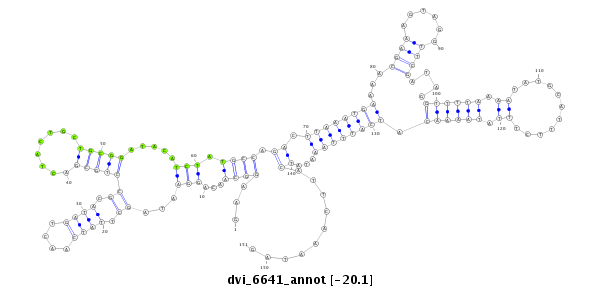
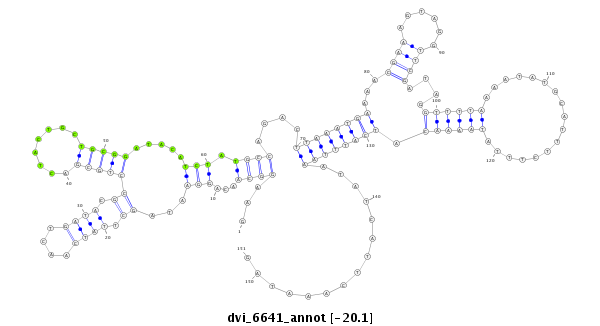
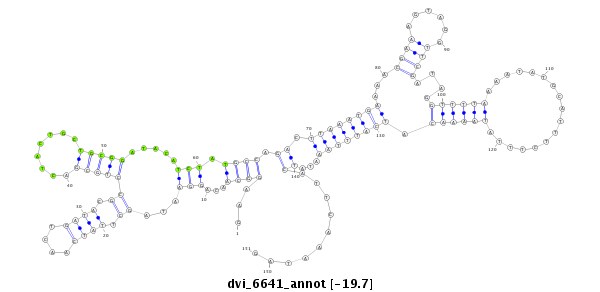
ID:dvi_6641 |
Coordinate:scaffold_12875:13274603-13274753 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.1 | -20.1 | -19.7 |
 |
 |
 |
CDS [Dvir\GJ21984-cds]; exon [dvir_GLEANR_7301:11]; intron [Dvir\GJ21984-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CAGACGAACAGGCAGAAGAGAACAGGAATAACTTAACAGCGAAACAGAAAGAAGGCAACAGGAATAGCTTATCAACTGATACGCCTGCGACTACTGCTGCGGATACATCTATGCCAGACTTAAATGAAAACGAAAGTAGGTTCGATAGGTTTTAAAATATGCATTTCTTTATAAAACATCATTTAAATATCATTCAAATAGCGCTGCCGCAATCAGTTTGTCCGTCCACACAACTTGAGTTGTCTCTGCGC **************************************************...((((...(((...((.((((....)))).))(((((........))))).....))).))))....((((((((...((((......))))....((((((.((...........)).)))))).))))))))...............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060666 160_males_carcasses_total |
SRR060663 160_0-2h_embryos_total |
V053 head |
M047 female body |
M061 embryo |
SRR060664 9_males_carcasses_total |
SRR060678 9x140_testes_total |
SRR060683 160_testes_total |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
SRR060687 9_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................................AATGAAAGCGAACGTAGGTT............................................................................................................. | 20 | 2 | 2 | 2.00 | 4 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 |
| ....................................CAGCGAAACAGAAAGAAGGC................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................AATGAAAGCGAACGTAGGTCCG........................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................CTACTGCTGCGGATACATCTAT........................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................TGCCGCAATCAGTTTGTCC............................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............AGAAGAGAACAGGAATAACTT......................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................GCTACTGATGCGGAGACATCT............................................................................................................................................. | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................CCGTCCACACAACTTGAGTT.......... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................GCCTGCGACTACTGCTGCGGA.................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................CTTAACAGCGAAACAGAAAGAAGGCA.................................................................................................................................................................................................. | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................ACCTCATTTAAAGATCATTC........................................................ | 20 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................TGAAAGCGAACGTAGGTT............................................................................................................. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................AATGAAAGTGAACGTAGGTTCG........................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................AATGAAAGCGAACGTAGGT.............................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................AATGAAAGTGAAAGTAGGTT............................................................................................................. | 20 | 2 | 6 | 0.33 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................ATGAAAGCGAACGTAGGT.............................................................................................................. | 18 | 2 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................ATACGCGTGTGACTACCGC.......................................................................................................................................................... | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................AATGAAAGGGAACGTAGGTT............................................................................................................. | 20 | 3 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................AATGAAAATGAACGTAGGTTG............................................................................................................ | 21 | 3 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................AAAGAAGGCAACAGGTAA.......................................................................................................................................................................................... | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........AGGCAGAGGAGAAAAGGA................................................................................................................................................................................................................................ | 18 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................TGCAACTACTGCTGGGGAGA.................................................................................................................................................. | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
|
GTCTGCTTGTCCGTCTTCTCTTGTCCTTATTGAATTGTCGCTTTGTCTTTCTTCCGTTGTCCTTATCGAATAGTTGACTATGCGGACGCTGATGACGACGCCTATGTAGATACGGTCTGAATTTACTTTTGCTTTCATCCAAGCTATCCAAAATTTTATACGTAAAGAAATATTTTGTAGTAAATTTATAGTAAGTTTATCGCGACGGCGTTAGTCAAACAGGCAGGTGTGTTGAACTCAACAGAGACGCG
**************************************************...((((...(((...((.((((....)))).))(((((........))))).....))).))))....((((((((...((((......))))....((((((.((...........)).)))))).))))))))...............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060667 160_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060675 140x9_ovaries_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..CTGCTTGACCTTCTTCTCTT..................................................................................................................................................................................................................................... | 20 | 2 | 1 | 2.00 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .TCTGCTTGACCTTCTTCTC....................................................................................................................................................................................................................................... | 19 | 2 | 1 | 2.00 | 2 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .TCTGCTTGACCTTCTTCTCTT..................................................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .TCTGCTTGACCTTCTTCTCTTT.................................................................................................................................................................................................................................... | 22 | 3 | 4 | 0.75 | 3 | 1 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| ..CTGCTTGACCTTCTTCTCT...................................................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| CTCTGCTTGACCTTCTTCTC....................................................................................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..CTGCTTGACCTTCTTCTCTTT.................................................................................................................................................................................................................................... | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................................................TTGTAGAAAAGTTTTAGTAAG........................................................ | 21 | 3 | 16 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................TGTAGAAAAGTTTTAGTAAG........................................................ | 20 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................CTTTGTCATTCTTTCGGTGT............................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:13274553-13274803 + | dvi_6641 | CAGACGAACAGGCAGAAGAGAACAGGAATAACTTAACAGCGAAACAGAAAGAAGGCAACAGGAATAGCTTATCAACTG-------ATACGCCTGCGACTACTGCTGCGGATACATCTATGCCAGACTTAAATGAAAACGAAAGTAGGTTCGATAGGTTTTAAAATATGCATTTCTTTATAAAACATCATTTAAATATCATTCAAATAGCGCTGCCGCAATCAGTTTGTCCGTCCACACAACTTGAGTTGTCTCTGCGC |
| droMoj3 | scaffold_6496:20563236-20563444 + | CAGAGGAGCGACAAGAAGAGATC------AACTCAACTCCGT-------------------------CGGATCAAACGGGTGCAACTGTACCTGAGACGCCTGAGGAGGCTATAGGTAAGTCGGAATTGAATGA-------------TGCGATTGCGACTAAGGCGTGCATTAAGCTAGTAAATATTT-----ATCCCATTTCAGAAGCATCGCAATCATCCACATGTCCACCCACACAACTTGATGTGTCTCCAAGT | |
| droGri2 | scaffold_15245:7080122-7080155 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGTCCGGCGACACAACTTGATGCATCGCTCTGC | |
| dp5 | 3:13544368-13544397 + | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCATTTGCCCGCACACTGAGCTCAACGC---------- | |
| droPer2 | scaffold_32:184123-184152 + | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCATTTGCCTGCCCACTGAGCTCAACGC---------- | |
| droRho1 | scf7180000780066:545795-545823 + | A-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACCAAGTTGTCCACCCACTCAGTTTAAC------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
Generated: 05/16/2015 at 08:16 PM