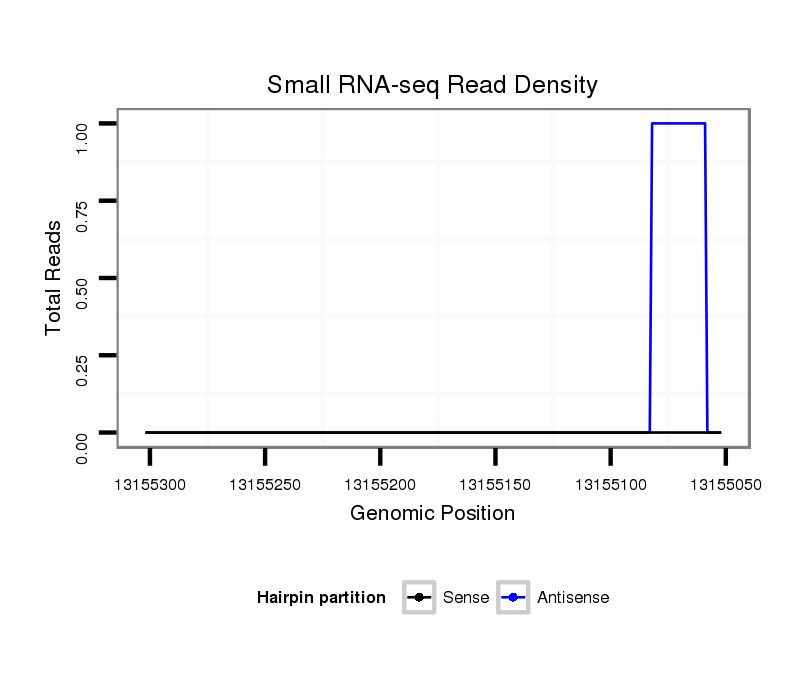
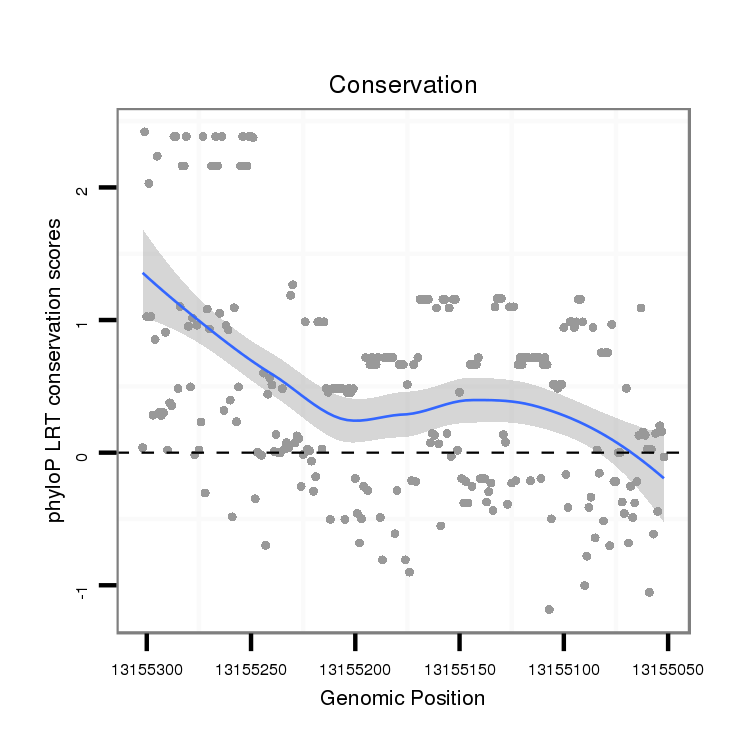
ID:dvi_6618 |
Coordinate:scaffold_12875:13155102-13155252 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_5801:1]; CDS [Dvir\GJ20359-cds]; intron [Dvir\GJ20359-in]
No Repeatable elements found
| -#################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AATGTGGACTAAAGAAAACGCAGCCAGACAGTGGCTGCTGGCCCTGACAGGTAAGAATTCCATTTCGAGTCCAGTATGATTATGATTAAGTATTAATGCTCAGCAAAAACAGCTTGTTTATGAGCCGTGACTTTTTATTAGCCAAATCGAAGGCAGACACGAGGCGCTTCAAAGACCACACAGATTATTTACGAGTTATCTAGCAATCAAATCGAACAATGCCCGACGACCACAAGAGGCAGACTCTTGTT **************************************************....(((......)))..((..(((((...(((......)))...)))))..))......(((((((..((((...(((..(((((....(((...(((..........)))))).....)))))...)))...))))..)))))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
M028 head |
V116 male body |
M061 embryo |
V053 head |
M027 male body |
V047 embryo |
SRR060689 160x9_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR1106729 mixed whole adult body |
SRR060655 9x160_testes_total |
SRR1106730 embryo_16-30h |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................GCAGACAGACAGTGGGTGCT.................................................................................................................................................................................................................... | 20 | 2 | 1 | 17.00 | 17 | 5 | 0 | 5 | 2 | 2 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................AGACAGACAGTGGGTGCT.................................................................................................................................................................................................................... | 18 | 2 | 7 | 7.57 | 53 | 18 | 10 | 0 | 5 | 2 | 2 | 0 | 3 | 4 | 2 | 2 | 2 | 2 | 1 | 0 | 0 |
| ...................GCAGACAGACAGTGGGTGC..................................................................................................................................................................................................................... | 19 | 2 | 2 | 6.00 | 12 | 6 | 4 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................GCAGACAGACAGTGGGTG...................................................................................................................................................................................................................... | 18 | 2 | 18 | 4.50 | 81 | 8 | 37 | 0 | 4 | 0 | 28 | 0 | 0 | 0 | 2 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..................GGCAGACAGACAGTGGGTGCT.................................................................................................................................................................................................................... | 21 | 3 | 4 | 3.00 | 12 | 6 | 0 | 1 | 2 | 1 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................CAGACAGACAGTGGGTGCT.................................................................................................................................................................................................................... | 19 | 2 | 3 | 2.33 | 7 | 1 | 1 | 2 | 0 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................GCAGACAGACAGTGGGTGCTA................................................................................................................................................................................................................... | 21 | 3 | 2 | 1.00 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................CAGACAGACAGTGGGTGC..................................................................................................................................................................................................................... | 18 | 2 | 20 | 0.55 | 11 | 2 | 6 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................GTTCGAGTCCAGTAAGAT........................................................................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................CAGACAGACAGTGGGTGCTA................................................................................................................................................................................................................... | 20 | 3 | 18 | 0.11 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................ACAGGTAAGTATTGCATT........................................................................................................................................................................................... | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................................................................................................................CAAACAATATCCGACGACC.................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................ATCCAGACAGTGGGTGCTA................................................................................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................AATGCTCAGCAAAAACGCCG......................................................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................CTGACAGACAGTGGGTGCT.................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................AGACAGACCGTGGGTGCT.................................................................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
TTACACCTGATTTCTTTTGCGTCGGTCTGTCACCGACGACCGGGACTGTCCATTCTTAAGGTAAAGCTCAGGTCATACTAATACTAATTCATAATTACGAGTCGTTTTTGTCGAACAAATACTCGGCACTGAAAAATAATCGGTTTAGCTTCCGTCTGTGCTCCGCGAAGTTTCTGGTGTGTCTAATAAATGCTCAATAGATCGTTAGTTTAGCTTGTTACGGGCTGCTGGTGTTCTCCGTCTGAGAACAA
**************************************************....(((......)))..((..(((((...(((......)))...)))))..))......(((((((..((((...(((..(((((....(((...(((..........)))))).....)))))...)))...))))..)))))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
M047 female body |
V116 male body |
SRR060684 140x9_0-2h_embryos_total |
SRR060657 140_testes_total |
SRR060665 9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................................................................................................CCGGCTGCTGGTGTTCTCCGTCTG....... | 24 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................CGGGCTGCTGGTGTTCTCCGTCTG....... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CGTCTGTCTGTCACCCACG..................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................CGGCTGTGCACCTCGAAGTT............................................................................... | 20 | 3 | 7 | 0.29 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................CGTCTGTGTACCTCGAAGTT............................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................CTGGCGTTCTCAGTCTCAG..... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................................................TTGTGCTCCGTGAAGTATC............................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................GATTAATAAGCGGTTTAGCT..................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:13155052-13155302 - | dvi_6618 | A-ATGTGGACTAAAGAAAACGCAGCCAGACAGTGGCTGCTGGCCCTGACAGGTAAGAATTCCATTTCGAGTCCAGT---------ATGATTATGATTAAGTATTAATGCTCA--------------------------------------------------------------------GCAAAAACAGCTTGTTTATGAGCCG--TGACTT---TTTATTAGCCAAA---TCGAAGGCAGACACGAGGCGCTTCAAAGACCAC---AC--AGATTATTTACGAGTT---------ATCTAGCAATCAA--AT--------CGAACAATGCCCGACGACCACAAGAGGCAGACTCTTGTT |
| droMoj3 | scaffold_6496:20439388-20439641 - | AGATGTGGACTAAAGAAATTGCAGCCAGACAATTGCTGCTGGCCTTGGCAGGTAAAGACAGCATA---AATGCAGTCGACAGCAAGTGATTACGATTAAGCATTAATACTCAATTAAGGCCAGCCAACAAGTA-----------------------------------------------G--CGAGCAGCTGCTTTATCGGCCCAT--ACTTGTTTTTATTAGCCAAAAAGTCGAAGGCAAACGCGAGGCACTTCAAAGACCAC---ACAAAGATTATTTACGAGGGCAG------------------A--A---------------------------T---GGAAGCAGAATCTTGTT | |
| droGri2 | scaffold_15245:7200906-7201195 + | G-ATGTGGACTAAAGCAAATGCAGCCGGGAAAGGGCTGCTCACCTTGGCAGGTAAGAAATCGCTGT------------------------------------------------TTAGGCCAACAAAAAACTGATTTGATGATTTGTGTGTGATTTGTGTATTAATGTAACAAAAGCAAAATCAAAGCAGCTGCTTTATGGGCCCCATCGCAT---TTTATTAGCTCAA---TCGAA--TCGCCACGAAATAAAACAAAGAACTCAGCGC--AGATTGTTTATGAGCT--GTTTTCCATCTAGTTATCAAAAATTGTACAACGCTGCCAC----------TGCCACCGGCAGTTCATAATT | |
| dp5 | 3:4512419-4512461 + | A-A----------GGGAAATGCAATCGGCAACTGGCTGTTGGCTATCACAGGTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_2:4701317-4701370 + | A-A----------GGGAAATGCAATCGGCAACTGGCTGTTGGCTATCACAGGTAAACCCTTCACT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13266:1043063-1043138 + | A-ATGTGGACCCCCCGAATTGCAGTCAACCACTGGCTGTTAGTCACCACAGGTAATCCCTTCACTT---ACCCAGT---------AAG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A | |
| droBip1 | scf7180000396730:2040682-2040735 + | A-ATGTGGATCCCCCGAATTGCAGTCAACCACTGGCTGTTGGTCACCACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droFic1 | scf7180000453773:265255-265308 + | G-ATGTGGACGCAGAGAAATGCAGTCGGAAATTGGCTGTTGGTCTTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEle1 | scf7180000491214:2781414-2781467 + | G-ATGTGGACGCAGAGAAATGCAGTCGGCAATTGGCTGTTGGTCTTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000779263:223280-223333 - | G-ATGTGGACGCAGAGAAATGCAGTCGGCAATTGGCTGTTGGTCTTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBia1 | scf7180000302292:91757-91810 + | G-ATGTGGACGCAGAGAAATGCAGTCGGCAACTGGCTGTTGGTCATAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droTak1 | scf7180000415991:609340-609393 + | G-ATGTGGACGCAGAGAAATGCAGTCGGCAATTGGCTGTTGGTCCTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEug1 | scf7180000409462:1174833-1174939 - | G-ATGTCGACGCAGAGAAATGCAGTCGGCAATTGGCTGTTGGTCTTAACAGGTAATCCCTTCACTC---ATCCATC---------ACCACACCCATCA-----------------------------------------------------------------------------------------------------------------------------------------------------------AGCAAAACACTCAGC------------------------------------------------------------------TATA--------------------GCA | |
| dm3 | chr2R:7332216-7332269 + | G-ATGTGGACGCAGAGAAATGCAGTTGGCAATTGGCTGTTGGTATTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droSim2 | 2r:8038471-8038524 + | G-ATGTGGACGCAGAGAAATGCAGTTGGCAATTGGCTGTTGGTATTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droSec2 | scaffold_1:4884870-4884923 + | G-ATGTGGACGCAGAGAAATGCAGTTGGCAATTGGCTGTTGGTATTAACAGGTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droYak3 | 2R:9630405-9630468 - | G-ATGTGGACGCAGAGAAATGCAGTTGGCAATTGGCTGTTGGTATTAACAGGTAATCTCTCCACT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4793:35270-35346 - | A-AAGATCAAGTAA----------ATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTATGTACCGAAGCTACCAAA----------------------------------------------------------------GCAATCAA--AT--------TCAACAGCGGCCAACG----------ACGAAATGAGGTT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:04 PM