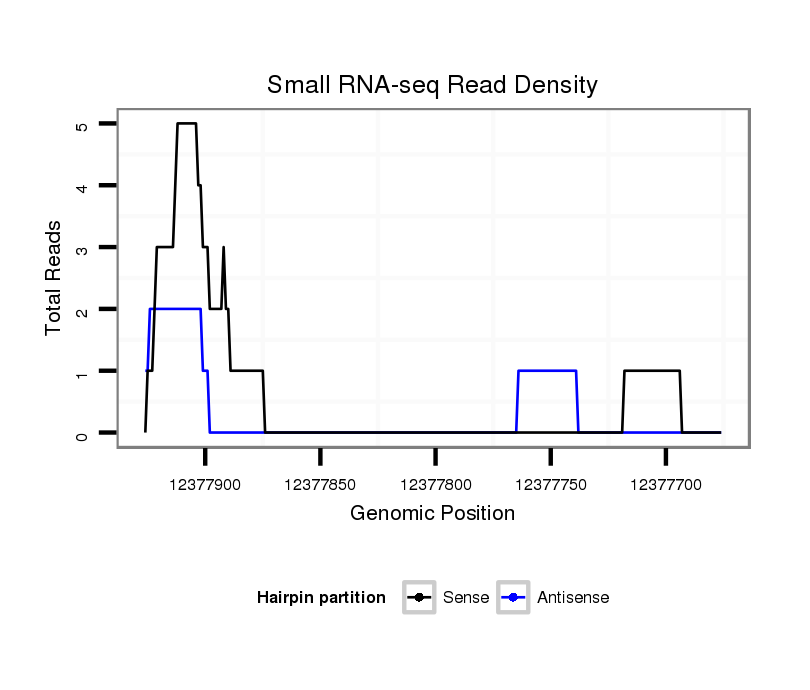
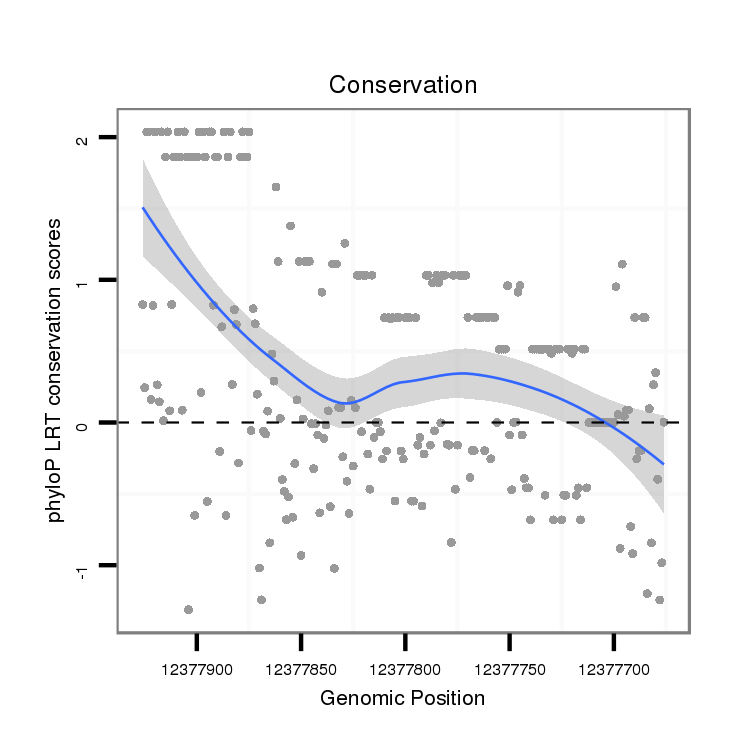
ID:dvi_6458 |
Coordinate:scaffold_12875:12377726-12377876 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_5846:9]; CDS [Dvir\GJ20406-cds]; intron [Dvir\GJ20406-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AAATCTTTCTGGATAGCACGACCCCAGAGACCAAAGCACTTGTATCTCAGGTAAGTTCATTCAAATTTGAATGTGATAAAAATTGTCCGATTGAAAATTTGAAATAAATTAGAAAATAAGTCTATATTTGGTTCCTTAAGCTGATATAAATATTTTTGTTTAATGTTTAAAAAAATCACTCTCTTTCAAATTATATCTATTGATAAGTATCAACGGCGGCATTGTCGGTCCGTTATTATATTAGGAAATCA **************************************************.........(((((((((.(((..(((((...)))))..)))..)))))))))....(((((....((((.(((....)))...))))..))))).((((((((.......))))))))................................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
M061 embryo |
SRR060665 9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
V116 male body |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
M047 female body |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............AGCACGACCCCAGAGACCAAAGC...................................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....CTTTCTGGATAGCACGACCCCAGA............................................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............TAGCACGACCCCAGAGACCAAA........................................................................................................................................................................................................................ | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .AATCTTTCTGGATAGCACGACCCC.................................................................................................................................................................................................................................. | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................TTGATAAGTGTCAACGGCGGC............................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....TTTCTGGATAGCACGACC.................................................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................AGCACTTGTATCTCAGGT....................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................ATCAACGGCGGCATTGTCGGTCCGT.................. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................CAAAGCACTTATATCGCAGGA....................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................TGTAGCTCAGGTTAGTTC................................................................................................................................................................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................................................................................................ATAAGTATCAGCGGCAGC............................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................................................................................GTATCAACGGCGACCTTC........................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTTAGAAAGACCTATCGTGCTGGGGTCTCTGGTTTCGTGAACATAGAGTCCATTCAAGTAAGTTTAAACTTACACTATTTTTAACAGGCTAACTTTTAAACTTTATTTAATCTTTTATTCAGATATAAACCAAGGAATTCGACTATATTTATAAAAACAAATTACAAATTTTTTTAGTGAGAGAAAGTTTAATATAGATAACTATTCATAGTTGCCGCCGTAACAGCCAGGCAATAATATAATCCTTTAGT
**************************************************.........(((((((((.(((..(((((...)))))..)))..)))))))))....(((((....((((.(((....)))...))))..))))).((((((((.......))))))))................................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060681 Argx9_testes_total |
GSM1528803 follicle cells |
SRR060675 140x9_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060670 9_testes_total |
SRR060680 9xArg_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060689 160x9_testes_total |
V116 male body |
M047 female body |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060655 9x160_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................................AAGGAACTAGACTATATTTA.................................................................................................... | 20 | 2 | 3 | 3.33 | 10 | 5 | 0 | 0 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................TACAAATTTTTTTAGTGAGAGAAAGT............................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..TAGAAAGACCTATCGTGCTGGGGTCT............................................................................................................................................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| TTTAGAAAGACCTATCGTGCTGGGG.................................................................................................................................................................................................................................. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................TAGGGTCCATTCAACTAACTT........................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................CAAGGAACTAGACTATATTTA.................................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................AAGGAACTAGACTATATTT..................................................................................................... | 19 | 2 | 4 | 0.50 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................ACAAAGGAACTAGACTATATTT..................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................AAGGAACTAGACTATATT...................................................................................................... | 18 | 2 | 10 | 0.40 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 1 | 1 | 0 |
| ........................................................................................................................................................................................................................GCCTTAACAGCCAAGCAA................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................AAAGGAACTAGACTATATTTA.................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................CCGTACCAGCTAGGCCATA............... | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................GGGTCCATTCAACTAACTTT.......................................................................................................................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................AAAGGAACTAGACTATATTT..................................................................................................... | 20 | 3 | 20 | 0.10 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..TAGAGATCCCTATCGTGCT...................................................................................................................................................................................................................................... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................TAGGGTCCATTCAACTAAC............................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:12377676-12377926 - | dvi_6458 | AAATCTTTCTGGATAGCACGACCCCAGAGACCAAAGCACTTGTATCTCAGGTAAGTTCAT------------TCAAATTTGAATGTGATAAAAATTGTC--------------------------------------------------------------------------------------CGATTGA-----------------------------AAATTT---GAAATAAATTAGAAAATAAGTCTATATTTGGTTCCTTAAGCTGATATAAA---TATTTTTGTTTAATGTTTAAAAAAATCACTCTCT----------------TTCAAATTATATCTATTGATAAGTATCAACGGCGGCATTGTCGGTCCGTTATTATATTAGGA----AATCA |
| droMoj3 | scaffold_6496:14485138-14485224 + | AAATCTTTCTGGATAGCACGACACCGGAGACCAAAGCACTCGTATCTCAGGTGAGAATTATAATTGCTT---TTAAATT---ATGTGACAAAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15245:7977387-7977599 + | AAATCTTTTTGGAAAGCACAACACCGGAAACCAAAGCACTTGTATCTCAGGTAAGGTGACTATT--ATTCATGCAAATAATTCTCAGAAGAAAACTGGCC--------------------ATTTATAT---------------------------------------TGATATGAAAAGT------------------GCTTGAGCCGCCGG--------------------------------------------------------------------------------------------AAAATCCCTCTCAAGACTTTAAGACTTGGCCAAAATTAAATCGATTTTAAAGTTCAAAT--------------CACTTTTG------TGTTTGTCATGTA | |
| droWil2 | scf2_1100000004382:1122286-1122342 - | AGATTTTCCTTGAAAGCACAACACCTGAGACTAAAGCTCTTGTTTCACAGGTGAGTT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 3:134458-134612 - | AAATTTTCCTTGATAGCACTACTCCTGAGACCAAAGCTCTCGTATCACAGGTGAGTGGATCAGTGGATCCATTAAAA-----------------------------------AGATTATAATTTTGTTTGATTA---------------TAAATCAAAATTGTATC------------------------------ATGCTGAC------TAAA---GTTAAAGTACATCGTG----------------------------------------------------------------------------------------------------------------------------------------------------------------AAATTTTTATT | |
| droPer2 | scaffold_2:324876-325030 - | AAATTTTCCTTGATAGCACTACTCCTGAGACCAAAGCTCTCGTATCACAGGTGAGTGGATCAGTGGATCCATTAAAA-----------------------------------AGATTATAATTTTGTTTGATTA---------------TAAATCAAAATTGTATC------------------------------ATGCTGAC------TAAA---GTTAAAGTACATCGTG----------------------------------------------------------------------------------------------------------------------------------------------------------------AAATTTTTATT | |
| droAna3 | scaffold_13266:19386555-19386773 + | AAATAATTTTAGAAAGCACAACTCCGGAGACAAAAGCATTAGTATCACAGGTATTTAT--------------AAAAA------TTAGAAAAAATTATTCTAATTAATTAATTACGTTATAAATTTCTTAGATATTTTAATATTTTGGTGTTAAAAAAATTTGTATTTTGCTTTGTA-----AAAAAATGTTAAATTT---------------------------------------------------------------------------------------------------------------TACTCCTAAAGACGCGTCGATTG---------------------------------------------GTCCGAAATTTCGTTCAAA----AGTTA | |
| droBip1 | scf7180000396710:7417-7587 - | AAATAATTTTAGAAAGCACAACCCCGGAGACTAAAGCATTAGTATATCAGGTAATTGT--------------TAACA-----------------------------------------------------------------------------------------------------CTAAAAAAATGTCAAATTTTGTTTAAGCCTTTTGAAGGGGTCAAAATAC---GAAATCAAATAT--C-----------------TACATAAGTTGTTACACAATCTATTTTTTCCTAATATTAAA------------------------------------------------------------------------------------------------AGAAA | |
| droKik1 | scf7180000301494:24319-24423 - | AAATTTTTTTAGAAAGCACAACTCCAGAAACTAAGGCTTTAGTATCACAGGTAAGTAAATTT-----TT---TTT--------------------------------------------------------------------------------------------TGTTATGTAAAGT------------------TCG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTACGTTGA------GGTAAATCAGTAA | |
| droFic1 | scf7180000453334:16311-16497 + | GCATTTTTTTAGAAGGCACAACACCGGAGACAAAAGCCTTGGTTGCCCAGGTAAGTGTTTCATTAACTT---TC--ATTTGCTTGTT------------------------------------------------------------------------------------CTGAAAACCGCGTACGTCTCAGATCTTGTTAAC------TAAA---ATTACCGTGGAACATAATCAATGAT--CTTGAGTGTACTTTTCCTCAGTTAGGCTGTTATGGA---GGTTTT------------------------------------------------------------------------------------------------------------------T |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
Generated: 05/17/2015 at 04:55 AM