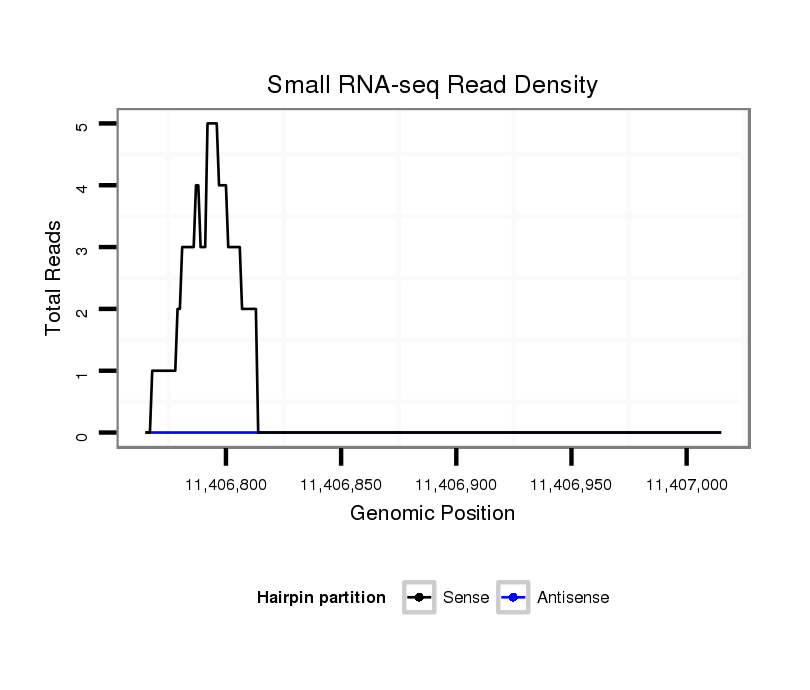
ID:dvi_6124 |
Coordinate:scaffold_12875:11406815-11406965 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_7160:1]; CDS [Dvir\GJ21833-cds]; intron [Dvir\GJ21833-in]
No Repeatable elements found
| ---------------------------#######################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GGCCTTGCCACAGTGAAGCTCGGCAAGATGTTCATCGAAGATGATGCCCAGTAAGTACTCTCCCATTGAACTATCTAATTGTCCAACCTTTATGTAAGTCATGTCAACACTTGCTTTAATTTGTGTACACTTTTACGTAGCGAAAGATTTGAGAATGCATGAGATACGCGCTATCAGGACAAGTCAAATGTAATTTTAATTTAAGCAGAAAATTCTCAACAATTCGATTCTTTTTTAAATTTAATAACATA **************************************************.............((((..(((.......(((((........((((..((((((.........((((....((((........)))).....))))..........))))))..))))........))))))))..))))...........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
V053 head |
SRR060663 160_0-2h_embryos_total |
SRR060680 9xArg_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
V116 male body |
M047 female body |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................ATGTTCATCGAAGATGATGCCC.......................................................................................................................................................................................................... | 22 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................ATGTTCATCGAAGATGCTGCCC.......................................................................................................................................................................................................... | 22 | 1 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...CTTGCCACAGTGAAGCTCGGC................................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................AGATGTTCATCGAAGATGATGCCCAAAT...................................................................................................................................................................................................... | 28 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............GAAGCTCGGCAAGATGTT........................................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........ACAGTGAAGCTCGGCAAGATGCT........................................................................................................................................................................................................................... | 23 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................AGCTCGGCAAGATGTTCATC....................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................GCAAGATGTTCATCGAAGAT................................................................................................................................................................................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................ATGTTCATCGAAGGTGATGCCC.......................................................................................................................................................................................................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................CGGTTCATCGAGGATGATGCC........................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................CTACAAGAAGTTCATCGAAGAT................................................................................................................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................CAAGAAGTTCATCGAAGAT................................................................................................................................................................................................................. | 19 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................GTGTTCATAGAAGATGATCC............................................................................................................................................................................................................ | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................AAGTTCAGCGATGATGATGCC........................................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................GCGCAAGATATGAGACTGCA............................................................................................ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................................................................................GTATGGATGAGATAAGCGCT............................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................GGGTCATCGAAGATGACGC............................................................................................................................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................AAGTTGTTCTTCGAAGATGT............................................................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................ATGGTCATCGATGATGAGGC............................................................................................................................................................................................................ | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................AGTTGTTCTTCGAAGATGT............................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CCGGAACGGTGTCACTTCGAGCCGTTCTACAAGTAGCTTCTACTACGGGTCATTCATGAGAGGGTAACTTGATAGATTAACAGGTTGGAAATACATTCAGTACAGTTGTGAACGAAATTAAACACATGTGAAAATGCATCGCTTTCTAAACTCTTACGTACTCTATGCGCGATAGTCCTGTTCAGTTTACATTAAAATTAAATTCGTCTTTTAAGAGTTGTTAAGCTAAGAAAAAATTTAAATTATTGTAT
**************************************************.............((((..(((.......(((((........((((..((((((.........((((....((((........)))).....))))..........))))))..))))........))))))))..))))...........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
SRR060663 160_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
V053 head |
SRR060667 160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................CATGAGAGGGTTAATTGTTAGATT............................................................................................................................................................................. | 24 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TTGAGCCGTTGAACAAGTAGC...................................................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................AGATGGTAACTTGATATA............................................................................................................................................................................... | 18 | 2 | 19 | 0.16 | 3 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................CGAGCAGATCTCCAAGTAG....................................................................................................................................................................................................................... | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................AGTGTAGTTGTGAACGAAAGT.................................................................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................ATGGTAACTTGACAGATTGA........................................................................................................................................................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................................................AAAAAATTTAAATTCTTGAAT | 21 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................GTCATTCATGCCAGGGTG......................................................................................................................................................................................... | 18 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................GCCAAGTAGCTTCAACTAC............................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .......................................................................................................................................................................................TGGTTACATTAAAATTAAACT............................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:11406765-11407015 + | dvi_6124 | GGCCTTGC--CACAGTGA-AGCTCGGCAAGATGTTCATCGAAGATGATGCCCAGTAAGTACTCT--CCCA-TTGA-ACT--ATCTAATTGTCCAACCTTTATGTAAGTCATGTCAACACTTGCTTTAATTTGTGTACACTTTTACGTAGCGAAAGATTTGAGAATGCATGAGATACGCGCTATCAGGACAAGTCAAA-TG---TAAT-------------------------------------TTTAATTTAAGCAGAAAATTCTCAACAATT-CGATTCTTTT-TTAAATTTAATAACATA |
| droMoj3 | scaffold_6496:15519389-15519476 - | TGGCTTGC--CACAGTAA-AGCTTGGCAACATGTTCATCGAAGATGATGCGCAGTAAGTACCGT--TTCA-TTCAAACTTTATCTA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTATCTA | |
| droGri2 | scaffold_15245:13317365-13317459 - | GCTTGCCACCCACAGTAAAAAGTCGGCAAGATGTTCATCGAAGATGATGCGCAGTAAGTACAATTTC-CATTTAA-ACT--ATCC-----CCCATTTTTTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGT | |
| droWil2 | scf2_1100000004961:225516-225584 - | GAACAAAT-----------------------A------------------------------------------------------------------------------------------------------------------------------------------------------------------AAT-TG-------AATTTATAATTTCTGTGAAAGAATTGATTATTCTGAT-----------------------------------------TTCAAACTTTGTAATATC | |
| dp5 | Unknown_group_200:16269-16307 - | TCTACA-----------------------TTT-----------------------------------------TG-GTT--TTTT-----TTTTATTTTCG----------------------------------------------------------------------------------------------------------------------------------------------------TG--------------------------------------------TCAATAAG | |
| droPer2 | scaffold_166:89329-89382 - | TATAAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGAATTTGGGAAATAAAATTCGAGCAAAT-GGGCACTTTT----------TTAAATTT | |
| droKik1 | scf7180000301900:9992-10046 + | ATGTTAAA--TGAAGAAAAACGGTGGCAAA---------------------------------------AATTAA-GTT--CGCC-----ACTGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTCGTTA | |
| droEle1 | scf7180000491271:9753-9783 - | AATATG-----------------------AATATGTATTAAAC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATGAGTATGTA | |
| droRho1 | scf7180000774982:28787-28868 - | AAGGCAAT-----------------------T-----------------------------------------------------------------------------------------------------------------------------------------------------------------CGAAATGTTTTTAT-------------------------------------TTACAATTAAATAAGAAGTCTTAAACAACTACAATGCTGTTAGTAAATCCAAAAACAAA | |
| droBia1 | scf7180000301585:66933-66982 + | TTAATTTA--A---AAAAAACATTAAATTTATTCTATTTGAAGATGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTTGCTTA | |
| droTak1 | scf7180000414726:9280-9350 + | AAATACAATCATGGGTTA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTTCTAATAATTTTGGGCAGCATAACGATTGGAAA------------------------------------------TTAAA-ATAAATAAATG | |
| droEug1 | scf7180000409183:219451-219487 + | CGGCAA-----------------------GATGTTCATCGAAGATGATGCCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTAAGTA | |
| droYak3 | 2R:1313890-1313928 - | CAAAAA-----------------------TTTGCCG---------------------------------ATTTCA-AAT--ATTC-----ACTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGATATA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/19/2015 at 01:49 PM