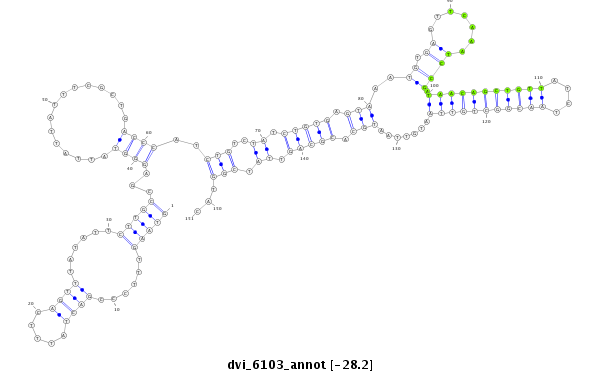

ID:dvi_6103 |
Coordinate:scaffold_12875:11310195-11310345 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -28.2 | -28.2 | -28.1 |
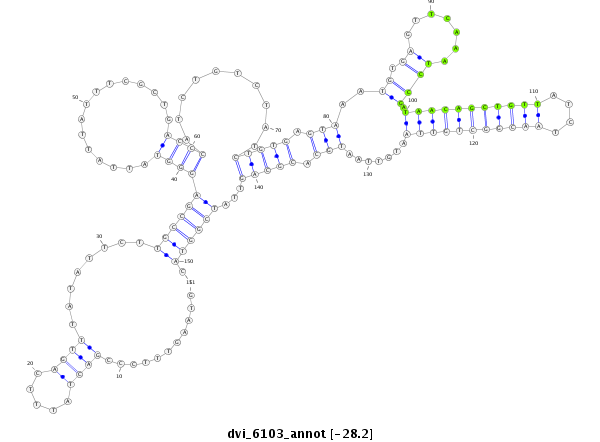 |
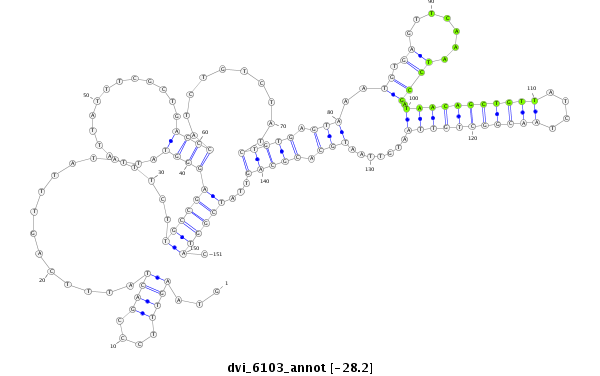 |
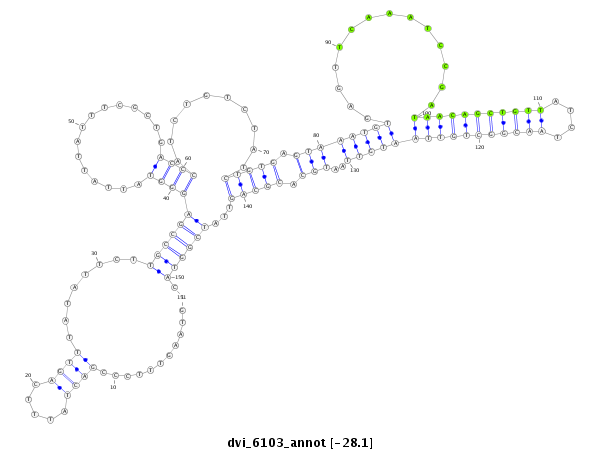 |
CDS [Dvir\HP2-cds]; exon [dvir_GLEANR_7152:10]; intron [Dvir\HP2-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AACAACAGCGTTCACGGATCCAAATGAAGCCAGCTGGAGGCAACAGCAAGGTAAGTTTCCCGACTATTTCAGTTTATATTCTTGCCGAGGGTATTATTATTTCGCTGACCCATCTGTCTATCTGTGAGTAAATGTGAGTTCAAATCCGATAACAGCTGTTATCTAACGGCTGTTAATGTTAATGCACGCAGTTATCGGTACATACTCGATTATTCGGTACTGTTATGTCGCTTTCAGCGAACTTTTCGAGT **************************************************(((((......((((.....))))......)))))...((((...............))))..(((..((.(((((.(((..((.((.......)))).(((((((((((....))))))))))).......))).))))).)).)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060663 160_0-2h_embryos_total |
V047 embryo |
V116 male body |
V053 head |
SRR060678 9x140_testes_total |
SRR060683 160_testes_total |
SRR1106722 embryo_10-12h |
M028 head |
SRR060666 160_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR1106727 larvae |
SRR1106728 larvae |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................ATCCCAATGATGCCAGCTGGTG.................................................................................................................................................................................................................... | 22 | 3 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................ATCCCAATGATGCCAGCTG....................................................................................................................................................................................................................... | 19 | 2 | 2 | 1.50 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............ACGGATCCAAATGAAGCCCGCT........................................................................................................................................................................................................................ | 22 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TCCCGACTATCTCAGTCTGTA............................................................................................................................................................................. | 21 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................TCAAATCCGATAACAGCTGTT........................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...AACAGCGTTCACGGATCCAAAT.................................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................TATGTCGCTTTCAGCGAACTTTTCGAG. | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................CAAATGAAGCCAGCTGGAG.................................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| AACAACAGCGTTCACGGATC....................................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........TTCACGGATCCAAATGAAGCCAGCTGGAGGCAAC............................................................................................................................................................................................................... | 34 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...AACCGCGTTGACTGATCCAAAT.................................................................................................................................................................................................................................. | 22 | 3 | 2 | 1.00 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................TGAAGACAGCTGGAGGGATCA.............................................................................................................................................................................................................. | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................TGAACCCAGCTGGAGGGATC............................................................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....ACCGCGTTGACTGATCCAAA................................................................................................................................................................................................................................... | 20 | 3 | 6 | 0.33 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...AACCGCGTTGACTGATCCAAA................................................................................................................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................ATCCCAATGATGCCAGCT........................................................................................................................................................................................................................ | 18 | 2 | 8 | 0.25 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................TGAAGACAGCTGGAGGGATC............................................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................GTGAAGCCTGCTGGAGGTA................................................................................................................................................................................................................. | 19 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................GGAAGACAGCTGGAGGCATC............................................................................................................................................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................CTATCTGAGAGTAACTGTT................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| .....................................................................................................................ATATCTGTGAGTAACTGT.................................................................................................................... | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................CGCTGACGGTTCTGTCTAT.................................................................................................................................. | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................TATCTGAGAGTAACTGT.................................................................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTGTTGTCGCAAGTGCCTAGGTTTACTTCGGTCGACCTCCGTTGTCGTTCCATTCAAAGGGCTGATAAAGTCAAATATAAGAACGGCTCCCATAATAATAAAGCGACTGGGTAGACAGATAGACACTCATTTACACTCAAGTTTAGGCTATTGTCGACAATAGATTGCCGACAATTACAATTACGTGCGTCAATAGCCATGTATGAGCTAATAAGCCATGACAATACAGCGAAAGTCGCTTGAAAAGCTCA
**************************************************(((((......((((.....))))......)))))...((((...............))))..(((..((.(((((.(((..((.((.......)))).(((((((((((....))))))))))).......))).))))).)).)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060660 Argentina_ovaries_total |
SRR060658 140_ovaries_total |
SRR060659 Argentina_testes_total |
SRR060687 9_0-2h_embryos_total |
V047 embryo |
SRR060656 9x160_ovaries_total |
SRR060673 9_ovaries_total |
SRR060688 160_ovaries_total |
SRR1106716 embryo_4-6h |
SRR060668 160x9_males_carcasses_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................................................................................................................CCATGAGAATACAGAGGAA................. | 19 | 3 | 15 | 0.53 | 8 | 1 | 1 | 2 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 0 | 0 |
| ....................................................................................................................................................................................................CCATGTACTAGCTACTAAGC................................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................TGACAATGCAGTGAAAGT............... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................TGACAATGCAGTGAAAGTTG............. | 20 | 3 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................AATGGCTGTTAAAGTCAA................................................................................................................................................................................. | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........AGTGCCTAGGTGTACTAAG............................................................................................................................................................................................................................. | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................................CGTCGCTTGAAAAGCTTA | 18 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................CTGCTCCCATACTAATAG...................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:11310145-11310395 + | dvi_6103 | AACAACAGC---------G-----TTCACGGATCCAAATGAAGCCAGCTGGAGGCAAC---AGCAAGGTAAGTTTCCCGACTATTTCAGTTTATATTCTTGCCGAGGGTATTATTATTTCGCTGACCCATCTGTCTATCTGTGAGTAAATGTGAGTTCAAATCCGATAACAGCTGTTATCTAACGGCTGTTAATGTTAATGCACGCAGTTATCGGTACATACTCGATTATTCGGTACTGTTATGTCGCTTTCAGCGAACTTTTCGAGT--------------------------- |
| droMoj3 | scaffold_6496:15614510-15614593 - | A-CAGCAAC---------G-----TGGACG---------TCAAAGTGC---AGCCAGC---GGCAAGGTGAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGAACTTCTCTAAATAATCCTTGTATAGAATACTAATACTT | |
| droGri2 | scaffold_15245:13419543-13419604 - | CACAGCAGCAGCTGCAACG-----CACACGCTTCCTTATGGTGCCC---AATGCCAGC---AGCAAGGTAAAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droFic1 | scf7180000453778:752388-752421 + | AATAAGAGT---------G-----CT-----------------------GGAGCTAACCCAAACCAGGTAG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415272:364555-364609 - | GCGTTCTAC---------GCAGGGAGGAAG---------GAAGAGTGCCGGAGGAAACGCAAGCCAGGTAAAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dm3 | chr2R:10495440-10495451 - | AAGATCCAC---------G-----TT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 05/19/2015 at 01:47 PM