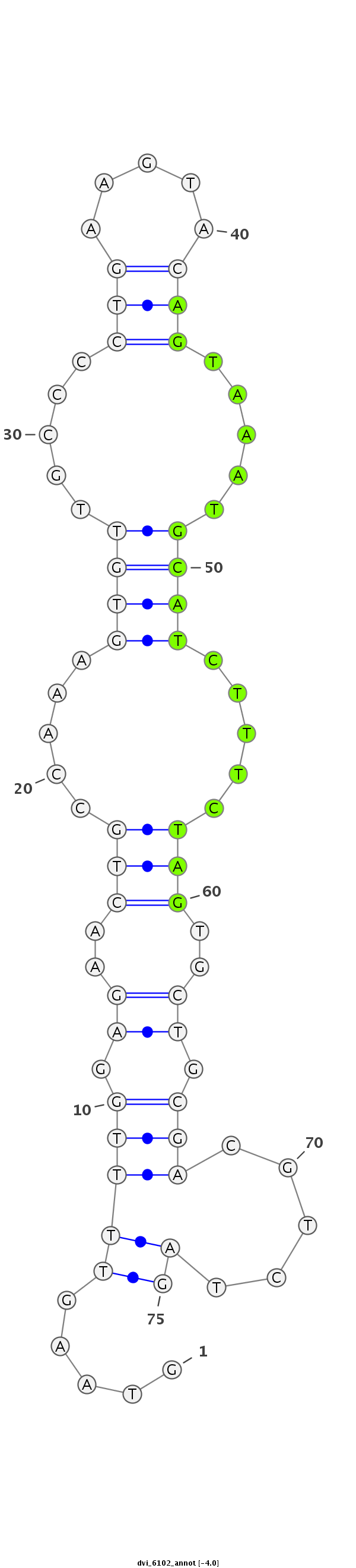
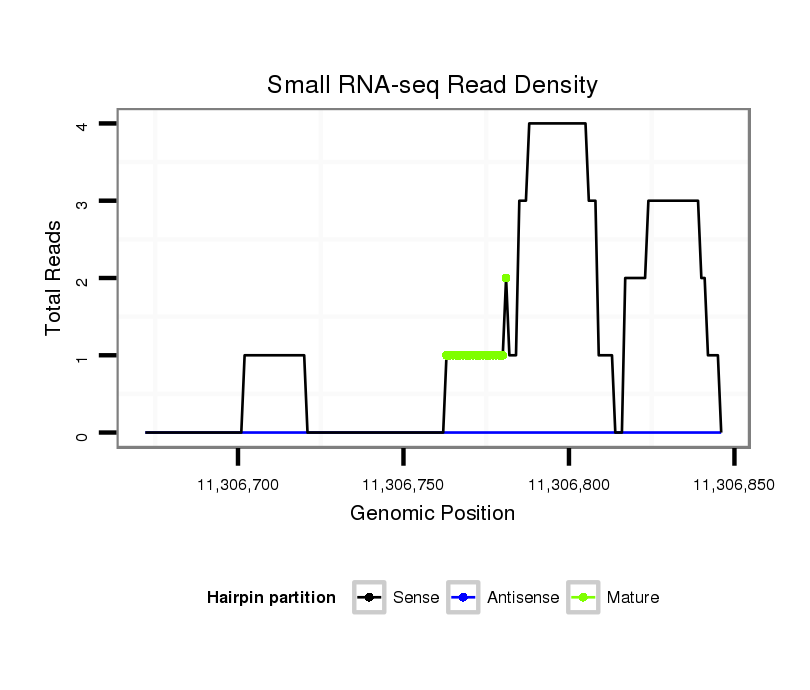
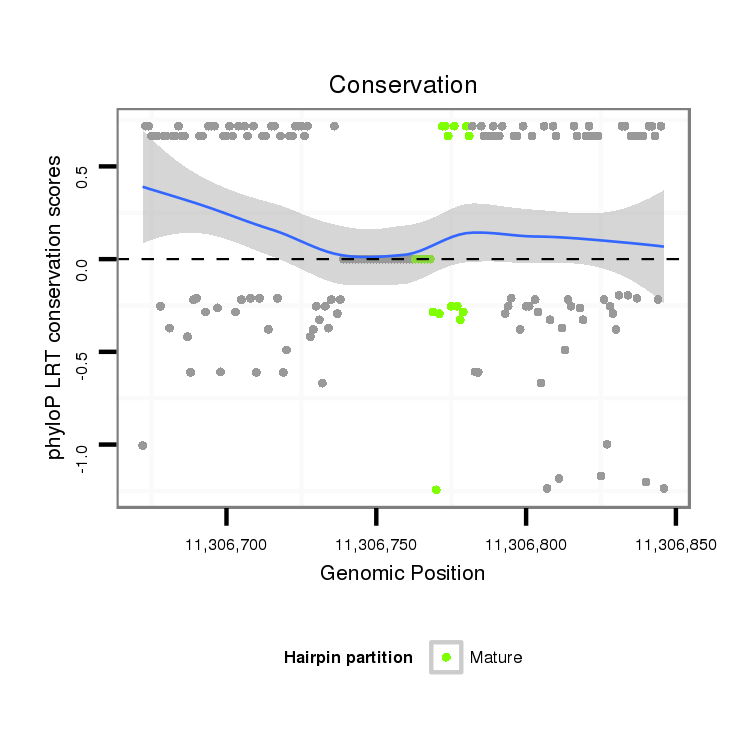
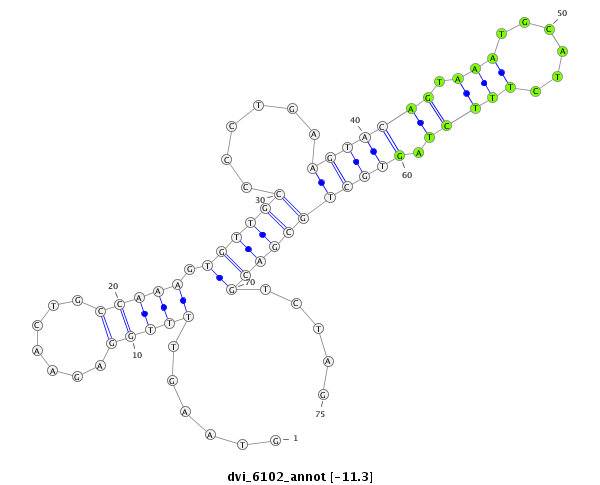
ID:dvi_6102 |
Coordinate:scaffold_12875:11306722-11306796 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -12.0 | -11.5 | -11.4 | -11.3 |
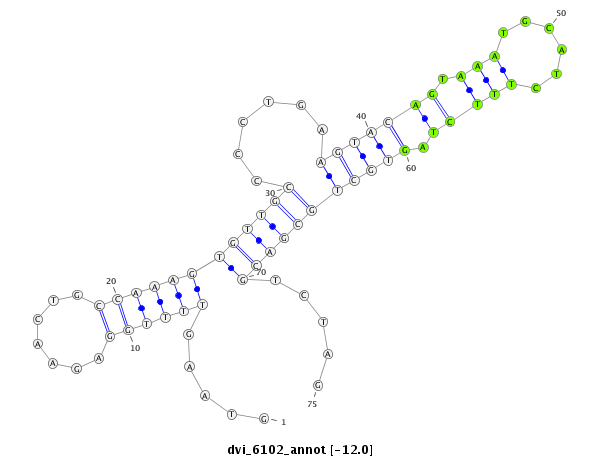 |
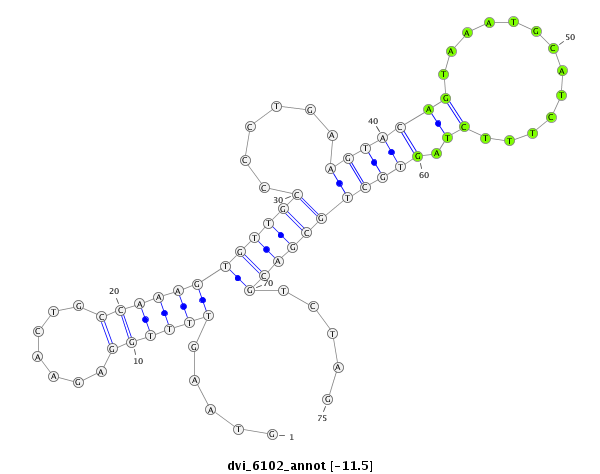 |
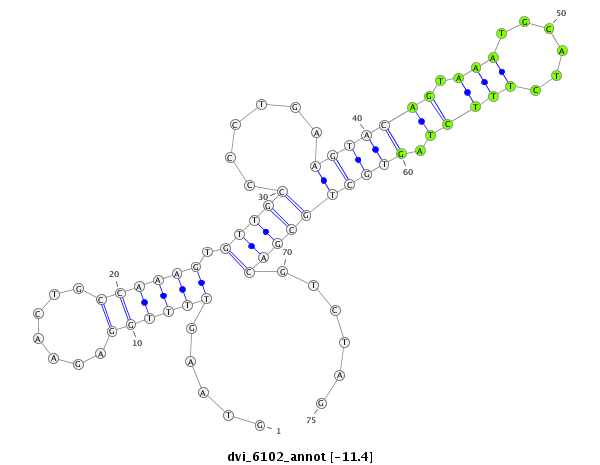 |
 |
exon [dvir_GLEANR_7152:9]; CDS [Dvir\HP2-cds]; CDS [Dvir\HP2-cds]; exon [dvir_GLEANR_7152:10]; intron [Dvir\HP2-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------------------------################################################## TATGGCACCGCCAGCTCCACCTAAAAGGGAGTTGTCTACTGGTAAAGCTGGTAAGTTTTGGAGAACTGCCAAAGTGTTGCCCCTGAAGTACAGTAAATGCATCTTTCTAGTGCTGCGACGTCTAGGAAAACCTCTGCAGACTGATGTGCAGCCTACACAGAAGCGTCGGAAGCTC **************************************************.....(((((.((..(((.....((((.....(((.....))).....)))).....)))..)).))).....))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060661 160x9_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060659 Argentina_testes_total |
SRR060674 9x140_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
V116 male body |
M061 embryo |
SRR060671 9x160_males_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060666 160_males_carcasses_total |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................AACTGGCAAAATGTTGCCCCT........................................................................................... | 21 | 2 | 1 | 3.00 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GACGTCTAGGAAAACCTCTGC...................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................GTGCTGCGACGTCTAGGAAAACCTC......................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................GTGCAGCCTACACAGAAGCGTCGGA..... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................GTTGTCTACTGGTAAAGCT.............................................................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TGCGACGTCTAGGAAAACCTCTGC...................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................AGTAAATGCATCTTTCTAG................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................GTGCAGCCTACACAGAAGCGTCG....... | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TGCGACGTCTAGGAAAACCTCTGCAGACT................................. | 29 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................CTACACAGAAGCGTCGGAAGCT. | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................TATTGCTAAAGCTGGTAA......................................................................................................................... | 18 | 2 | 10 | 0.20 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................TGCCAAAGTGTTGCCCGTTAC........................................................................................ | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................CCGTGAAGTACAGAAAAT............................................................................. | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................AGCTGGCAAGTTTTCGAG................................................................................................................ | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................ACAGTAAAGGCATTTTTC.................................................................... | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................GCCAAAGTGTTGCCCGTT.......................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ATACCGTGGCGGTCGAGGTGGATTTTCCCTCAACAGATGACCATTTCGACCATTCAAAACCTCTTGACGGTTTCACAACGGGGACTTCATGTCATTTACGTAGAAAGATCACGACGCTGCAGATCCTTTTGGAGACGTCTGACTACACGTCGGATGTGTCTTCGCAGCCTTCGAG
**************************************************.....(((((.((..(((.....((((.....(((.....))).....)))).....)))..)).))).....))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060661 160x9_0-2h_embryos_total |
M027 male body |
|---|---|---|---|---|---|---|---|---|
| ........................................................................................ATGTCATTTACGTAGAGAGTA.................................................................. | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 |
| .........CGGTCGACGTGGATTTTC.................................................................................................................................................... | 18 | 1 | 3 | 0.33 | 1 | 0 | 1 | 0 |
| ...............................................................................................................................................CACAAGTCGGTTGTGTCTTC............ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:11306672-11306846 + | dvi_6102 | TATGGCACCGCCAGCTCCACCTAAAAGGGAGTTGTCTACTGGTAAAGCTGGTAAGTTTTGGA--------------------------------GAACTGCCAAAGTGTTGCCCCTGAAGTACAGTAAATGCATCT--TTCTAGTGCTGCGACGTCTAGGAAAACCTCTGCAGACTGATGTGCAGCCTACACAGAAGCGTCGGAAGCTC |
| droMoj3 | scaffold_6496:15617935-15618109 - | GATGGCGCCACCAGCAGTACCCAAATCGGAGCTATCTAGTGGTAAAGGGGGTAAGTATCTTGTATATATAT----AATAATATCAAAATTAATAAAACT------------------------------CTCATCCTTTCACAGTCGTGCGACGTCCAGGAAGGCTCATCAAGCTGAGTGATCAGCCCAAGCAGAAGCGTCGAAAGTTG | |
| droGri2 | scaffold_15245:13422828-13423006 - | AATGGCACCACCAGCACCGCCCAAAAGGGAGCTGTCCACCGGGAAGGCGGGTAAGTAGTGTATCAAATTATGCTTTCAAGTTTCAAATTCAAATATAAA------------------------------CCAATCTTATTCCAGTGCTGCGACGTATGGGCAAACCCATACAGGTGGATGTGCAGCCGTTAACAAAACGCCGCAAGCTT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/19/2015 at 01:47 PM