
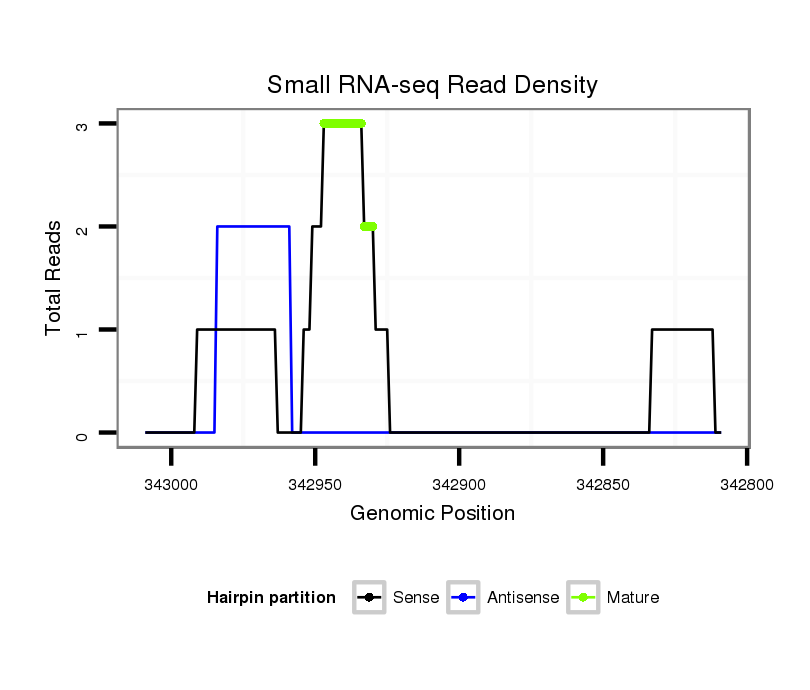
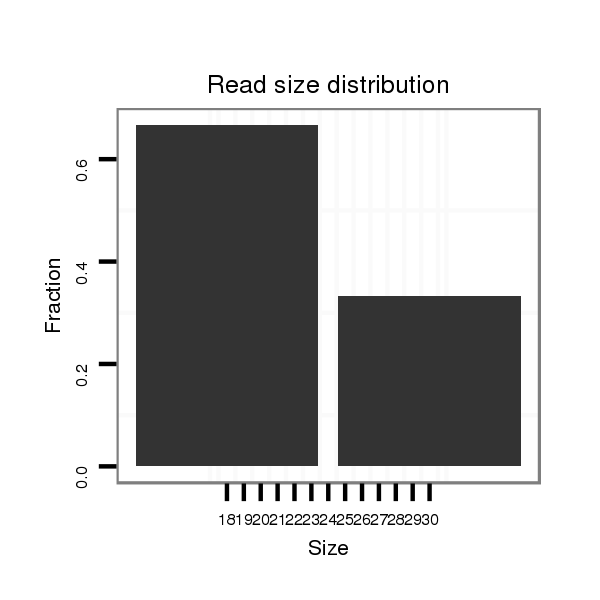
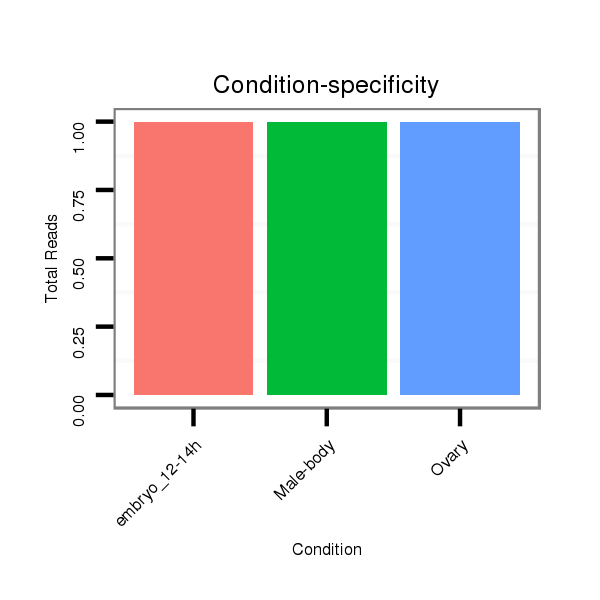
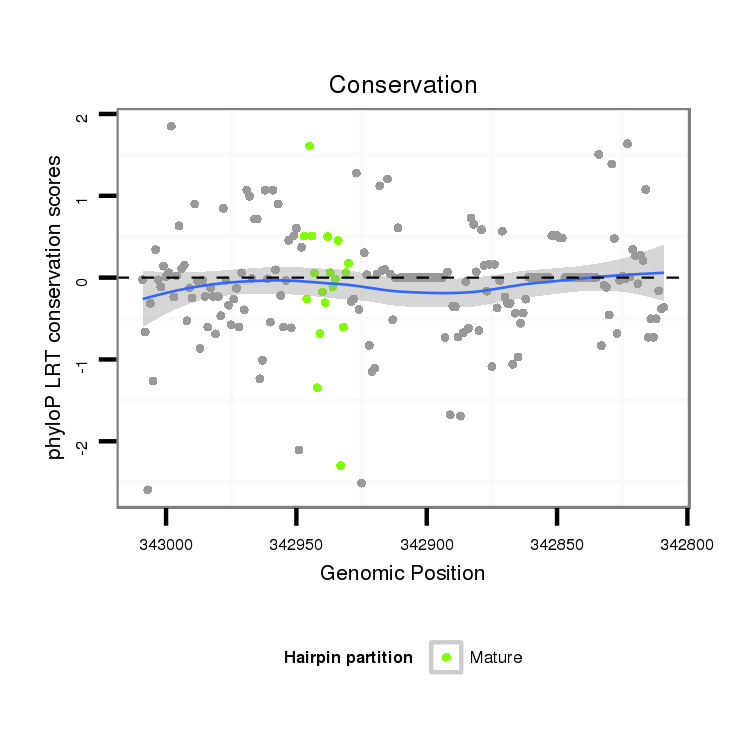
ID:dvi_604 |
Coordinate:scaffold_10322:342859-342959 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -17.6 | -17.2 | -17.1 | -17.1 |
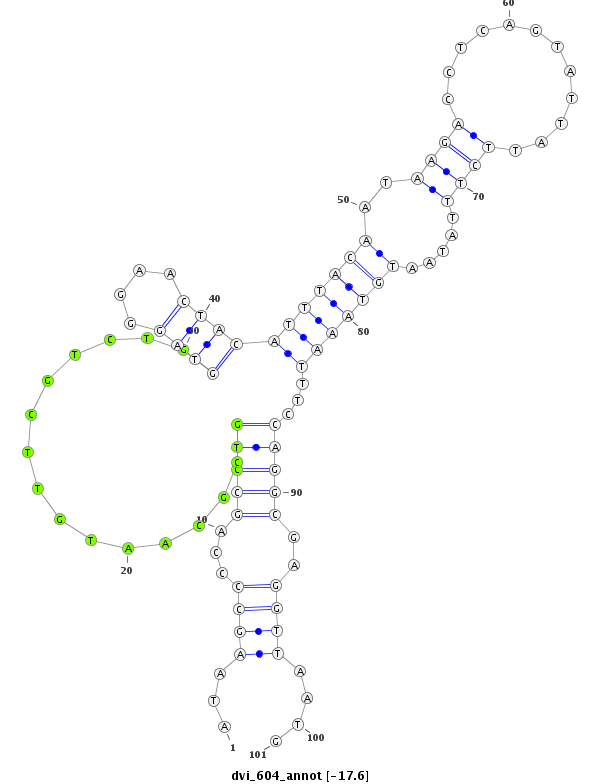 |
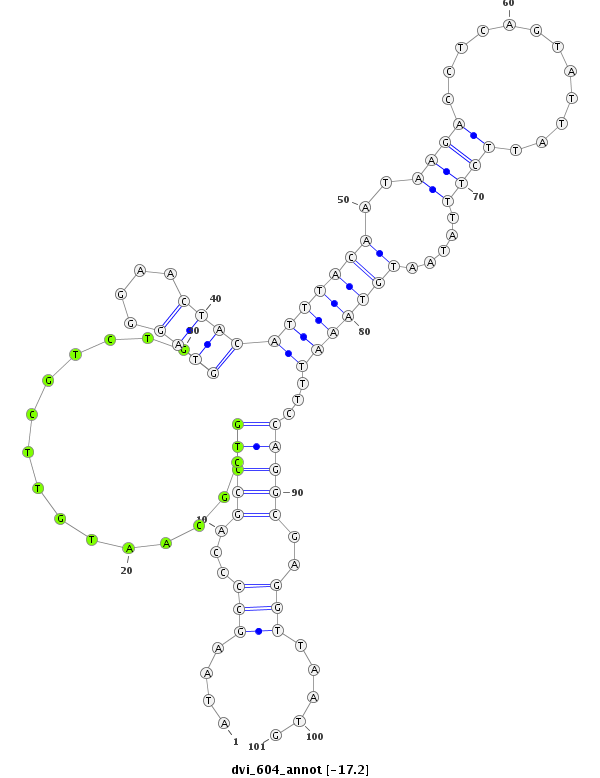 |
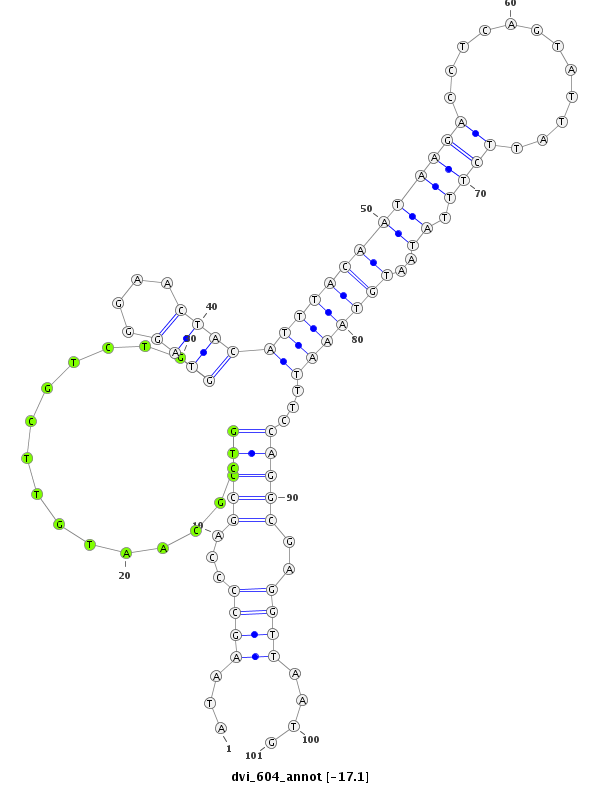 |
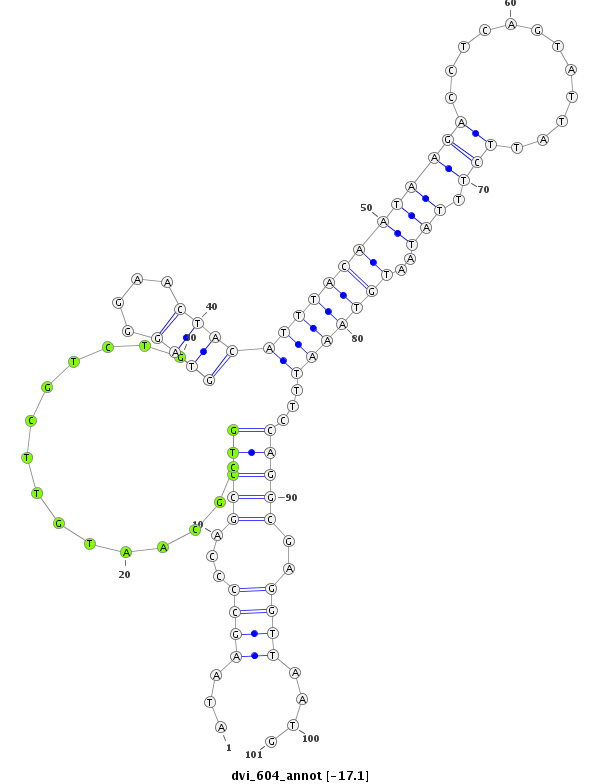 |
exon [dvir_GLEANR_17651:4]; exon [dvir_GLEANR_17651:3]; CDS [Dvir\GJ17121-cds]; CDS [Dvir\GJ17121-cds]; intron [Dvir\GJ17121-in]
No Repeatable elements found
| ##################################################-----------------------------------------------------------------------------------------------------################################################## TGCACATGATTTTGATGATCAACGACAAAAAGTGCTGGATTCGAACGTGGATAAGCCCCAGCCTGCGCAATGTTCGTCTGGTAGGGAACTACATTTACAATAAGACCTCAGTATTATTCTTTATAATGTAAATTTCCAGGCGAGGTTAATGGACTCGATTGGCCGAGCTCAAAGGAGGTAGGGTTAAATCTTTCTTGTGAT **************************************************.((.......(((..........((((((((.(((....((((((...(((((.((....)).)))))......))))))..))))))))))))))...))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060667 160_females_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR1106724 embryo_12-14h |
M061 embryo |
SRR060666 160_males_carcasses_total |
V053 head |
V047 embryo |
V116 male body |
M028 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................CTGCGCAATGTTCGTCTG......................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................GGTAGGGTTAAATCTTTCTTGT... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................CCCCAGCCTGCGCAATGTTCGTCTGGTAGG.................................................................................................................... | 30 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................CAGCCTGCGCAATGTTCG............................................................................................................................. | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................TCAACGACAAAAAGTGCTGGATTCGAAC........................................................................................................................................................... | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................AAGGTGGTAGGCTGAAATCTT......... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................GTACTGGATTCCAACGTGGC...................................................................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................AAGGTGGTAGGCTGAAATCT.......... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................CGTAGGGTTTAATCTTCCT...... | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................AGGTGGTAGGCTGAAATCT.......... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| .......GACTTCGATGAACAACGAC............................................................................................................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................................................................................CTCAAATCAGGTAGGGGTA............... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................................AGAGGACTCGCTTGGCCGA................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACGTGTACTAAAACTACTAGTTGCTGTTTTTCACGACCTAAGCTTGCACCTATTCGGGGTCGGACGCGTTACAAGCAGACCATCCCTTGATGTAAATGTTATTCTGGAGTCATAATAAGAAATATTACATTTAAAGGTCCGCTCCAATTACCTGAGCTAACCGGCTCGAGTTTCCTCCATCCCAATTTAGAAAGAACACTA
**************************************************.((.......(((..........((((((((.(((....((((((...(((((.((....)).)))))......))))))..))))))))))))))...))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060656 9x160_ovaries_total |
V116 male body |
M047 female body |
SRR060681 Argx9_testes_total |
SRR1106729 mixed whole adult body |
SRR060664 9_males_carcasses_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................GTTTTTCACGACCTAAGCTTGCACCT...................................................................................................................................................... | 26 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................TGTTGTTCTGGGGTCATATTA.................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................TCGGTCGCGTTACTAGCAG........................................................................................................................... | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................TAGCCGGCTCCCGTTTCCTC........................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................TCCACTTCCCTGAGCT........................................... | 16 | 2 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ........................................................................................................TGGAGTCATAATAAGTACAA............................................................................. | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................TCTTTGTGGAGTCATAATAA................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_10322:342809-343009 - | dvi_604 | TGCACAT------------------------GATTTTGAT--------------GATCAACGACAAAAAGTGCTGG--A--TTCG---AACGTGGATAAGCCCCAGCCTGCGC-------------AATGTTCGT-CTGGTAGGGAAC----TACATTTAC------------------------------------------------------------------------------------------------AATAAGACCTCAGTATTAT--TCTTT----------------------------------ATA-------------ATGTAAATTTCCA---------------GGCGAGGTTAATGGACTCGATTGGCCGAGCTCAAAGGAGGTAGGGTTA----AA---------TCTTTCTTGTGA-T |
| droMoj3 | scaffold_6680:2376466-2376732 - | TAGACCT------------------------GATTTGGAA--------------GATCAATGGGAGTGCGTA--ACAAG--CTCCCATAAGCTCAATAGCCCACATCCTGCGTGTATTCATGA----------------------AGGTAAGAACACTTTGTGAACTACAATTTATAAAATAATTTGTACATTTTATAATAGCTGTAG----CAAAA----T-AAAATTATCATA--------------------TTA--------------------TATAATTAATATTCACTAATTAATGTTCACTAAAATATTCATATACTATTTATAGTTGATGCCAACCAAC------CCGCTGAGATAGTTTCT------------------------------------------AA---------TATTTCGCTCGA-T | |
| droGri2 | scaffold_15110:183626-183719 + | TGTGAATAATCAGAAGAAGCCGAGGATACAAA----------------------------------------------A--CTCC---AATCTGGAT-------------------ACGCTGATAACT-GAAAGCGGA----GTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAATCGAATCG--------ATTGG-------------------AAACC-------------------------TT-T | |
| dp5 | XR_group8:937926-938096 - | TATAGAA------------------------GAAGTCGGT--------------GCGGATGAACCAGACGCAGAGG--ATGTTCCATT---ATGTACAGGT-----------------------AACA------------------------GGCGTTCTG-----------------------------------------------TAAA----------------------------TATCAGCTATC------A-----------------AACCTGC---------------------------TATTTAATGGCATTCTATTCACAGAAGATGATTACTTGCTTGGCACTGCTTGGACGCCAAACG--------------------------ATGCCGAGGTC-------------------------AA-C | |
| droPer2 | scaffold_32:537649-537819 + | TATAGAA------------------------GAAGTCGAT--------------GCGGATGAACCAGACGCAGAGG--ATGTTCCATT---ATGTACAGGT-----------------------AACA------------------------GGCGTTCTG-----------------------------------------------TAAA----------------------------TATCAGCTATC------A-----------------AAACTGC---------------------------TATTTAATGGCATTCTATTCACAGAAGATGATTATAGAGCGGACACTGCTTGGACGCCAAACG--------------------------ATGCCGAGGTC-------------------------AA-C | |
| droAna3 | scaffold_13337:584378-584538 - | AGTACAA------------------------GATCTCGAAGAGGGCGTGATGAGAT-----------------------T----------------------ACATCAAGATCGTAACCATGAAATAG-GTAAGCCCA----AGG-------CACGTTTAG-----------------------------------------------AATA----------------------------TGTAGGACACTCGCCTCT-----------------T--TCATT----------------------------------TCATTTTTACTTTCAGCTGAAGAATCC------------GCCT--------------------------------------------------GTAAGGACGACGGTTCTGT-----AA-C | |
| droBip1 | scf7180000395155:175729-175906 + | AGAACAGGAGGAGAACGCCCCGCAAGTACAAGATCTCGAAGAGGGCGTGATAAGGT-----------------------T----------------------GCACCAAGATCGAAACATTGATATAG-GTATGTCCA----TGGTGA--------------------------------------------------------------------------------------------TATAGGTATTCCGTTCCT-----------------TAATTGTT----------------------------------GCATTTGGTTTTTCAGTTGAAGAACCC------------GCCT--------------------------------------------------GTAAGGACGACGGATCCGT-----AACT | |
| droFic1 | scf7180000454105:216419-216585 - | CACGAAA------------------------AGCTTTCAAAACGTAATGGTGAGAT-----------------------T----------------------ACACCAGGATAGTAACGAAGGAACTTGTTCAGG-TATAGACTGAGA----------------------------------------------------------------CAAGA----TTAATGCTAACATACTTCT-----------------A-----------------AAATAGAA-----------------------------------TAAATATCTTCGCAGATAAGGACTGCCG---------------ATATTCAGACG--------------------------ACGGGGACACCGTC----------------------AA-G | |
| droRho1 | scf7180000762853:62125-62293 - | AAGAGGT--------------------------------------GATAGCGAGGA-----------------------T----------------------TAACGAGGATGGTAATGACGAAATTAGGCCAGG-TATGGATCAAAT----------------------------------------------------------------TAGAGTAACATGGTGGTAGCGAGCTTTC-----------------A-----------------CAACAGAC---------------------------------------TTCCTTCGCAGATAAGGACTGTAG---------------AACGTCAAACG--------------------------ATAGTGAAACCATCAAGGACAA--GTCCCGAC----TG-T | |
| droBia1 | scf7180000300910:166572-166579 - | CTTTCAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------C | |
| droEug1 | scf7180000409184:155763-155918 - | CCTAGAA------------------------GTCTTTGAAGACGGTGTACACAGGT-----------------------T----------------------ACATGAGGAAGGTATTTATGAAATTAGGTCACA-TTTAAATAAAAT----------------------------------------------------------------TACGC------------------------------------TTTTA-----------------TAATGAAC----------------------------------TTACTTTTATTTTTAGATAGGGACTTCCG---------------AACGTTCATCG--------------------------ATGGTGACACCGTCAAAA-------------------A-T | |
| droYak3 | 3L:159733-159873 + | CAG----------------------------GACTTTAAAGAGAAGGCACAGGGGA-----------------------C----------------------ACAGGAAGATCGGAACTTTGAGGCAATGTCAGG-CATGGATGAGGA----------------------------------------------------------------TAGGC------------------------------------CT--------------------------------------------------------------------------CTAGATAGAGCCTTCCG---------------ATCGGCGTTAA--------------------------ACAGTGACACTGGCCAGGACGA--GATCCGAC-----T-T | |
| droEre2 | scaffold_4784:170207-170350 + | CATTGAA------------------------TACTTTAAAGAGAAGCCACAGGAGA-----------------------T----------------------ACCCGAAGATCCAAACTTTGAGGCAATGTCAGT-TATGGATCAAAA----------------------------------------------------------------TAGGG------------------------------------AT--------------------------------------------------------------------------CTATCTAGGGCCTTCCG---------------AACGGCGTTAA--------------------------ATAGTGACACTGGCCAGGACGA--GATCCG------AG-T |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 10:45 AM