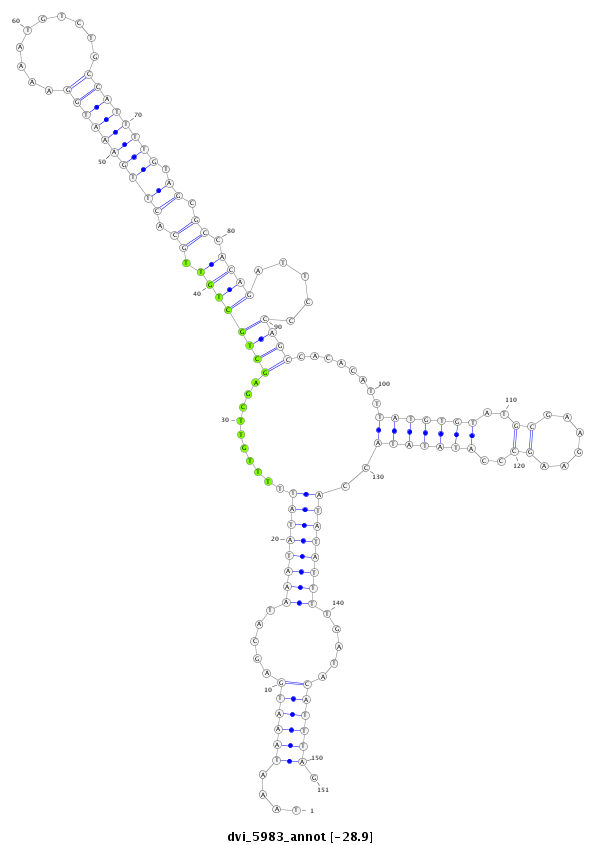
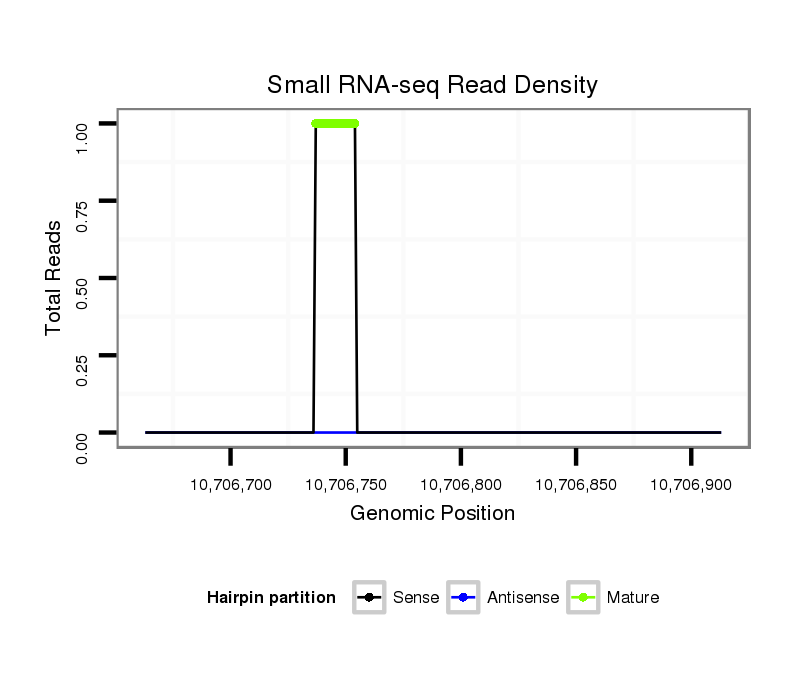
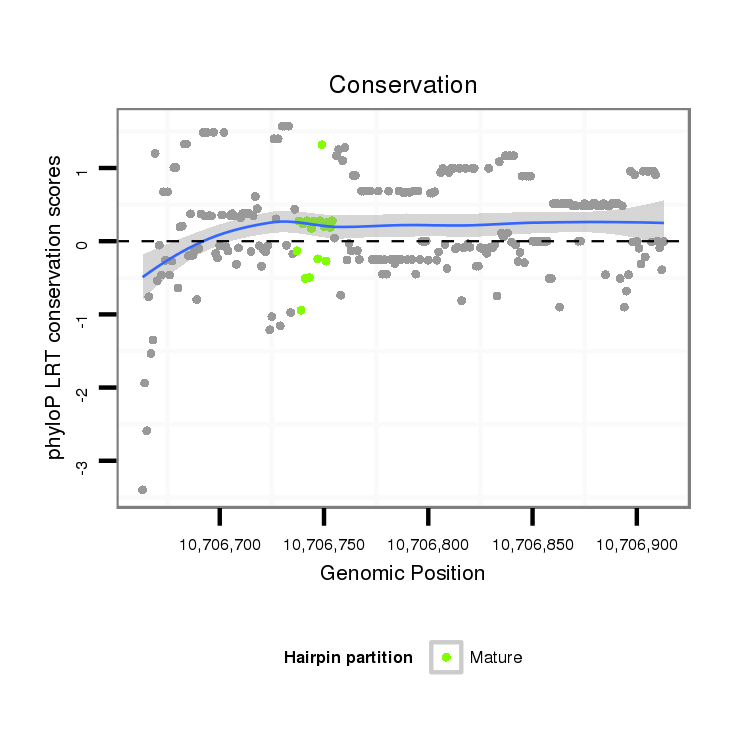
ID:dvi_5983 |
Coordinate:scaffold_12875:10706713-10706863 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -28.8 | -28.7 | -28.6 |
 |
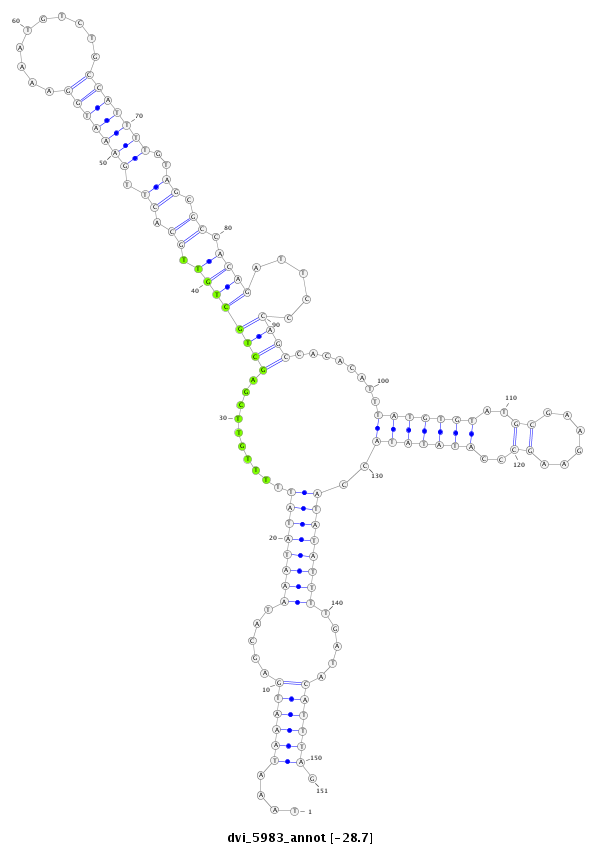 |
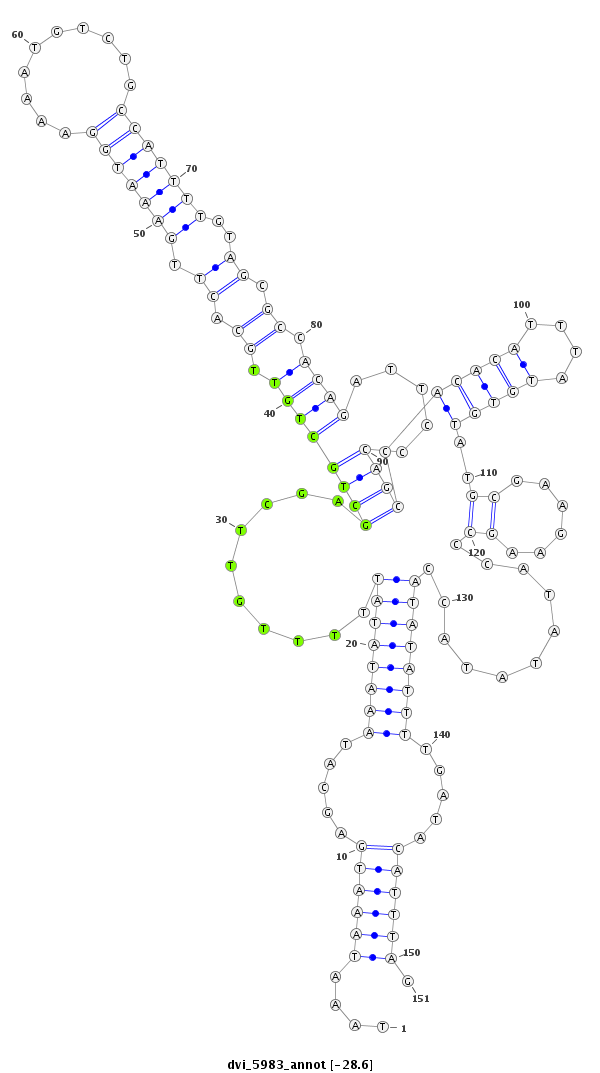 |
exon [dvir_GLEANR_7108:3]; CDS [Dvir\GJ21779-cds]; intron [Dvir\GJ21779-in]
No Repeatable elements found
| mature | star |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGCCATATTGTCAGTTTAATTTGTCTTAAATATTAAAGTTCTTAACTTTATAAATAAATGAGCATAAATATATTTTTGTTCGAGCTGCTGTTGCACTTGAAATGGAAAATGTCTGCCATTTTGTAGCGCCACAGATTCCCAGCCACACATTTATGTGTATGCGAAGAAGCCCATATATACCATATATTTTGATACATTTAGAAAATGGCTTCTCGCATCCCTCGCAAAATCGTGATGCTCAGATTTGCGAC **************************************************....((((((.....((((((((..........((((((((.((.((((((((((..........)))))))).)).)).)))).....))))........(((((((..((......))..)))))))..)))))))).....)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR1106729 mixed whole adult body |
M061 embryo |
V053 head |
SRR1106727 larvae |
V047 embryo |
SRR060684 140x9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR1106728 larvae |
M047 female body |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................................................................................CGCAAAGTCGTGAGGCT............ | 17 | 2 | 15 | 1.67 | 25 | 0 | 17 | 0 | 0 | 6 | 0 | 0 | 0 | 2 | 0 | 0 |
| ..........................................................................TTTGTTCGAGCTGCTGTT............................................................................................................................................................... | 18 | 0 | 2 | 1.50 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................TGGCTTCTCGCTTCCCTCCCC......................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................TTTATGTGTACGCCAAGAATC................................................................................. | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................GCGAAGTCGTGATGCTAAG......... | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................GTATGCGACGAAGCCTAGA............................................................................ | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................GTTGCACTTGAAAGGGAAG............................................................................................................................................... | 19 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................TTGATACAACTACAAAATGGC.......................................... | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................................................................................................TCGCAAAGTCGTGAGGCT............ | 18 | 2 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................CTCGCGAAGTCGTGACGCT............ | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................CTGCACTTGCAATGGAAAA.............................................................................................................................................. | 19 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................CACATGAACTGGAAAATG............................................................................................................................................ | 18 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACGGTATAACAGTCAAATTAAACAGAATTTATAATTTCAAGAATTGAAATATTTATTTACTCGTATTTATATAAAAACAAGCTCGACGACAACGTGAACTTTACCTTTTACAGACGGTAAAACATCGCGGTGTCTAAGGGTCGGTGTGTAAATACACATACGCTTCTTCGGGTATATATGGTATATAAAACTATGTAAATCTTTTACCGAAGAGCGTAGGGAGCGTTTTAGCACTACGAGTCTAAACGCTG
**************************************************....((((((.....((((((((..........((((((((.((.((((((((((..........)))))))).)).)).)))).....))))........(((((((..((......))..)))))))..)))))))).....)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060669 160x9_females_carcasses_total |
V053 head |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|
| ....TATAACACTCAAATTAAA..................................................................................................................................................................................................................................... | 18 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| ...........................................................................................................................................................GCATACGCTTCCTCGGGTGT............................................................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 |
| .......................................................................................................................AAACATTGCGATGTCTAAAG................................................................................................................ | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:10706663-10706913 + | dvi_5983 | TGCCATATTGTCAGTTTAATTTGTCTTAAATATTAAAGTTCTTAACTTTATAAATAAATGAGCA----------TAAATA-----TATT-TTTGTTCGAGCTGCTGTTGCACTTGAAATGGAAAATGTCTGCCATTTTGTAGCGCCACAGATTCCCAGCCACACATTTATGTGTATGCGAAGAAGCCCATATATACCA-TATATTTTGATACATTTAGAAAATGGCTTCTCGCATCCCTCGCAAAATCGTGATGCTCAGATTTGCGAC |
| droMoj3 | scaffold_4164:3203-3297 + | TCAGTCAA----------ATTTGTAAAAAACAACAAAGTTCAAAGCTGTATAAAAAAAAAATAA----------AACATTTTATCTTATTTCATCTCGAAATGCCATT-----TGAAATG---------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15245:3815256-3815366 + | GCAAC-----------------A-------------------------------TCTCTATATATATATCTATATATATA-----TATC-T-------------------------------------------------------------------------------------AT--------ACATATATACCAATGTAT--------TATTACAAAATGGC--CTCGCATCCCTTGCAAAAACCGAATGCTTAGATTTGCGTC | |
| droWil2 | scf2_1100000004822:3487650-3487772 + | TATAA-----------------A-------------------------------TATATATATA----------TATATT-----TTTT-ATTGTCAGAGTCGCAGTCGCAGTTCGCATTAAAAATACTTAATCTCCAATGGCACTAAAA--------ACACACATACACATACATACATAGAGACACGGACACA------------------------------------------------------------------------- | |
| dp5 | XL_group1a:4181204-4181259 + | TATTTTATTTTTATATTTTTTTGTATAAAATATTATTTTTTTTAACTTTATATGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_22:1067569-1067627 + | CCCCTAATT------------------------------------------------------------------------------------------------------------------------------------------------TCCAACCATCCATATAGATGTATGTATGTAAATCCATATACATGC-TATAT---------------------------------------------------------------- | |
| droKik1 | scf7180000302621:296238-296275 + | AATTT-----------------GTCTTTAATATTAAAATTCATTAAATTATTAAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491214:3813527-3813570 - | AAATA---------------------------------------------------AATGACAA----------TAAATA-----TTAT-TTGGCTAGAGTTGTTGCTTCA------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000770948:55752-55817 - | TATTATACA-----TTTAATTTGACTTTTATATTAGCAGTTTTAATTTTAAAAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGTGTACATATGTTTT | |
| droTak1 | scf7180000414560:184671-184720 + | CATTTT-----------TACTTTTCCAAAATATTAAAATTTTTAAAATCGTATATAAGCGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4845:8428264-8428300 - | GCATG-----------------------------------------------------------------------TATA-----TGTT-GTTGTTTCGGTTGCTGTTGCATTT---------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:08 AM