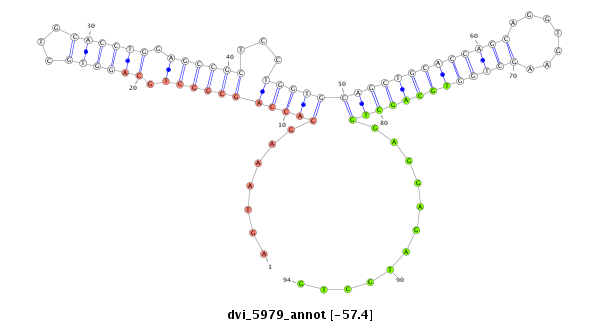
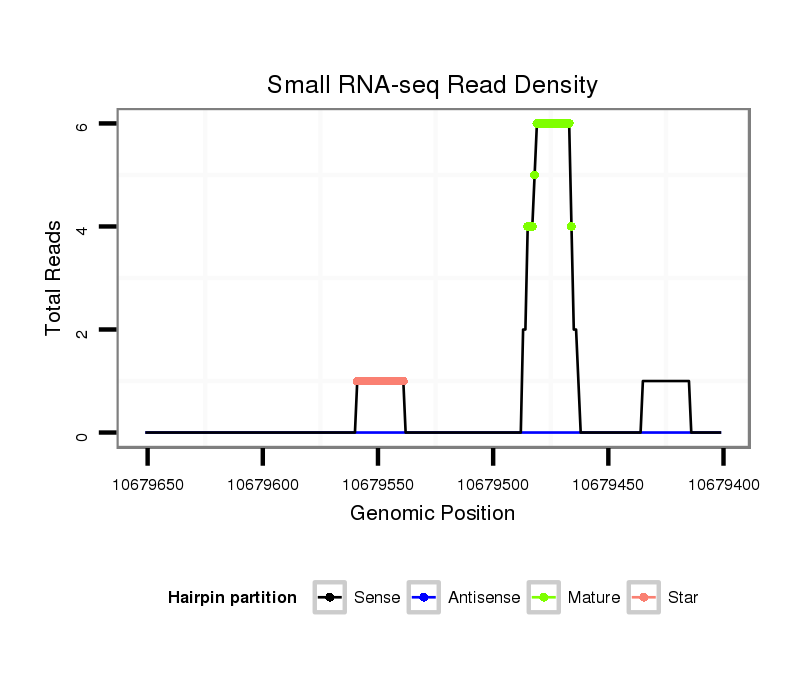
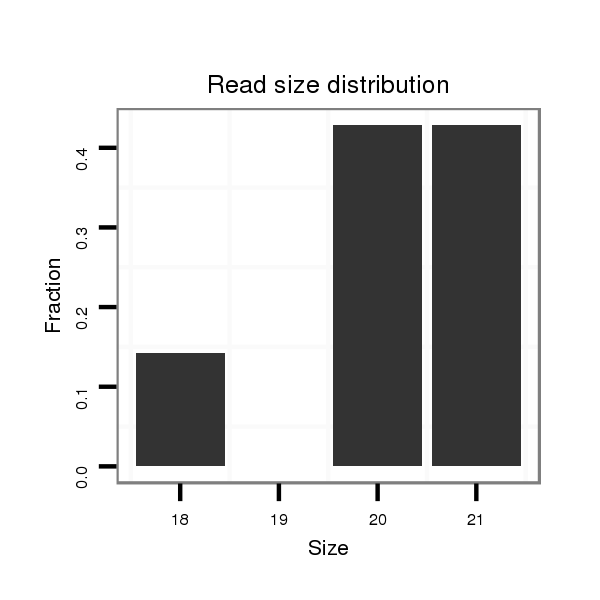
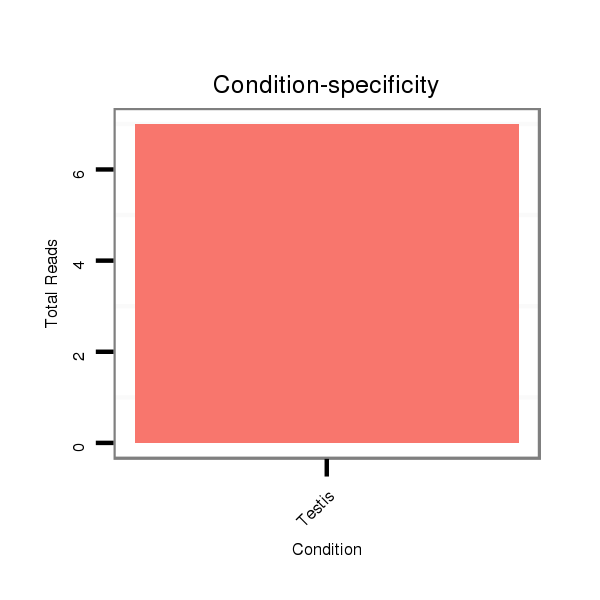
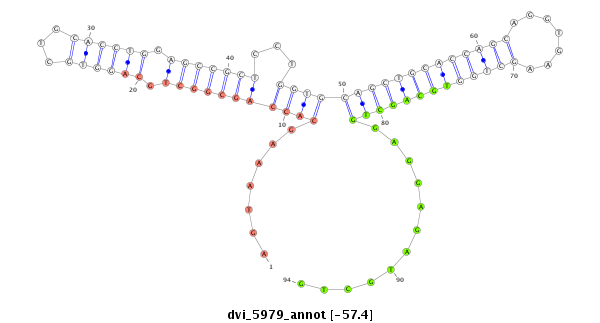
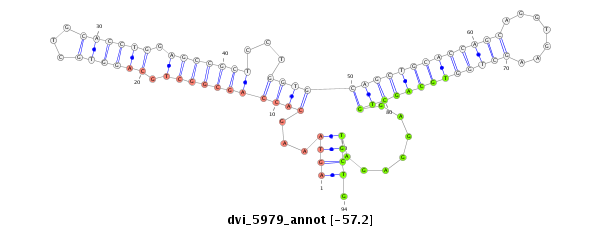
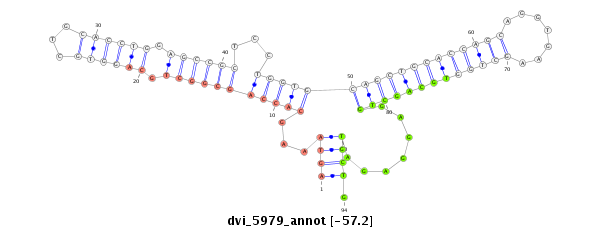
ID:dvi_5979 |
Coordinate:scaffold_12875:10679451-10679601 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
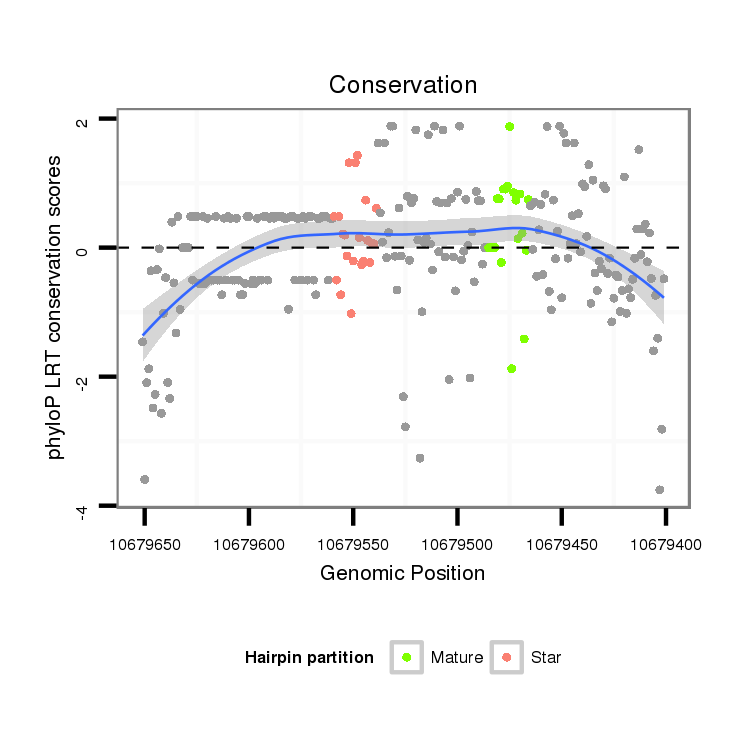
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -57.4 | -57.2 | -57.2 |
 |
 |
 |
CDS [Dvir\GJ20562-cds]; exon [dvir_GLEANR_5993:2]; intron [Dvir\GJ20562-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AATGGAGAAAGGAGGTAACCAACAAATATTTGAATTGGTCGAGATGCACTTATGTGTCAATGTGTTTAGCGAACGGGTGTAAAACCAAAAGCAGTAAAGCACCAGCGGCTGCAGGTGCTGCACCTGGAGCCGCTCCTGGTGCAGCTGCACCAGCAGGTGAAGCTGGTGCAGCTGGAGGAGATGCTGCAGCGCCGGCCGCAGGAGAAACTGCAGCATCTCGAACTGGGGCCAGTGAAGGAACGGAACTTCTA ********************************************************************************************.......(((((((((((.((((((...)))))).))))))...)))))(((((((((((((.......)))))))))))))............***************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060659 Argentina_testes_total |
SRR060679 140x9_testes_total |
M027 male body |
SRR060657 140_testes_total |
SRR060680 9xArg_testes_total |
SRR060683 160_testes_total |
SRR060675 140x9_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
V053 head |
SRR060669 160x9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060667 160_females_carcasses_total |
V116 male body |
V047 embryo |
SRR1106728 larvae |
GSM1528803 follicle cells |
SRR060663 160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................TGCAGCTGGAGGAGATGCTG................................................................. | 20 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................GGTGCAGCTGGAGGAGATGCT.................................................................. | 21 | 0 | 1 | 2.00 | 2 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................GCTGGAGGAGATGCTGCA............................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................AGTAAAGCACCAGCGGCTGCA.......................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................GTCCAGTGAACGAACGGAACT.... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................AGCTGGAGGAGATGCTGCAG.............................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................CGGCCGTAGGAGAACCTGCGGC..................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................CTCGAACTGGGGCCAGTGAAG.............. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................GCAACAGCAGGTGAAGCCGGT.................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................CAGGTGAAACTGCAGCAGCTCG............................... | 22 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................AACAGCAGGTGAAGCCGGTG................................................................................... | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................CTGCATCTGAAGCCGCTCC................................................................................................................... | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................GGAGCCGCTCTTGGTCCCGCT......................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TACTGCATCTGAAGCCGCTC.................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................ACTGCATCTGAAGCCGCTCCT.................................................................................................................. | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................CTGCATCTGAAGCCGCTCCTC................................................................................................................. | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................................................GCATGGAGCCGCTCCTGGT............................................................................................................... | 19 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................TCTCGAACTGGGGCT..................... | 15 | 1 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................................................................................................................................AACTGCAGCTTCTAGCACTG.......................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................GAGATGTTGCAGTGACGGC....................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................................GCTGCAGGATCTGCACCTGAA........................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TTACCTCTTTCCTCCATTGGTTGTTTATAAACTTAACCAGCTCTACGTGAATACACAGTTACACAAATCGCTTGCCCACATTTTGGTTTTCGTCATTTCGTGGTCGCCGACGTCCACGACGTGGACCTCGGCGAGGACCACGTCGACGTGGTCGTCCACTTCGACCACGTCGACCTCCTCTACGACGTCGCGGCCGGCGTCCTCTTTGACGTCGTAGAGCTTGACCCCGGTCACTTCCTTGCCTTGAAGAT
*****************************************************************.......(((((((((((.((((((...)))))).))))))...)))))(((((((((((((.......)))))))))))))............******************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060672 9x160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
M047 female body |
SRR060664 9_males_carcasses_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................CGTGGAGCTCGGCGAGGACACC.............................................................................................................. | 22 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................TTGCCCTCATTCTGGTTTGC................................................................................................................................................................ | 20 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................TTGCCCTCATTCTGGTTTG................................................................................................................................................................. | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................ACCAATCGCTTGCCCAAC........................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................................................................................CTTCTGTGACGTCGTCGAGC............................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................TGGGCGAGGACCATATCGA......................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:10679401-10679651 - | dvi_5979 | AATGGAGAAAGGAGGTAACCAACAAATATTTGAATTG--------GTCGAGATGCACTTATGTGTCAATGTGT-TTAGCGAACGGGTGTAAAACCAA---------------------------AAGCAGTA-----------------AAGCACCAGCGGCT------------------GCAGGTGCTGCACCTG------------------------GAGCCGCTCCTGGT------------------GCAGCTGCACCAGCAGGTGAAGCTGGTGCAGCTGGAGGAGATGCTGCAGCGCCGGCCGCAG---------------------GAGAAACTGC------------------AGCATCTCGAACTGGGGCCAGTGAAGGAACGG---------------------AACTT--------CTA |
| droMoj3 | scaffold_6496:16279936-16280173 - | AATGGACAAGGGAGGTAAG----AGATTATAGGGCTACCAATTCAAGCAAGGCGTCAATAAAAGCTAACGTGTCTTAGCCAATGGATGCAAGCCCAAGCCCCCAGCAGCCAAACAGGATAAGCCAGGCAATC-----------------CAGCGCCAAATAC---------AAGAAACG---------ACGCAGCAA------------------------AGCCAGACCCAGAT------------------GCGGCAGGAGGAGGAGGAGCA---------------GGAGGAGGAGGTGGAGGAGGAGGAG---------------------GAG------------GAGCAGGAGGAG---------------------------------------------------------GA--------GGA | |
| droGri2 | scaffold_14853:9674572-9674748 - | GCAGGAGCAGCAGC----------------------------------------------------------------------------------------------------------------------AGCAG------------CAGCAACAGCAGCA------------------GCAGGAGCAGCAGCAG------------------------CAGCAGCAACAGCA------------------GCAGCAGGAGCAGCAGCAGCA---------------GCAGCAACAGCAGCAGCAGGAGTAG---------------CAGCAGC---AGCAG---CAGCAGGAGCAACAGCAGCAACATGGACAACAACAAGAAGCAGACAACAAC---------------------GA--------GCA | |
| droWil2 | scf2_1100000004513:6375678-6375703 + | GCAAGGGAAAAAGC----------------------------------------------------------------------------------------------------------------------GGTGA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TATT--------GAA | |
| dp5 | 3:8774209-8774396 - | CCGAAGGAGGAGCAGCT------------------------------------------------------------------------------------CC-----------------------------AGCGGCCGAAGGAGAAGCTACGCCAGCGGCC------------------GAAGGAGAAGCTACGCCAGCGGCCGAAGGAGAAGCAGCTCCAGCAGCCGAAGGA------------------GATGCAGCTGCAGCGGCCGAA---------------------------------------G---------------------GAGATGCAGCTGCAGCGGCCGAAGGAG---------------------------GAGCAGCTCCAGCGGCCGAAGGAGAAGCAACT--------CCA | |
| droPer2 | scaffold_17:667417-667547 - | CAGGAGCAGCAGCAGGA--------------------------------------------------------------------------------------------------------------------------------------------GCAGCC------------------GCAGGAGCAGCAGCAG------------------------GAGCAGCAGCAGGA------------------GCAGCCGCAGGAGCAGCAGCA---------------GGAGCAGCAGCAGGAGCAGCCGCAG---------------------GAGCAGCAGCAGCAGCAGGAGCAGCCG---------------------------------------------------------CA--------GCA | |
| droAna3 | scaffold_13340:4422241-4422389 - | AACAGCT------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGAGCAGCCACTGGAGCTGCTACTGGAGCAGCCACAG------------------------AAGTAGCTTCTGGA------------------GCGACCAGAGGGGCGGCCACA---------------GGGACAGCTGCAGGAGCAGCCACTG---------------------GAGCTGCTACTGGAGCCGGTTCAGCAA---------------------------------CGGCTGG--------------AACTA--------CTG | |
| droKik1 | scf7180000299625:16770-16890 - | AGCCGCA------------------------------------------------------------------------------------------------------------------------------------------------------GGAGCAGCAGCCGCAGGAGCTG---------GAGCCGCAC------------------------CATCAGCCGCAGGA------------------GCAGCAGTAGCAGGAGCAGCA---------------GCAGCAAGAGCTGGAGCAGCAGCCG---------------------CAG---------------------------------------------------GAGCAGCAGCAGC---------------TGCT--------CAA | |
| droFic1 | scf7180000454072:2428188-2428343 - | AGGACCA------------------------------------------------------------------------------------------------------------------------------------------------------ACAGCAACAACAGCAC------ATTCAGATGCAGCAGCAG------------------------CATCAGCAACAGGT------------------GCAGCAGCATCAGCAACAAATGCA------------GCAGCAGCAACAGGAGCAGCAGCAG---------------------CAGCAGCAGCAACAACAACAACTGCAA---CAGCAACAACTGGTGTCCGC----------------------------------GG--------AGG | |
| droEle1 | scf7180000491001:258025-258161 - | CCTAAAACAACTGG-----------------------------------------------------------------------------------------------------------------------------------------ACATCAGGAGCA------------------GCAGGAGCAGCA---------------------------------------GGTGCAGCAGGAGCAGCAGGTGCA------GCAGGTGCAGCA------------GGAGCAGCAGGTGCAGCAGGAGCAGCAG---------------------GAGCAGCAGA----------------A---CAGC--------CTAG-----------------------------------AGTTGCCTGTGCAGAA | |
| droRho1 | scf7180000779776:33294-33458 - | GAGCGGCTCCAGCAGCG--------------------------------------------------------------------------------------------------------------------------CAAGGAGCAGCAGCTCCAGCAGCT------------------GAAGGAGCAGCAACTC------------------------CAGCAGCTGAAGGA------------------GCAGCAGCACCAGCAGCCGAA---------------GGAGCAGCAGCTCCGGTAGTCGAAG---------------------GAGCAGCAGCTCCAGCCGCTGAAGGAG---------------------------CAGCAAATCCTGCAGC--------------TG--------AGG | |
| droTak1 | scf7180000415389:225779-225795 + | ACTGGAGAAAGGCCGTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409672:2421431-2421448 - | GATGGCAAAG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATT--------CTT | |
| dm3 | chr2R:12714568-12714584 + | AGGGTACAGAGATGGCT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 2r:13385701-13385717 + | AGGGTGCAGAGATGGCA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_1:10219469-10219485 + | AGGGTGCAGAGATGGCA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | X:17615979-17616170 - | GAGCAGCTGTGGAATCA-----------------------------------------------------------------------------------------------------------------------------------------------GGAGCAGCAGGAG------ATGCAGGAGCAGCAGGAG------------------------AAGCAGGA------------------------GCAGCAGGAGAAGCAGGAGCA---------GCAGGAGAAGCAGGAGCAGCAGGAGAAGCAGGAGCAGCTGGAGAATCAGGAGC---AGCAGGAACAGCAAGAGCAGCTGGAGAAT---------CAAGAGGATCCGGAGCAGGAGGAGAAGC--------------TG--------AAC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/17/2015 at 03:55 AM