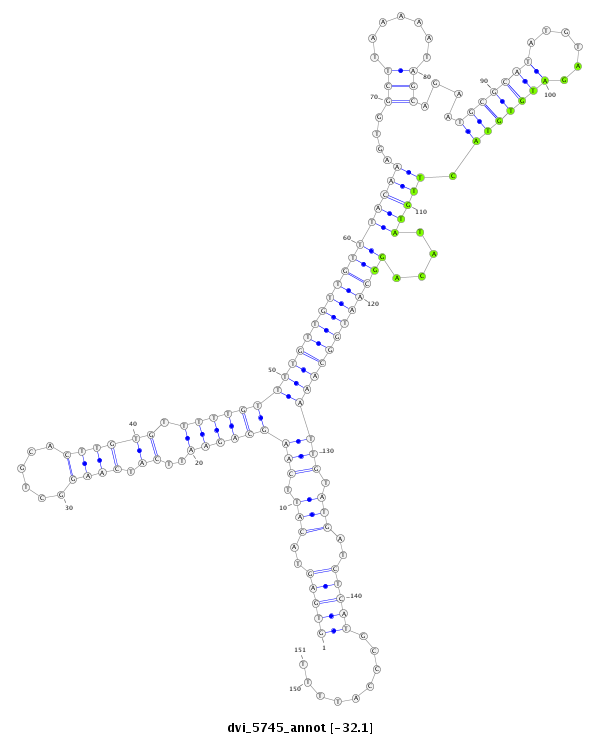
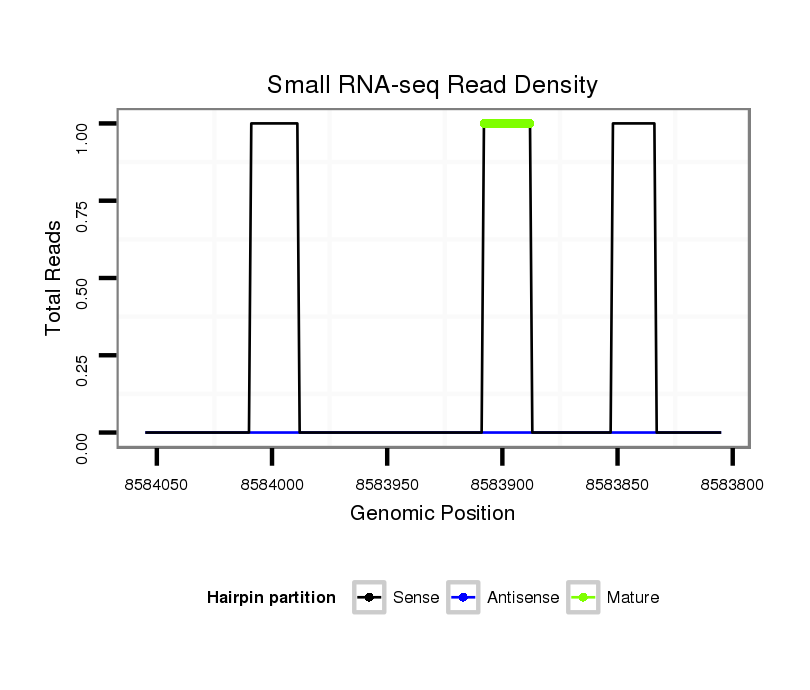
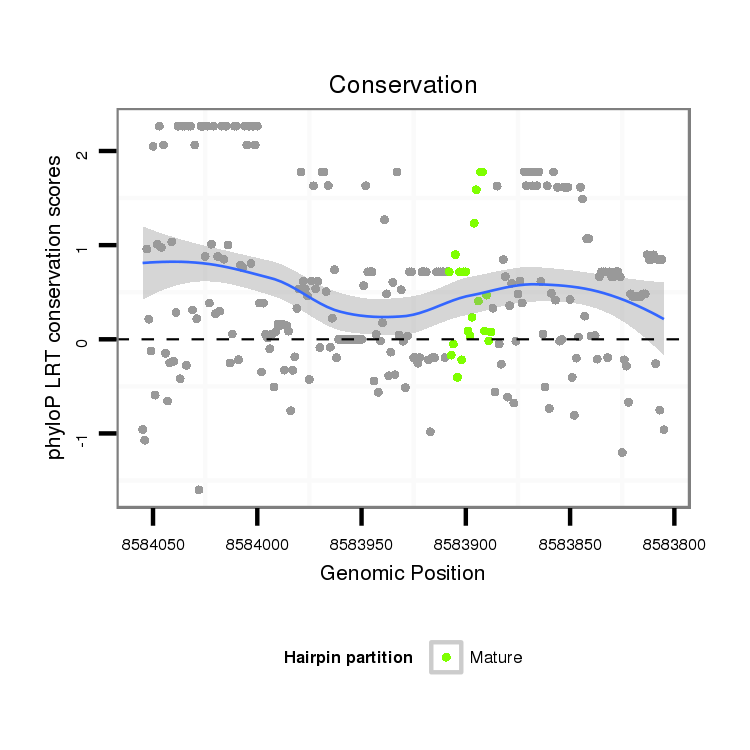
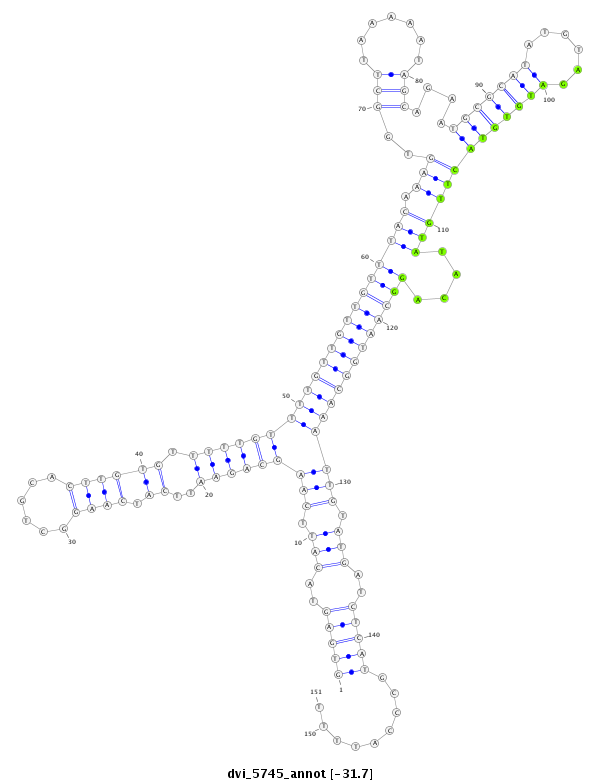
ID:dvi_5745 |
Coordinate:scaffold_12875:8583855-8584005 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -31.7 | -31.7 | -31.4 |
 |
 |
 |
CDS [Dvir\GJ20680-cds]; exon [dvir_GLEANR_6100:1]; intron [Dvir\GJ20680-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TTTGTTATTTGGCATTATAATTATCGGGTACAGAATCATCACTTTAACAAGTGAGTACATTCAAGCAGAATTCATCAAGGCTGCACTTGTGTTTTTGTTTTGTTGTTGTTTACAAAGTGGCTTAAAAATAGCAGAATGCGCATATGTAGATGTGTACTTGTATACAGGCAATGGCAAATTGTATGATCTCATGCCCATTTTCATTCTCATTGCTTTTGTGTTTCACCTTCGTAGTGCGCCTTTCCAGCGCT **************************************************(((((..(((.(((((((((..((.((((......)))))).))))))(((((((((((((((((....(((.......)))....(((((((......))))))).)))))....))))))))))))))).)))..))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060672 9x160_females_carcasses_total |
M027 male body |
M047 female body |
SRR060688 160_ovaries_total |
SRR060666 160_males_carcasses_total |
V053 head |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................GAATCCATCAAGGCTGCTC..................................................................................................................................................................... | 19 | 2 | 2 | 1.00 | 2 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................AGATGTGTACTTGTATACAGG................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TTCTCATTGCTTTTGTGTT............................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................ACAAGTGAGTACATTCAAGCA........................................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................CTGAATCCATCAAGGCTGC....................................................................................................................................................................... | 19 | 2 | 3 | 0.67 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................TTGTGTGTTTCACCTTCGC................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................CTGAATCCATCAAGGCTGCTC..................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................GAATCCATCAAGGCTGCTCA.................................................................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................TGTGTACTTGTAGCCAGGCC................................................................................. | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AAACAATAAACCGTAATATTAATAGCCCATGTCTTAGTAGTGAAATTGTTCACTCATGTAAGTTCGTCTTAAGTAGTTCCGACGTGAACACAAAAACAAAACAACAACAAATGTTTCACCGAATTTTTATCGTCTTACGCGTATACATCTACACATGAACATATGTCCGTTACCGTTTAACATACTAGAGTACGGGTAAAAGTAAGAGTAACGAAAACACAAAGTGGAAGCATCACGCGGAAAGGTCGCGA
**************************************************(((((..(((.(((((((((..((.((((......)))))).))))))(((((((((((((((((....(((.......)))....(((((((......))))))).)))))....))))))))))))))).)))..))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060686 Argx9_0-2h_embryos_total |
V116 male body |
V047 embryo |
SRR060679 140x9_testes_total |
SRR060670 9_testes_total |
SRR060666 160_males_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................GTAAGTTCGTCGTAAGTAGTT............................................................................................................................................................................. | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................GTCTTACGCGTGTACATCTAC................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................CCCATGTCTTAGTAGTGAGGT............................................................................................................................................................................................................. | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................AACCACAAATGGCTCACCGAAT............................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................GTAGCGAAATTACTCACTCAT.................................................................................................................................................................................................. | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................ATCTCGCGGAAAGGCTGCG. | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................TAACGTACTAGGGTACGTGTA..................................................... | 21 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................AAAGTAAGAGTACCCAAA................................... | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................ATGGTAGTGAGATTGTTCA....................................................................................................................................................................................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:8583805-8584055 - | dvi_5745 | TTTGTTATTTGGCATTATAATTATCGGGTACAGA------ATCATCACTTTAACAAGTGAGTACATTCAAGCAGAAT-----------------------------------------------------------------------------------------------------------------------------------TCATCAAGGCT---GCACTTGTGTTTTTGTTTTGTTG-TTGTTTACAAAG----------------------------------------TGGCTTAA-AAATAGC--AGAATGCGCATATGTAGAT--------------------G------TGTACTTGTATACAGG----CAATGGCAAA-----------TTGTATGATCTCATGCCCA-----TTTTCATTCTCATTGCTTTTGT---GTTTCACCTTCGTAGTGCGCCTTTCCAGCGCT |
| droMoj3 | scaffold_6496:18623136-18623393 + | CAC---ATTTGTCATTCTAATTATCGTCTACAAA------ATCATAATTTTAGCAAGTGAGTATATTCATT---------------------------------------------------------------------------------------------------------------------------------------------CAAGGTA---GCACTTGTGT--------------CTGTTTCCC----------------------ATTTGATTGTGTTGTTTAGAGTAGTGTAA-AAATAGC--GGAATATGCATATGTA----CTTATGCACAGATGTTTTTACATACATATATGTGTATATGGA-----------CAA-----------TTGAATGATCTCATAACGA-----TTTCTATTCGTATTGCTTTTGT---GTTTCACCTTCATGTTGCGCCTTTCCAGCACG | |
| droGri2 | scaffold_15245:1993079-1993340 - | ACTCCTATTTGCCATCATAATTATCGGCTACAGACTTATCATCATCATGTTAGCAAGTGAGTAAATGTTTT---------------------------------------------------------------------------------------------------------------------------------------------TATACTTGTTGTTATTT-----------------TTGTTTACCAAGTGACCTAT-------GTCAGTTTGCA---------------AATGTGA-ATATAATATAAAATGAATATATATAAATATATATATATAAACTATTATAC-TATTTGTACTTGTATAT--G----CAATGG-CAA-----------GTGTATGATCTCATACCGA-----TTTTCATTTGTATTGTTTATACGTCGTTTTACCTTCCAGT----------------- | |
| droWil2 | scf2_1100000004513:5765020-5765265 - | CATTATCTTTGCTATACTTATAATTGCCTATAAA------AGTGTCATTCTAACAAGTGAGTAAACCCCCC----------------------------------------------------------------------------------TCGATTTGAGGAGTGGGAGGTGGAGTGTGGCAGTGGGTGGGCATTAGTTGAAGTAAACTCAAGGTT---GCATTTGTTT--------------GTG---TTTA-----TATGA-------ATATAAATGTA---------------CATATTT-G------------------------GTT--------------------G--------------TATAAATATAGCCCAACGCAAA-----------CGTTATGATCTCACCTCAACGCATTTTCCATTCCTCCTG---------------------------------TCCACCTCC | |
| dp5 | 3:6572237-6572461 - | GCTTTTTCTGGGTATACTCATCATTGGATACAAG------AGCGTCATTCTAAGCAGTAAGTGTGTCAAGTTGATGGAGAAGCAGGATG------------------------------------------------------------------------GGGAGGGGGA-------------------------------GAAGCGTTCCCAAGGCT---GCAATTGTCG--------------GTG---ACGTCGTTGCCTAA-------GCTTATTTGTT---------------TGTGTTT-GCC-------------------------------------------------------------TTATAAATAGCCAA----G-AAATGTGTGTGTTTGTGTGATCTCATAGCCA-----TTTTCATTCCGTTTGCTTTGG------------------------------------ | |
| droPer2 | scaffold_2:6793092-6793314 - | dpe_1091 | TTT--TTCTGGGTATACTCATCATTGGATACAAG------AGCGTCATTCTAAGCAGTAAGTGTGTCAAGTTGATGGAGAAGCAGGATG------------------------------------------------------------------------GGGAGGGGGA-------------------------------GAAGCGTTCCCAAGGCT---GCAATTGTCG--------------GTG---ACGTCGTTGCCTAA-------GCTTATTTGTT---------------TGTGTTT-GCC-------------------------------------------------------------TTATAAATAGCCAA----G-AAATGTGTGTGTTTGTGTGATCTCATAGCCA-----TTTTCATTCCGTTTGCTTTGG------------------------------------ |
| droBip1 | scf7180000396427:1195593-1195648 - | CCTTCTGTTTGGATTTCTAATTATTGGATACAAA------AGCATCATCTTAGCCAGTAAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302682:1148963-1149206 - | CGTTCTCTTTGGCCTACTGATTATCGGCTACAAA------ATCGTCATCCTAGCCAGTAAGTGTGCCAAGTTGAGGGCCACATATCATATTTTTTGCTTGTTTAT--------------------------ATACATTGTCCGTGCCCCCTTCCCCATTTGAGGGGGAGGA--------------------------------CACTACACTCAAGGCT---GCAATTGTCG--------------GCG---GTGACG-----CGTACTCATCGGTAGTTTGTT---------------TATATTTTG------------------------------------------------------------CGTTTATAAATAGTTTA----AGA-----------GCGTGTGATCTCATAGCGG-----T-------------------------------------------------------- | |
| droFic1 | scf7180000453809:312843-312898 + | CCTTCTCTTTGGATTTCTAATTATTGGCTACAAA------AGCGTCATATTAACCAGTAAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491214:3445260-3445315 + | TCTTCTCTTTGGCATTCTCATCATTGGCTACAAA------AGCGTCATTTTAACCAGTAAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000779932:350640-350688 + | -------TTTGGCATTCTCATTATTGGGTACAAA------AGCGTCATTTTAACCAGTAAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415820:329107-329365 + | CCTTCTTTTTGGAGCTCTAATCATTGGATACAAA------AGCGTCATTTTAACCAGTAAGTGTGTCAAGTTGAGGGCTATAT-------TGTGGGTTTTTTTATCGAATTTCCAATTATTTGTACAATATATA-----------CCCCCC--TTAATT-GCGGGGGTGGA-------------------------------------AATTCAAGGTT---GCAATTGTCGTC-----------GGTG---ACGTCGT-------CTTCACTCG--TTTTGTT---------------TATATTT-T----------------------------------------------------------CTCGTTTATAAATAGTTGA----AGT-----------TCGTGTGATCTCATAGCGG-----TGTTTATTCCCCCTGC----------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/16/2015 at 09:18 PM