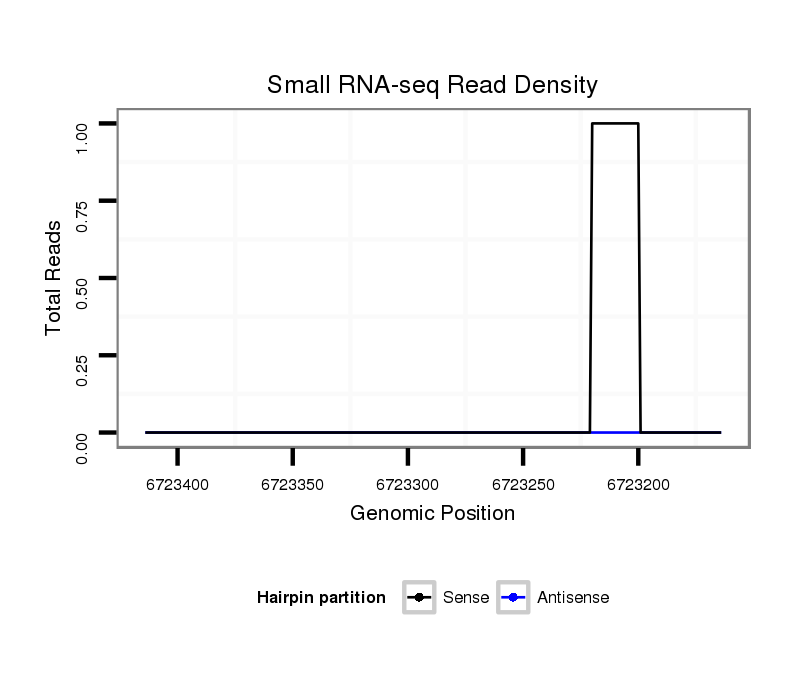
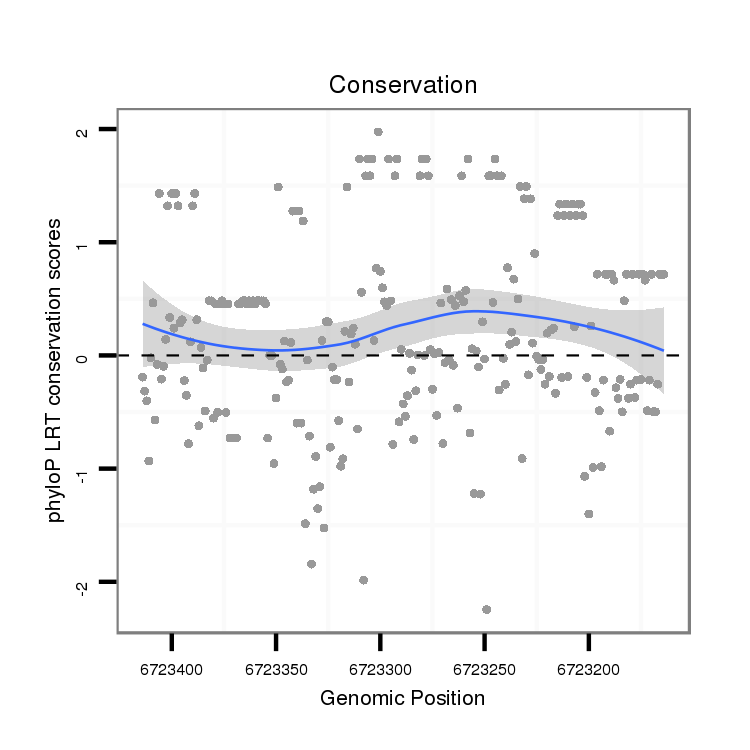
ID:dvi_5535 |
Coordinate:scaffold_12875:6723214-6723364 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_6189:1]; CDS [Dvir\GJ20776-cds]; intron [Dvir\GJ20776-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCAAAGATTGGCCAATTGCAAGTCCAACAGCAATACACTGACAAACGCATGTGAGTTATGAGTGCTAAAAGTATGTGCACACACGATTTTCTTAACCCTGCCATTTGCAGTTTATCAAAGAGTCAACATAAAACTCTGCTCGGGCATGTCAATCAATCAAAGCTTGCCCTGCGTCAGTGGCACCAACAACGCCAAGTAACAACAGCAGCAACAGCATGAAGTTTCTGTATTATTTACATATGATTATTATT **************************************************.((.(((.((.(((((((((((.((((....))))))))).......((((.....))))........(((((.........)))))((..(((((.((............)).))))).)).....))))))))..)))..)).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060685 9xArg_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................................................................................................................................AGTAACAACAGCAGCAACAGC.................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ..............ATTGCAATTCCAACAGGAAT......................................................................................................................................................................................................................... | 20 | 2 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................TATCCATCAAAGCTTACCC.................................................................................. | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 |
| GCAAAGATTGGCCAAGTT......................................................................................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
|
CGTTTCTAACCGGTTAACGTTCAGGTTGTCGTTATGTGACTGTTTGCGTACACTCAATACTCACGATTTTCATACACGTGTGTGCTAAAAGAATTGGGACGGTAAACGTCAAATAGTTTCTCAGTTGTATTTTGAGACGAGCCCGTACAGTTAGTTAGTTTCGAACGGGACGCAGTCACCGTGGTTGTTGCGGTTCATTGTTGTCGTCGTTGTCGTACTTCAAAGACATAATAAATGTATACTAATAATAA
**************************************************.((.(((.((.(((((((((((.((((....))))))))).......((((.....))))........(((((.........)))))((..(((((.((............)).))))).)).....))))))))..)))..)).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
V047 embryo |
SRR060655 9x160_testes_total |
M061 embryo |
M047 female body |
SRR060682 9x140_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................................................................TCGGTGTCGTACTTCAAAGGC........................ | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................CTAAACCTCAATTAGTTTCTCA................................................................................................................................ | 22 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................AAGTGGCACGGTAAGCGTCA............................................................................................................................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................CAAATAGATTCCCAGTTGCA.......................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................CGTTCTGTGACTGTTAGC............................................................................................................................................................................................................ | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................................................................ACCGTAAGCCTCAAATAGTTT.................................................................................................................................... | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................................................................................................CCTTGTCGTAGTTCAAGGAC........................ | 20 | 3 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................TTGGGACGCTAAACCTCAG........................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................................................................................GTGGTTACTGCGGTTCATC.................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:6723164-6723414 - | dvi_5535 | GCAAAGATTGGCCAATTGCAAG------TCCAACAGCA---ATACACTGACAAACGCATGTGAGTTATGAGTGCTAAAAGTATGTGCACA--------------------------------------------CACG-ATTTTCTTAACCCTGCCATTTGCAGTTTATCAAAGAGTCAACATAAAACTCTGCTCGGGCATGTCAATCAATCAAAGCTTGCCCTGCGTCAG---TGGCACCAACAACGCCAAGTAAC------------AACAGCAGCAACAGCATGAA-------GTTTCTGTATTA-------------------TTTACATATGATTATTATT |
| droMoj3 | scaffold_6496:13507664-13507950 + | ACAAAGGTTGGCCGATTGCAGGGCAGCAGCCAACAACAATAATTCGCTGGCCCCAGCATGTGAGTTATGC--CCTGTATATATATAC--------T-----------AGTATAACACTCTCACAATCGTTAACC-------CTTGTGCCCGCTT---TTGCCAGTTTATCAAAGAGTGAACAGAAAACTGTGCTCGAGCAACCGAATCAATCAAAGTTTGCCCTGTGCCAA---CACC-------------AACACC------------AACAACAACAACTGGAATACTGCAATAATTTATGCATGAAATGTGAGCTGGGGGAAATTTCATATATGCTTCGCATT | |
| droGri2 | scaffold_15112:1600433-1600686 - | GATAAGGTTAGCCAATTGCAGAAGTAGCACCAGAGGAA-----------------------------------GTAAGT----------CGAAAAT-----------AGAATAACCCCAAAAGGACTCTCACTT-------ATTTTGCCAACTGTCAATTTCAGTATATGAAAGGGTAACCATGCGACTCTGTGCAGGTATACAAGTCAATGACAAGCAACCATGCGGCTCCAACGGCAGCAGCAAC----AGCAAC------------AACAGCAACAACGAGGGGACACCAATTTATTATGCCCT-------------TTGAAGATGTATTTGTGCATATTATT | |
| droWil2 | scf2_1100000004921:2667946-2667998 - | GCTCTGC--------------------------------------------------------------------------------------------------------------------------------------------------------------CCATC-----------------------------------------------------------AG---TGACAACAGCAAGGACCAACAAC------------AACAACAACAACTGTA------------------------------------------------------------- | |
| dp5 | 3:2888563-2888773 - | GCGCAGGATATCCAACTGCAAC------AGCAAG-GGC-----------------------------------CTGAGCATATGTGCGTGGAATCTGCGGATTTTCG---------------------------TGAC-TTTAATCTATAAATTTCCCTTTCAGTCTACCGCAAAGTGAGTGCGAATCTCTGCAAAAATATGCCCTACGATAAGAAGCTCCCCTGTGGTAG---TGGCAGCAACG-CACTAGGCGGCAACTCTGGTGCTACCAGCAACAACAACA------------------------------------------------------------- | |
| droPer2 | scaffold_2:3079897-3080104 - | GCGCAGGATGTCCAACTGCAAC------AGCAAG-GGC-----------------------------------CTGAGCATATGTGCGTGGAATCTGCGGAGTTTCG---------------------------TGAC-TTTAATCTATAAATTTCCATTTCAGTCTACCGCAAAGTGAGTGCGAATCTCTGCAAAAATATGCCCTACGATAAGAAGCTTCCCTGTGGTAG---TGGCAGCAACG-CACTAAGCGGCAACTCTGGTGCTAGCAGCAACAAC---A------------------------------------------------------------- | |
| droTak1 | scf7180000412303:6091-6168 + | AT------------------------------------------------------------------------------------------------------------------------------------------------------------TTTCAGTCTATCGCAAGGTGGAGCTCAATCTCTGCCGTGGCCTGCCCCTCGATACAGTTCGTCCCTGCGGAAG---TGG------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409462:1992952-1993028 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------TTACAGTCTATCGCAAAGTGGAACTGAATCTATGCCGCGGCATGCCCATCGCTGTGGTTCGACCCTGTGGCAG---TGGC------------------------------------------------------------------------------------------------------------ | |
| dm3 | chr2R:8128998-8129170 + | GCGTCAACTGAGCAACTGTGGC------TCCAAAAAAT-----------------------------------GTGAGTGTATCTCCTCACAAAAAGGGGGTCAATA---------------------------TATAAATATTATTAAACATTTCCATTGCAGTCTATCGCAAGGTTGACGTGGCCCTCTGTCGTGGATTGCCCATCGATGTCGTGCGTCCCTGCGGAAG---TGGTAACAAC------------------------------------------------------------------------------------------------------ | |
| droSim2 | 2r:8835477-8835641 + | GCGGAAACTGAGCAACTGTGGC------TCCAAAAAAT-----------------------------------GTGAGTGAATCTCCCCAGAGAAAGAGGGGGGTCA---------------------------ATAT-CCCTTATTAACCATTTTCATTGCAGTCTATCGCAAAGTTGACGTGTCCCTCTGTCGTGGTTTGCCCATCGATGTCGTGCGTCCCTGCGGAAG---TGG------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4845:17664593-17664716 - | AA----------------------------------AT-----------------------------------GTGTGTG----------AAGATAC---AATATCT---------------------------CTAT-CTCTTACTAACCATTTCCATTACAGTCTATCGCAAGGTTAGCCTGGTCCTCTGTCGTGGCTTGCCCATCGATGTCCTGCGACCCTGCGGAAG---TGG------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 05:06 AM